| |
| Med Sci (Paris). 37(4): 379–385. doi: 10.1051/medsci/2021034.La cryo-microscopie électronique révèle une nouvelle vision de la cellule et de ses composants Daniel Lévy,1* Aurélie Di Cicco,1 Aurélie Bertin,1 and Manuela Dezi1 1Institut Curie, Université PSL, Sorbonne Université, CNRS UMR 168, Laboratoire Physico- Chimie Curie
,
11 rue Pierre et Marie Curie
,
75005Paris
,
France |
Comment des journaux scientifiques anglo-saxons se sont-ils convertis à un vocabulaire révolutionnaire, traditionnellement réservé à des expressions en langue française ? En décrivant la cryo-microscopie électronique (cryo-EM), comme dans les éditoriaux de
Science
en 2014 «
The resolution revolution
», et de
Nature
en 2015 «
The revolution will not be crystallized
», et 2020 «
Revolutionary cryo-EM is taking over structural biology
». La cryo-EM s’est en réalité développée depuis les années 1970, la dernière décennie ayant connu une accélération considérable de l’instrumentation et des techniques d’analyse d’images. Aujourd’hui, la cryo-EM est la méthode la plus complète pour déterminer la structure tridimensionnelle (3D) d’objets biologiques à des résolutions permettant de construire des modèles atomiques. Tous les composants cellulaires sont concernés, qu’ils soient purifiés (protéines cytosoliques et membranaires, filaments du cytosquelette, complexes ribonucléiques, etc) ou associés avec des substrats ou des inhibiteurs, ainsi également que les composants présents au sein des cellules eucaryotes, des bactéries ou des virus. La cryo-EM a ainsi décrit l’architecture 3D de protéines jusqu’alors inconnues, souvent humaines, dont l’analyse par cristallographie aux rayons X ou par résonance magnétique nucléaire (RMN) était difficile
(
Figure 1
)
. Les modèles 3D ainsi obtenus ont permis de proposer de nouveaux mécanismes fonctionnels. Ils servent aussi à la conception de nouveaux agents thérapeutiques. La cryo-EM est également une approche multi-échelles, qui permet de caractériser des échantillons biologiques de quelques nanomètres à quelques microns, et de contextualiser les protéines au sein d’assemblages fonctionnels multiprotéiques, ou au sein d’environnements aussi complexes que l’intérieur des cellules. La cryo-EM dépasse largement le cadre de la biologie structurale. Elle stimule en effet des recherches en mathématiques appliquées, en physique expérimentale et théorique, dans de nombreux domaines de la biologie, jusqu’en biomédecine, et permet des développements industriels en pharmacologie, en ingénierie instrumentale et computationnelle. C’est pour toutes ces avancées et le potentiel de la cryo-EM que Jacques Dubochet, Joachim Frank et Richard Henderson ont reçu le prix Nobel de chimie en 2017 [
38
] (
→
).
(→) Voir le Repères de E. Neumann
et al.
,
m/s
n° 12, décembre 2017, page 1111
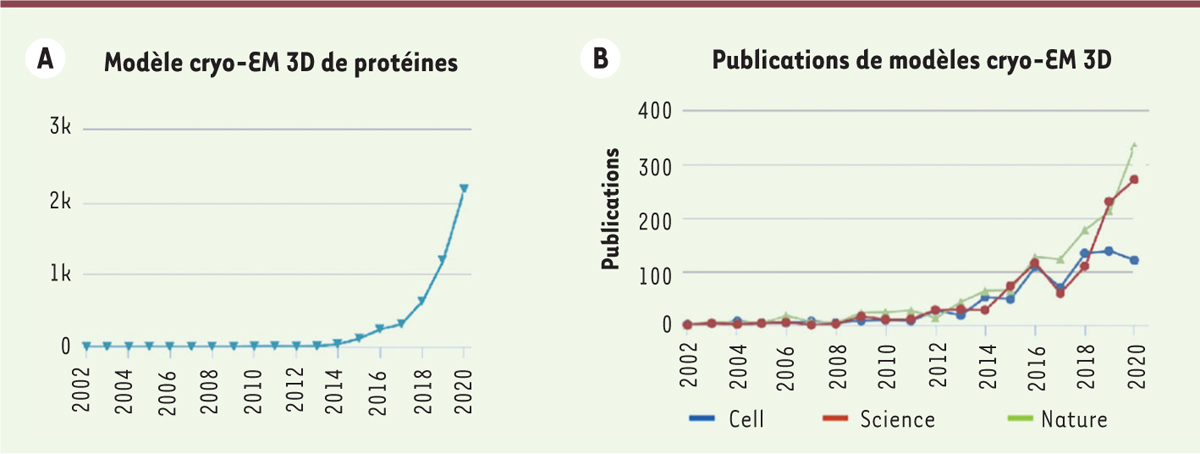
 | Figure 1.
L’impact de la cryo-microscopie électronique en biologie en chiffres (Janvier 2021). A.
Nombre de modèles 3D de protéines depuis 2002.
B.
Nombre d’articles publiés dans
Cell
,
Nature
, et
Science
, dont les principaux résultats sont issus des modèles 3D obtenus par cryo-EM (
https://www.ebi.ac.uk/pdbe/emdb/
).
|
|
La cryo-microcopie électronique
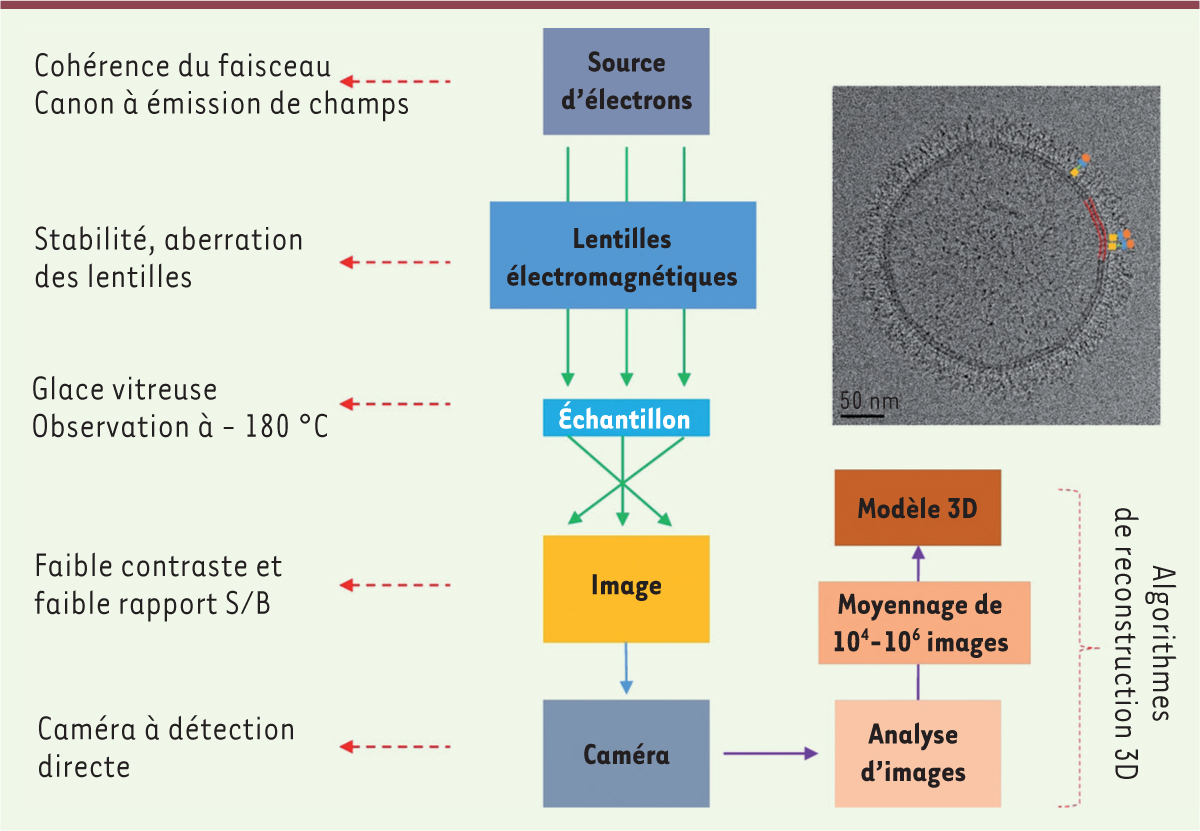
La cryo-EM est fondée sur l’utilisation d’un microscope électronique à transmission, pour imager des échantillons biologiques congelés. Le microscope électronique, dont le principe a été établi par le physicien allemand Ernst Ruska, en 1931, est constitué d’une source d’émission d’électrons accélérés dans une colonne sous vide et sous haut voltage, d’une série de lentilles électromagnétiques, dont on peut varier le courant pour définir le chemin optique du faisceau, de l’échantillon à imager, de lentilles de projection pour agrandir l’image, et enfin, d’un système d’acquisition d’images
(
Figure 2
)
.
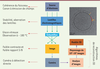 | Figure 2.
Représentation schématique d’un microscope électronique, des avancées récentes de la cryo-EM et de l’analyse d’images pour la détermination de modèles 3D d’échantillons biologiques.
Image de cryo-EM de molécules de myosine 1B attachées à une vésicule lipidique ; les deux feuillets de la bicouche (traits rouges) et les protéines individuelles sont résolus.
|
La résolution théorique d’un microscope électronique est liée à l’accélération des électrons. Elle peut-être évaluée à 0,01 ångström (Å) pour une accélération de 300 kVolts. La plus haute résolution actuelle obtenue est de 0,63 Å, pour une couche monomoléculaire de nitrure de gallium (GaN) qui montre les atomes individuels et les défauts atomiques dans la couche.
Le faisceau d’électrons a cependant posé deux difficultés pour imager des échantillons biologiques : 1) la nécessité de travailler sous vide poussé, afin d’éviter que le faisceau d’électrons ne perde en cohérence en interagissant avec des molécules d’air présentes dans la colonne. Mais ce vide provoque l’évaporation de l’eau des échantillons biologiques ; et 2) la haute énergie associée au faisceau, qui brûle les échantillons, ce qui nécessite de travailler avec de très faibles doses d’électrons et conduit à des images à très faible rapport signal sur bruit (S/B), et donc de mauvaise qualité [
1
].
Pour éviter l’évaporation de l’eau, la solution a été trouvée en congelant les échantillons biologiques dans une couche mince de glace vitreuse, transparente au électrons, et en les imageant sous forme congelée. L’échantillon est donc plongé dans un bain d’éthane liquide (ou un mélange éthane/propane) qui transforme l’eau liquide en glace vitreuse en quelques millisecondes, évitant ainsi la formation de cristaux de glace qui peuvent abîmer les structures. Les échantillons sont ainsi figés dans l’état « natif » dans lequel ils étaient avant congélation [
2
]. C’est cet ensemble de préparations à l’observation des échantillons, qui doivent être maintenus à - 180 °C, qui constitue la cryo-EM.
Le problème du faible rapport signal sur bruit des images a également été résolu par des avancées en analyse d’images et des progrès obtenus dans les instruments utilisés en cryo-EM. À la différence de la microscopie électronique ultrastructurale à température ambiante, en cryo-EM, les échantillons ne sont pas colorés. Pour augmenter le rapport signal sur bruit, l’idée a été de moyenner un nombre important d’images pour reconstruire des modèles 3D des protéines, soit par l’approche dite d’analyse de particule isolée (
single particle analysis
, SPA), soit par moyennage de sous-volumes (
sub-tomogram averaging
, STA) [
3
,
4
]. Les principaux développements en termes d’instrumentation ont, quant à eux, concerné la qualité du faisceau d’électrons, avec une source plus cohérente, de type FEG (
field emission gun
), des lentilles électromagnétiques plus stables, ou des diaphragmes automatisés. En 2012, l’arrivée des caméras à détection directe d’électrons, qui ont remplaçé les caméras CCD (
charge coupled device
ou, en français, dispositif à transfert de charge), a permis d’obtenir des images avec un rapport signal sur bruit très supérieur. Grâce à ces caméras, on « voit » beaucoup mieux, ce qui permet des analyses plus pertinentes, plus rapides et à meilleures résolutions.
|
L’analyse de particules isolées
Le principe de l’analyse des particules isolées (SPA) consiste à prendre des images de protéines purifiées (ou
particules
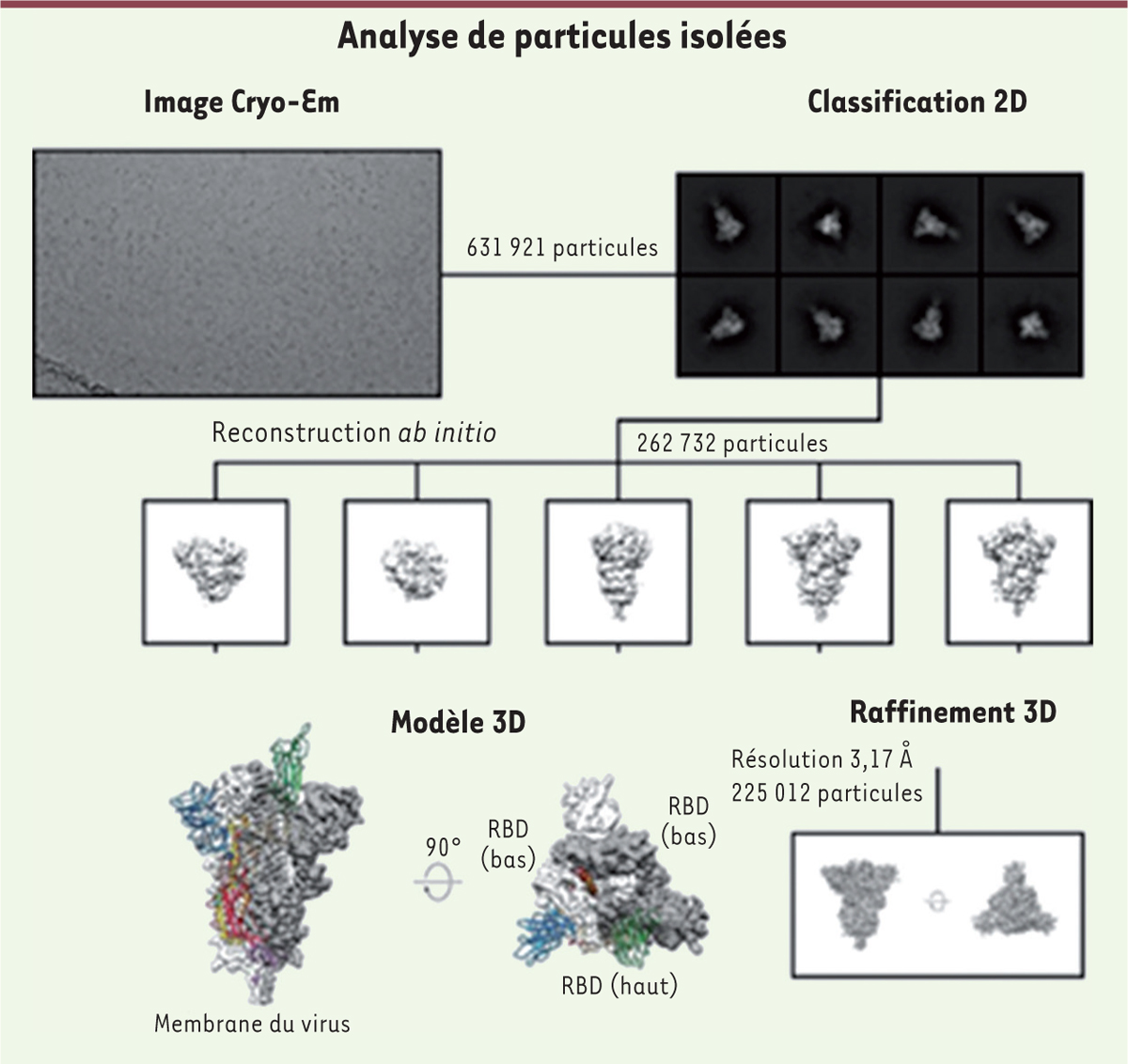
), immobilisées dans une couche de glace vitreuse de 30 à 100 nm d’épaisseur, dans différentes orientations. Des modèles 3D sont ensuite reconstruits à partir de ces images, selon une procédure de plus en plus standardisée
(
Figure 3
)
[
5
]. L’analyse commence alors par une correction du mouvement des particules, induit par le faisceau électronique, dans la glace. Les particules, dont le nombre varie en général entre 10
4
et 10
6
, sont ensuite identifiées à l’aide d’algorithmes à partir d’images de protéines homologues, ou en utilisant des filtres gaussiens
1
, puis alignées en rotation et translation 2D, triées dans différentes classes 2D, en fonction de leurs orientations dans la glace, et, enfin, moyennées dans le but d’augmenter le rapport signal sur bruit. Le sous-ensemble de particules est ensuite soumis à des cycles itératifs d’alignement et de classification 3D, en utilisant une approche de correspondance de projection, pour déterminer les relations angulaires entre classes 2D. Les relations angulaires sont ensuite affinées séparément afin d’améliorer la résolution de la reconstruction 3D. Différents programmes de reconstructions 3D et des tutoriaux sont accessibles en ligne, les principaux étant Relion, Cryo-SPARC, EMAN, WRAP.
 | Figure 3.
Principe de la détermination de structures par cryo-EM et analyse de particules isolées. Exemple de la structure de la protéine Spike (S) du SARS-CoV-2 en conformation pré-fusion
(adapté de [
20
]). Des images de protéines (ou particules) dans la glace vitreuse sont alignées, classées et moyennées selon leur orientation dans la glace pour augmenter le rapport signal/bruit, puis les moyennes servent à construire des modèles 3D qui sont enfin affinés pour construire un modèle atomique.
|
La SPA présente plusieurs atouts. Il n’est en effet pas nécessaire d’obtenir de cristaux pour construire un modèle 3D. Les protéines sont imagées quelques minutes après leur purification, ce qui permet l’analyse de protéines instables. Elles sont « complètes », c’est-à-dire qu’elles contiennent les domaines flexibles, les éventuels cofacteurs associés, et, pour les protéines eucaryotes, leurs modifications post-traductionnelles. L’image contient en outre les informations de phase et d’intensité, ce qui permet de reconstruire des modèles 3D quelle que soit la résolution. Les processus de classification 2D et 3D permettent enfin de déterminer les différentes conformations des protéines présentes en solution, donnant ainsi une vision pseudo-dynamique de ces molécules biologiques. Ces modèles 3D, de même que les caractéristiques de leur obtention, sont référencés et identifiés par un numéro de type EMD-XXXX, et accessibles dans une base de données
2
.
La qualité de la reconstruction dépend de la taille et de la flexibilité des protéines : plus l’objet est grand et rigide, meilleurs sont les alignements et la résolution finale. Ainsi, la streptavidine (de masse moléculaire 58 kDa), l’une des plus petites protéines analysées, a été reconstruite à une résolution de 2,6 Å (EMD-20907). La plus haute résolution actuelle est de 1,15 Å, pour un modèle d’apoferritine, où sont visualisées les molécules d’eau, les ions liés et les liaisons hydrogène (EMD-11668). |
Beaucoup d’échantillons biologiques, comme les complexes protéiques associés aux membranes, les machineries multicomposantes, ou les virus, ont des organisations natives en 3D qui sont hétérogènes et variables. Ces complexes sont difficiles à purifier et ils n’existent qu’au sein d’environnements natifs ou pseudo-natifs. Ces échantillons sont cependant analysables par cryo-tomographie électronique (Cryo-ET), l’autre branche de la cryo-EM avec la SPA, sur laquelle les cryo-microscopistes portent beaucoup d’espoirs [
6
].
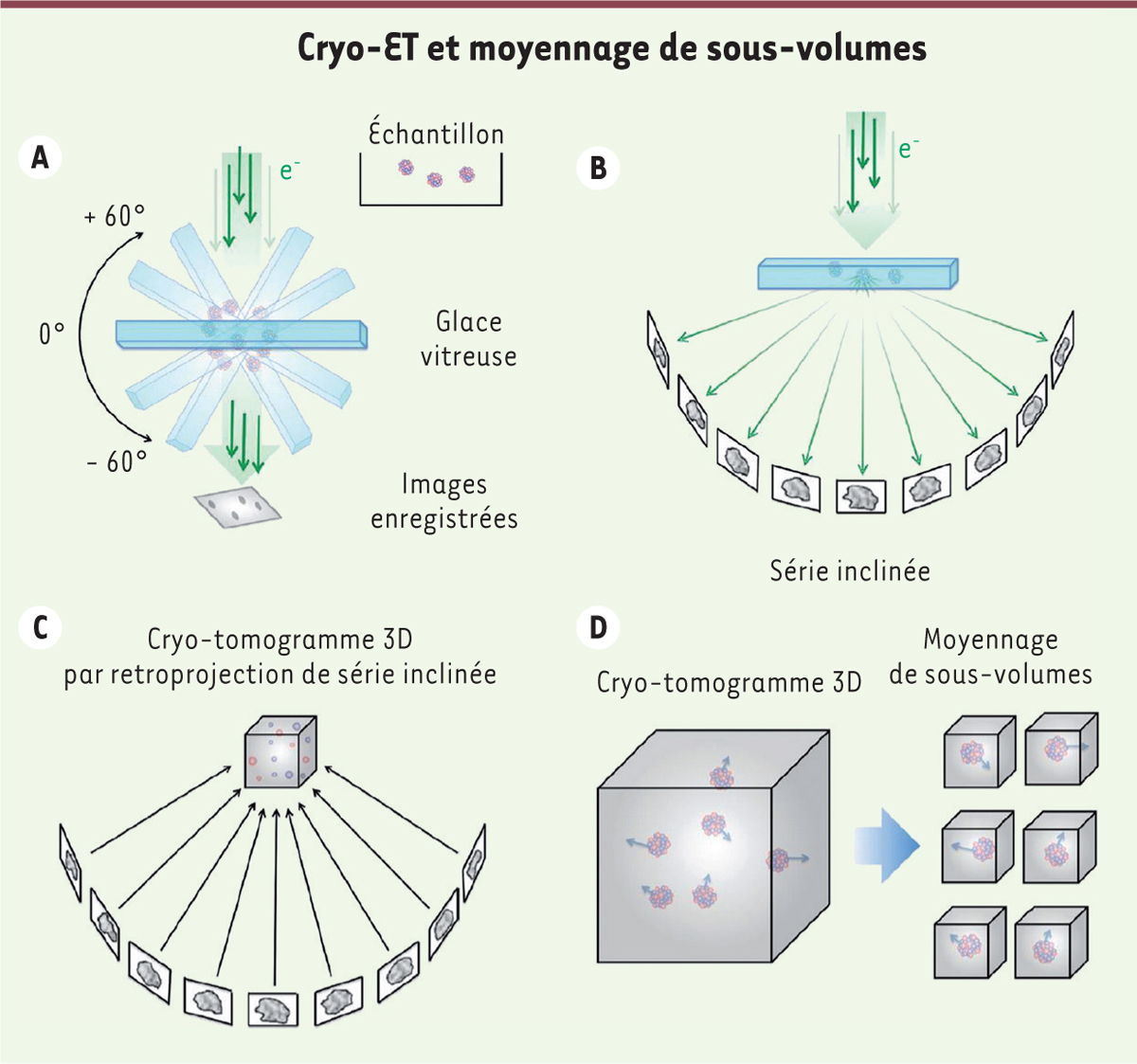
La cryo-ET consiste à prendre 40 à 60 images du même échantillon, incliné à différents angles par rapport au faisceau d’électrons, pour ensuite, après alignement de la série d’images inclinées, obtenir un cryo-tomogramme de l’échantillon. Comparé à la SPA, la résolution est de quelques nm, le rapport signal sur bruit étant plus faible. En effet, la dose d’électrons par image est réduite, afin d’éviter que la dose cumulée pour la série d’images ne détériore l’échantillon ; de plus, le faisceau d’électrons traverse des échantillons plus épais, entre 100 et 300 nm. On peut ensuite établir des modèles 3D des protéines présentes dans les reconstructions 3D, selon le même principe que celui de la SPA, mais les particules sont dans ce cas des sous-volumes plutôt que des projections bidimensionnelles. Le calcul de la moyenne des sous-tomogrammes (
sub-tomogram averaging
, STA) s’effectue par itération, l’alignement et le moyennage d’un grand nombre de sous-volumes augmentant le rapport signal sur bruit et la résolution
(
Figure 4
)
[
4
]. Les principales applications, disponibles en ligne, qui sont utilisées pour ces déterminations de structure, sont DYNAMO, PEET, AV3, TOM EMclarity.
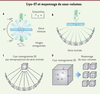 | Figure 4.
Principe de la cryo-tomographie électronique et du moyennage de sous-volumes
(adapté de [
36
]). Un échantillon présent dans la glace vitreuse est imagé à différents angles par rapport au faisceau d’électrons. à partir de cette série d’images inclinées, un cryo-tomogramme ou volume 3D de l’échantillon est reconstruit. Plusieurs sous-volumes contenant des sous-domaines identiques de l’échantillon sont extraits, alignés et moyennés pour augmenter le rapport signal sur bruit et obtenir un modèle 3D. Les différents modèles 3D sont ensuite recalés dans le cryo-tomogramme pour avoir une vision complète de l’échantillon incluant les hétérogénéités.
|
La preuve de principe que l’on peut obtenir la même résolution de 2,3 Å par STA et par SPA (EMD-11603) avec l’apoferritine, une protéine rigide, a été faite dans une très fine couche de glace. Mais l’intérêt de la cryo-ET est d’ouvrir un champs d’analyse à des échantillons qui ne sont pas accessibles à la SPA.
La cryo-ET a en effet permis, par exemple, de déterminer les différentes architectures et conformations locales de protéines présentes à la surface de capsides de virus assemblées
in vitro
(comme le
severe acute respiratory syndrome coronavirus 2
[SARS-CoV-2]), le virus Zika, le virus Ebola, le virus de l’immunodéficience humaine (VIH), etc.). Elle a permis également de déterminer les différentes architectures de machineries protéiques impliquées dans le remodelage membranaire, comme les complexes COP (c
oat protein comple
x) I et II [
7
] ou ESCRT (
endosomal sorting complexes required for transport
) III [
8
], de protéines du centriole impliquées dans la division cellulaire [
9
], ou de complexes protéiques participant à la communication entre organelles [
37
].
Le Graal de la cryo-ET est d’analyser les protéines directement au sein des cellules. La principale limitation de cette analyse est l’épaisseur de la cellule, trop importante pour des images par microscopie à transmission. La cryo-ET de cellule s’est donc tout d’abord adressée aux bactéries, aux virus et aux bords des cellules eucaryotes. Des développements instrumentaux ont ensuite permis de réaliser des « cryo-lamelles » de cellules dont l’épaisseur était adaptée à la cryo-ET. Pour cela, les cellules sont déposées sur une grille, congelées dans la glace vitreuse, puis « évidées » jusqu’à des épaisseurs de 200 à 300 nm avec un faisceau électronique dans un microscope à balayage (
cryo-FIB milling
) [
6
]. Les cryo-lamelles sont ensuite analysées par cryo-ET. Une technique encore plus poussée a été développée pour les organismes entiers, comme le vers
Caenorhabditis elegans
[
10
]. Grâce à ces méthodes, il est ainsi possible d’obtenir des reconstructions 3D d’une portion de l’intérieur d’une cellule (par exemple une portion de 200 nm × 2-5 mm × 2-5 mm) présentant toute la variété de ses composants (protéines, membranes, microtubules, noyau, etc.) [
11
]. On peut ainsi mesurer et décompter ces composants individuellement et relier ces données à l’échelle moléculaire à des propriétés fonctionnelles, de régulation ou de mécanique intracellulaire [
12
] [
13
]. Des modèles 3D, déterminés par STA, peuvent être obtenus à des résolutions qui dépendent de la taille de l’objet et du nombre de copies souvent très limité dans la cryo-lamelle, la meilleure résolution obtenue étant de 3,4 Å pour des ribosomes (EMD-11999). La cryo-ET
in situ
, qui nécessite des instruments et des expertises particulières pour réaliser les cryo-lamelles, est pratiquée aujourd’hui par un petit nombre d’équipes mais qui va croissant, en raison des promesses d’obtenir de nouvelles visions en 3D de la cellule [
39
] (
→
).
(→) Voir la Nouvelle de J. Lafaurie-Janvore et M. Piel,
m/s
n° 12, décembre 2013, page 1089
|
Les limitations de la cryo-EM
La cryo-EM présente cependant plusieurs limitations qui freinent encore son développement : les cryo-microscopes et les contrats d’entretiens annuels sont coûteux (les microscopes de très haute résolution comme le Titan Krios coûtent entre 5 et 7 M€) ; la préparation des grilles reste semi-empirique. Lors de la congélation, les protéines s’accumulent et se dénaturent partiellement à l’interface avec l’air en fonction de leurs propriétés physico-chimiques. Différentes stratégies existent, mais sans solution unique applicable à tout type de protéine. L’étape d’optimisation des grilles peut ainsi être longue, allant jusqu’à plusieurs mois, et nécessite des allers-retours entre préparation des grilles, observations en cryo-microscopie sur un cryo-microscope intermédiaire, analyses d’image et biochimie [
14
] ; l’analyse d’image et la construction de modèles atomiques restent également un processus long et qui demande des expertises spécifiques [
15
].
|
L’impact de la cryo-EM en biologie/santé
L’impact de la cryo-EM en biologie/santé est d’ores et déjà immense, tant en recherche fondamentale qu’en recherche appliquée. Plusieurs grands groupes pharmaceutiques, comme
Novartis
,
Pfizer
ou
AstraZeneca
, disposent désormais de centres dédiés à la cryo-EM. Depuis 2015, environs 1 500 structures de protéines humaines ont été déterminées par SPA
2
. La liste comprend un grand nombre de protéines transmembranaires. Ces protéines, qui étaient réfractaires jusqu’à présent à l’analyse structurale, sont les cibles de 50 % des médicaments disponibles. Les modèles 3D déterminés par cryo-EM servent ainsi de base pour la conception de nouveaux médicaments.
Parmi ces protéines, on peut citer les membres de la famille des transporteurs membranaires de type ABC, dont CFTR (
cystic fibrosis transmembrane conductance regulator
), une protéine responsable du flux d’ions chlorure et mutée chez les patients atteints de mucoviscidose, BRCP (
breast cancer resistance protei
n) et PGP (
P-glycoprotein
), deux protéines qui participent à la résistance aux agents anticancéreux, ainsi que ceux de la superfamille des récepteurs couplés aux protéines G, impliqués dans des désordres neurologiques, endocriniens, cardio-vasculaires et immunitaires [
16
,
17
]. L’approche SPA a également permis de déterminer les structures de protéines directement extraites de cerveaux de malades et impliquées dans la formation des fibres amyloïdes, responsables de maladies neurodégénératives [
18
]. Enfin, les architectures moléculaires d’agrégats de protéines responsables de la maladie de Huntington, de troubles fronto-temporaux, de sclérose latérale amyotrophique (SLA), ou de filaments d’uromoduline, une protéine qui bloque certaines infections urinaires, ont été visualisées directement à l’intérieur des cellules [
19
].
Depuis février 2020, la cryo-EM a montré toute sa pertinence dans l’étude du SARS-Cov-2, avec la détermination, par SPA, de la structure de la protéine de surface Spike (S), qui permet au virus de se fixer à son récepteur ACE2 (enzyme de conversion de l’angiotensine 2) [
20
], de la protéine ACE2 elle-même [
21
], de l’ARN polymérase virale, seule ou liée à l’antiviral remdésivir [
22
], ou de la protéine S en complexe avec des anticorps monoclonaux potentiellement à usage thérapeutique [
23
]. La reconstruction 3D complète par cryo-ET et STA de la protéine S du virus, extraite de cellules pulmonaires infectées, a révélé les différentes conformation de cette protéine, pré- et post-fusion [
24
]. Elle a ensuite permis de définir la structure du virus isolé de patients [
25
], ainsi que les étapes de l’assemblage du virus
in situ
[
26
].
|
Les autres approches de cryo-EM
La cryo-EM se développe vers des techniques autres que la SPA et la cryo-ET. Parmi celles-là, citons les approches de microscopie corrélative photonique-électronique (Cryo-CLEM) en microscopie photonique classique ou en super-résolution. Cette technique permet d’identifier et de visualiser, en combinant la détection par fluorescence, une protéine ou un évènement d’intérêt, comme l’entrée d’un virus au sein d’une cellule, avant ou après congélation, et ensuite de les imager par cryo-EM dans leur contexte cellulaire [
27
]. Une autre approche est la micro-diffraction électronique (micro-ED), qui utilise la capacité des cryo-microscopes d’acquérir des données en mode diffraction pour la détermination de structures à partir de cristaux 3D de très petites tailles (entre 50 et 500 nm), de petites protéines ou de peptides, jusqu’à 0,6 Å de résolution. Enfin, la microscopie à balayage en mode à froid (cryo-STEM) permet l’étude d’échantillons de plusieurs micromètres d’épaisseur.
|
Il est difficile de prédire le futur de la cryo-EM, d’autant que plusieurs communautés scientifiques, motivées par des ambitions différentes, s’investissent dans son développement. Les mathématiques appliquées développent en effet de nouveaux algorithmes de classification, d’apprentissage automatique ou de visualisation 3D ; la physique théorique et expérimentale améliore les microscopes électroniques et les caméras ; la chimie s’intéresse à la synthèse de sondes fluorescentes pour les approches de cryo-CLEM, ou à l’amélioration des surfaces des grilles d’observation ; la biochimie conçoit des systèmes reconstitués
in vitro
de plus en plus complexes ; la biologie structurale et la bio-informatique imaginent des approches de modélisation et de dynamiques moléculaire et cellulaire à partir des reconstructions 3D de cryo-EM
(
Figure 5
)
[
28
,
29
].
 | Figure 5.
Quelques développements attendus de la cryo-EM concernant chacune des étapes pour la détermination de modèles 3D de macromolécules biologiques purifiées et
in cellulo
.
|
Citons quelques preuves de concept déjà obtenues. Pour la préparation des échantillons, plusieurs approches sont développées pour automatiser la congélation des échantillons, réduire le temps de congélation à quelques millisecondes, et réduire la quantité de protéines nécessaire en utilisant des volumes de moins de 50 nanolitres, grâce à des robots qui projettent des gouttes d’échantillon sur des grilles [
30
]. Les plans ayant permis de concevoir ces robots sont disponibles, ce qui assure leur diffusion rapide dans les laboratoires. Différents types de grilles ont été imaginés pour améliorer le dépôt des protéines ainsi que des grilles micro-usinées pour imposer des contraintes mécaniques à des cellules avant congélation [
13
,
31
]. En ce qui concerne les microscopes, des améliorations ont été apportées : amélioration du contraste des images, avec des
« laser phase plate »
de meilleure stabilité que les
« volta phase plate »
actuelles [
32
] ; la possibilité d’utiliser des cryo-microscopes opérant à 200 kVolts, pour déterminer des structures de protéines jusqu’à au moins 1,75 Å de résolution (EMD-21024), pour un coût moins élevé qu’avec les cryo-microscopes Titans Krios opérant à 300 kVolts [
33
]. Henderson et ses collègues travaillent ainsi sur un cryo-microscope à 100 kVolts, pour un coût estimé à environ 1 M€ [
34
]. Des gains significatifs sont encore possibles dans l’enregistrement des images par les caméras [
15
]. Au niveau de l’analyse des images, des approches d’apprentissages automatiques et en profondeur (
deep learning
) sont développées pour identifier les zones de la grille de cryo-EM à imager, pour la sélection automatique des particules sur les images, la détermination de structures en s’appuyant sur des comparaisons avec les banques de données existantes, la segmentation automatique et la reconnaissance de formes dans les cryo-tomogrammes [
35
]. Des solutions sont également proposées pour la gestion et l’archivage des données en quantités croissantes [
35
]. Le but est de fournir des solutions à des chercheurs moins experts pour déterminer plus rapidement, et en nombre plus important, des structures de protéines, et leur permettre de se focaliser sur l’interprétation de ces structures.
Des efforts ont été réalisés par la communauté de chercheurs impliqués dans la cryo-EM, afin d’établir des standards de qualité et favoriser l’accès des autres chercheurs aux données brutes ou après analyse, avec les banques de données EMDB et EMPIAR
3,
. Ces données peuvent ainsi être utilisées pour développer de nouveaux programmes d’analyse et de modélisation moléculaire ou cellulaire [
29
].
Tous ces développements sont intégrés à des plateformes internationales, comme celles du réseau d’infrastructures européennes INEXT
4
. Les utilisateurs académiques, après évaluation de leur projet par des pairs, peuvent ainsi obtenir des jeux d’images acquises par des opérateurs experts gratuitement ou à faible coût. Une partie du prétraitement des images est déjà réalisée sur le site. Dans le futur, il est envisagé que les structures complètes soient déterminées et disponibles.
En France, plusieurs groupes de recherches ont participé au développement de la cryo-EM : l’Institut de génétique et de biologie moléculaire et cellulaire (IGBMC), à Illkirch ; l’Institut de biologie structurale (IBS) et l’
European molecular biology laboratory
(EMBL), à Grenoble ; l’Institut génétique & développement (IGDR), à Rennes ; le Centre de biologie structurale (CBS) de Montpellier ; l’Institut Curie et l’Institut Pasteur, à Paris ; l’Institut européen de chimie et biologie (IECB) à Bordeaux ; le Centre de biologie intégrative (CBI) à Toulouse.
En 2020, trois cryo-microscopes Titans Krios étaient en service en France, avec un nombre de personnels techniques associés très faible. Ce chiffre est à comparer aux 25 à 30 microscopes Titans Krios disponibles au Royaume-Uni ou en Allemagne, et aux 7 appareils du
New York Center for Structural Biology
. Le modèle économique imposé en France n’est pas optimal et ne favorise pas l’expansion de la cryo-EM
5
.
Il reste qu’un nombre croissant de chercheurs en biologie structurale, en biologie cellulaire et du développement, en bioinformatique, en physique de la cellule, ou en mathématiques appliquées, ont des problématiques et des questions auxquelles la cryo-EM pourrait répondre. ‡ |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Les auteurs remercient Cynthia Taveneau, Julien Maufront, Eugenio de la Mora, Pierre Fribourg, Johan Busselez pour leur contribution aux recherches en cryo-EM de l’équipe Molecular Microscopy of Membranes de l’UMR 168 à l’Institut Curie, ainsi que l’Institut Curie pour son soutien à la cryo-EM depuis 1995, et le CNRS.
|
Footnotes |
1.
Henderson
R
.
The potential and limitations of neutrons, electrons and x-rays for atomic resolution microscopy of unstained biological molecules.
.
Q Rev Biophys.
1995;
;
28
:
:171.
–
193
.
2.
Dubochet
J
,
Adrian
M
,
Chang
J
,
et al.
Cryo-electron microscopy of vitrified specimens.
.
Q Rev Biophys.
1988;
;
21
:
:129.
–
128
.
3.
Frank
J
.
Single-particle imaging of macromolecules by cryo-electron microscopy.
.
Annu Rev Biophys Biomol Struct.
2002;
;
31
:
:303.
–
319
.
4.
Wan
W
,
Briggs
JAG
Cryo-Electron tomography and subtomogram averaging.
.
Methods Enzymol.
2016;
;
279
:
:329.
–
367
.
5.
Cheng
Y
,
Grigorieff
N
,
Penczek
PA
,
et al.
A primer to single-particle cryo-electron microscopy.
.
Cell.
2015;
;
161
:
:439.
–
449
.
6.
Beck
M
,
Baumeister
W
Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail?
.
Trends Cell Biol.
2016;
;
26
:
:825.
–
837
.
7.
Hutchings
J
,
Stancheva
V
,
Miller
EA
,
et al.
Subtomogram averaging of COPII assemblies reveals how coat organization dictates membrane shape.
.
Nat Commun.
2018;
;
9
:
:4154.
.
8.
Bertin
A
,
Franceschi
N
de,
la Mora
E
de,
et al.
Human ESCRT-III polymers assemble on positively curved membranes and induce helical membrane tube formation.
.
Nat Commun.
2020;
;
11
:
:2663.
.
9.
Guichard
P
,
Hamel
V
,
Le
Guennec M
,
et al.
Cell-free reconstitution reveals centriole cartwheel assembly mechanisms.
.
Nat Commun.
2017;
;
8
:
:1.
–
9
.
10.
Schaffer
M
,
Pfeffer
S
,
Mahamid
J
,
et al.
A cryo-FIB lift-out technique enables molecular-resolution cryo-ET within native Caenorhabditis elegans tissue.
.
Nat Methods.
2019;
;
16
:
:757.
–
762
.
11.
Mahamid
J
,
Pfeffer
S
,
Schaffer
M
,
et al.
Visualizing the molecular sociology at the HeLa cell nuclear periphery.
.
Science.
2016;
;
351
:
:969.
–
972
.
12.
Delarue
M
,
Brittingham
GP
,
Pfeffer
S
,
et al.
mTORC1 controls phase separation and the biophysical properties of the cytoplasm by tuning crowding.
.
Cell.
2018;
;
174
:
(338–49)
:e20.
.
13.
Vignaud
T
,
Copos
C
,
Leterrier
C
,
et al.
Stress fibres are embedded in a contractile cortical network.
.
Nat Mater.
2020;
;
20
:
:410.
–
20
.
14.
Noble
AJ
,
Dandey
VP
,
Wei
H
,
et al.
Routine single particle CryoEM sample and grid characterization by tomography.
.
Elife.
2018;
;
7
:e34257.
.
15.
Wu
M
,
Lander
GC
.
Present and emerging methodologies in cryo-em single-particle analysis.
.
Biophys J.
2020;
;
119
:
:1281.
–
9
.
16.
Polovinkin
L
,
Hassaine
G
,
Perot
J
,
et al.
Conformational transitions of the serotonin 5-HT3 receptor.
.
Nature.
2018;
;
563
:
:275.
–
279
.
17.
Kumar
A
,
Planchais
C
,
Fronzes
R
,
et al.
Binding mechanisms of therapeutic antibodies to human CD20.
.
Science.
2020;
;
369
:
:793.
–
9
.
18.
Zhang
W
,
Tarutani
A
,
Newell
KL
,
et al.
Novel tau filament fold in corticobasal degeneration.
.
Nature.
2020;
;
580
:
:283.
–
7
.
19.
Weiss
GL
,
Stanisich
JJ
,
Sauer
MM
,
et al.
Architecture and function of human uromodulin filaments in urinary tract infections.
.
Science.
2020;
;
369
:
:1005.
–
10
.
20.
Wrapp
D
,
Wang
N
,
Corbett
KS
,
et al.
Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation.
.
Science.
2020;
;
367
:
:1260.
–
3
.
21.
Yan
R
,
Zhang
Y
,
Li
Y
,
et al.
Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2.
.
Science.
2020;
;
367
:
:1444.
–
8
.
22.
Yin
W
,
Mao
C
,
Luan
X
,
et al.
Structural basis for inhibition of the RNA-dependent RNA polymerase from SARS-CoV-2 by remdesivir.
.
Science.
2020;
;
368
:
:1499.
–
504
.
23.
Lv
Z
,
Deng
YQ
,
Ye
Q
,
et al.
Structural basis for neutralization of SARS-CoV-2 and SARS-CoV by a potent therapeutic antibody.
.
Science.
2020;
;
369
:
:1505.
–
9
.
24.
Ke
Z
,
Oton
J
,
Qu
K
,
et al.
Structures and distributions of SARS-CoV-2 spike proteins on intact virions.
.
Nature.
2020;
;
588
:
:498.
–
502
.
25.
Yao
H
,
Song
Y
,
Chen
Y
,
et al.
Molecular architecture of the SARS-CoV-2 virus.
.
Cell.
2020;
;
183
:
:730.
–
8.e13
.
26.
Klein
S
,
Cortese
M
,
Winter
SL
,
et al.
SARS-CoV-2 structure and replication characterized by in situ cryo-electron tomography.
.
Nat Commun.
2020;
;
11
:
:5885.
.
27.
Moser
F
,
Pražák
V
,
Mordhorst
V
,
et al.
Cryo-SOFI enabling low-dose super-resolution correlative light and electron cryo-microscopy.
.
Proc Natl Acad Sci USA.
2019;
;
116
:
:4804.
–
4809
.
28.
Cuniasse
P
,
Tavares
P
,
Orlova
E V
,
et al.
Structures of biomolecular complexes by combination of NMR and cryoEM methods.
.
Curr Opin Struct Biol.
2017;
;
43
:
:104.
–
113
.
29.
Earnest
TM
,
Watanabe
R
,
Stone
JE
,
et al.
Challenges of integrating stochastic dynamics and cryo-electron tomograms in whole-cell simulations.
.
J Phys Chem B.
2017;
;
121
:
:3871.
–
3881
.
30.
Dandey
VP
,
Budell
WC
,
Wei
H
,
et al.
Time-resolved cryo-EM using spotiton.
.
Nat Methods.
2020;
;
17
:
:897.
–
900
.
31.
Toro-Nahuelpan
M
,
Zagoriy
I
,
Senger
F
,
et al.
Tailoring cryo-electron microscopy grids by photo-micropatterning for in-cell structural studies.
.
bioRxiv.
2019
;
:676189.
.
32.
Schwartz
O
,
Axelrod
JJ
,
Campbell
SL
,
et al.
Laser phase plate for transmission electron microscopy.
.
Nat Methods.
2019;
;
16
:
:1016.
–
1020
.
33.
Herzik
MA
,
Wu
M
,
Lander
GC
Achieving better-than-3-Å resolution by single-particle cryo-EM at 200 keV.
.
Nat Methods.
2017;
;
14
:
:1075.
–
1078
.
34.
Naydenova
K
,
McMullan
G
,
Peet
MJ
,
et al.
CryoEM at 100keV: a demonstration and prospects.
.
IUCrJ.
2019;
;
6
:
:1086.
–
1098
.
35.
Baldwin
PR
,
Tan
YZ
,
Eng
ET
,
et al.
Big data in cryoEM: automated collection, processing and accessibility of EM data.
.
Curr Opin Microbiol.
2018;
;
43
:
:1.
–
8
.
36.
Galaz-Montoya
JG
,
Ludtke
SJ
The advent of structural biology in situ by single particle cryo-electron tomography.
.
Biophys Reports.
2017;
;
3
:
:17.
–
35
.
38.
Neumann
E
,
Farias Estrozi
L
,
Effantin
G
,
et al.
Prix Nobel de Chimie 2017: Jacques Dubochet, Joachim Frank et Richard Henderson.La révolution de la résolution en cryo-microscopie électronique.
.
Med Sci (Paris).
2017;
;
33
:
:1111.
–
1117
.
39.
Lafaurie-Janvore
J
,
Piel
M
Mécanique et division cellulaire : contrôle temporel de l’abscission.
.
Med Sci (Paris).
2013;
;
29
:
:1089.
–
1091
.
|