| |
| Med Sci (Paris). 37(4): 372–378. doi: 10.1051/medsci/2021033.Une moisson de nouvelles structures de mTORC1 Coup de projecteur sur les GTPases Rag Agata Nawrotek1* and Jacqueline Cherfils1** 1CNRS, LBPA, UMR 8113,École normale supérieure Paris-Saclay, Université Paris-Saclay
,
4 avenue des Sciences
,
91190Gif-sur-Yvette
,
France |
La croissance cellulaire est le processus par lequel les cellules augmentent de masse et de taille, par exemple à l’occasion de la division cellulaire. C’est un processus étroitement contrôlé qui répond de façon coordonnée à des signaux extracellulaires, comme les facteurs de croissance ou les nutriments, et qui module à son tour des réseaux de signalisation intracellulaire contrôlant le métabolisme et la synthèse des macromolécules. Un acteur clé de ce processus est mTOR (
mechanistic target of rapamycin
), qui contrôle, chez les animaux, la croissance en réponse à divers signaux cellulaires [
1
]. Le revers de la médaille est que mTOR est aussi impliqué dans de nombreuses maladies, notamment les cancers et certains dysfonctionnements du système nerveux central, comme l’épilepsie ou le retard mental [
1
]. Il existe en fait au moins deux complexes mTOR, mTORC1 (
mTOR complex 1
) et mTORC2 (
mTOR complex 2
), qui ont des localisations, des compositions protéiques et des rôles différents. mTORC1, qui est l’objet de cette revue, joue un rôle majeur dans le métabolisme de la cellule en s’assemblant à la membrane du lysosome en réponse à la présence d’acides aminés. mTORC1 est une sérine/thréonine protéine kinase, qui phosphoryle différents substrats pour déclencher un programme de synthèse des protéines [
1
]. L’activité de son domaine catalytique est l’objet d’une régulation d’une grande complexité, qui s’effectue à la fois par l’assemblage d’un complexe multiprotéique de grande taille et au moyen de régulateurs externes.
|
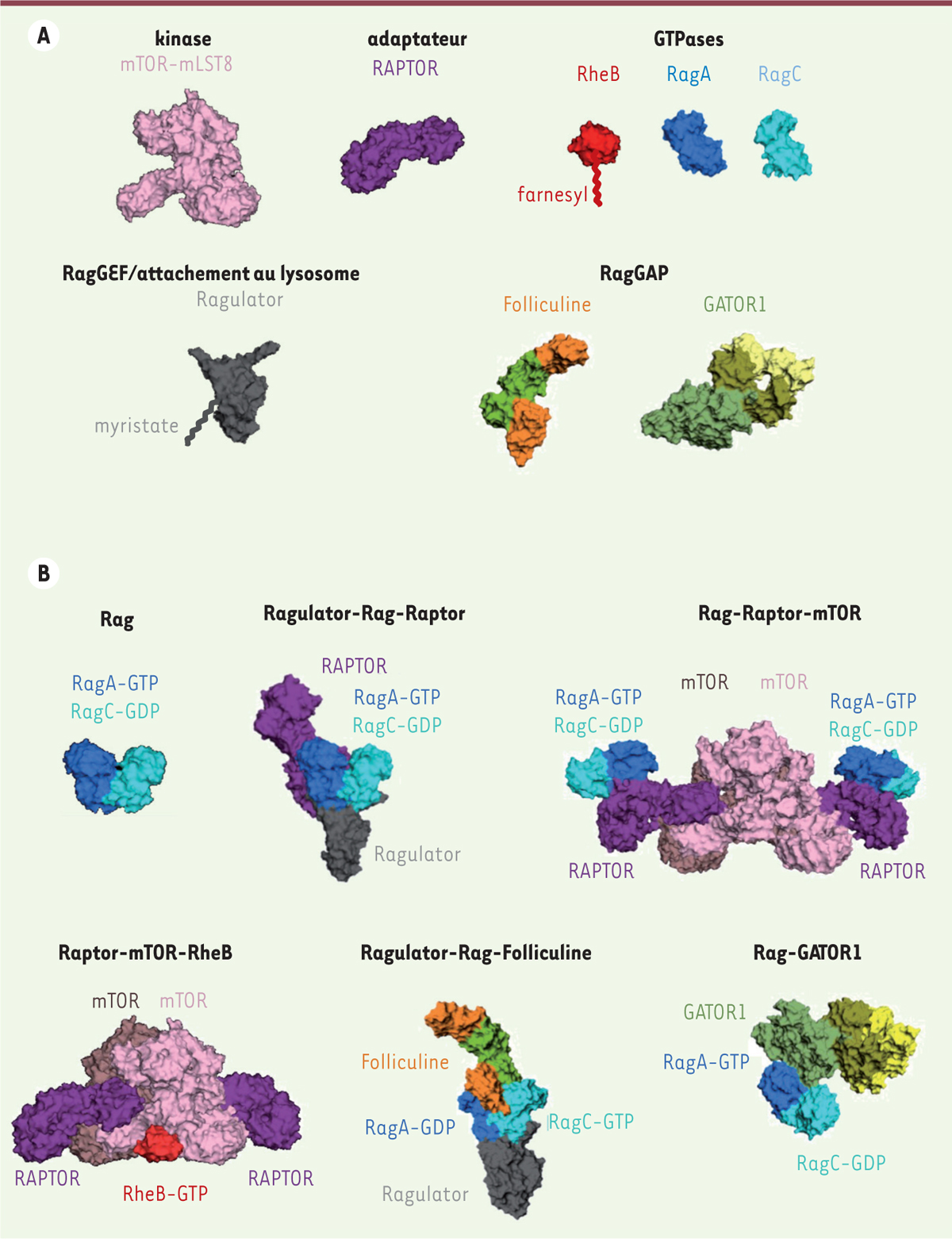
Pour former le complexe actif mTORC1, capable de phosphoryler ses substrats, un certain nombre de protéines doivent s’assembler à la surface des lysosomes
(
Figure 1A
)
. Le cœur de ce complexe est la sous-unité mTOR, qui porte l’activité kinase et qui est associée à mLST8, une protéine accessoire dont le rôle exact n’est pas bien compris [
2
]. mTOR est recruté à la membrane du lysosome par un complexe de protéines que nous appellerons « module Rag », constitué de deux GTPases (guanosine triphosphatases) régulatrices atypiques, RagA/B et RagC/D, et d’un complexe de 5 sous-unités, appelé Ragulator, qui est lié à la membrane du lysosome par une modification lipidique post-traductionnelle [
3
]. L’interaction entre mTOR et le module Rag s’effectue par l’intermédiaire d’une protéine adaptatrice, RAPTOR (
regulatory-associated protein of mTOR
), qui n’interagit avec l’hétérodimère de GTPases Rag que lorsque RagA est liée au nucléotide GTP (guanosine triphosphate) et RagC au nucléotide GDP (guanosine diphosphate) [
4
]. Ainsi, c’est l’état nucléotidique de ces deux GTPases qui contrôle la première étape de l’activation, à savoir l’adressage de mTOR à la membrane du lysosome. La sous-unité kinase peut alors interagir avec RheB, une GTPase de la superfamille des petites GTPases, qui est ancrée à la membrane du lysosome
via
un lipide, le farnesyl, lié à son extrémité C-terminale. L’interaction de mTOR avec RheB-GTP parachève alors l’activation du site catalytique quand tous les autres composants du complexe sont réunis et associés à la membrane [
5
]. Il faut noter que la sous-unité kinase étant un dimère, le complexe mTORC1 activé comprend au final 2 sites catalytiques, 6 GTPases régulatrices et 4 ancrages lipidiques à la membrane (voir plus loin).
 | Figure 1.
La structure du complexe mTORC1 en trois dimensions. A.
Les composants du complexe mTORC1 et leurs principales fonctions au sein du complexe. Les modifications post-traductionnelles par des lipides sont indiquées par des lignes brisées. Le même code couleur est utilisé pour toutes les figures.
B.
Les principales structures discutées dans cette Synthèse. La structure de l’hétérodimère RagA-GTP/RagC-GDP a été déterminée par cristallographie aux rayons X (PDB 6S6A, [
14
]), les autres structures l’ont été par cryo-microscopie électronique : Ragulator/Rag/RAPTOR (PDB 6U62, [
12
]), Rags-RAPTOR-mTOR (PDB 6SB2 , [
14
]), RAPTOR-mTOR-RheB (PDB 6BCU, [
10
]), Ragulator-Rag-folliculine (PDB 6NZD, [
15
] et 6ULG, [
13
]), Rag-GATOR1 (PDB 6CES, [
11
]). PDB :
protein data bank
(
https://www.rcsb.org
).
|
Comme toutes les GTPases régulatrices, RagA/B, RagC/D et RheB alternent entre deux formes, l’une liée au GTP et l’autre au GDP. Pour les petites GTPases, l’alternance GDP/GTP dépend en général de deux types de régulateurs : les
guanine nucleotide exchange factors
ou GEF, qui accélèrent l’échange du GDP par du GTP pour produire la forme active, et les
GTPase-activating proteins
ou GAP, qui stimulent l’hydrolyse du GTP en GDP [
6
]. Le cycle nucléotidique des GTPases Rag est plus complexe, car la forme active du module Rag, capable de recruter mTOR, contient RagA-GTP et RagC-GDP, et sa forme inactive, RagA-GDP et RagC-GTP. Des protéines à activité GEF et GAP ont néanmoins été décrites pour ces deux GTPases
(
Figure 1A
)
. Ainsi, le Ragulator a été identifié comme un GEF pour RagA [
7
], GATOR1 (
Gap activity toward Rags
), qui est constitué de 3 sous-unités, comme une GAP pour RagA [
8
] et le complexe FLCN (folliculine)-FNIP2 (
folliculin-interacting protein 2
), que nous appellerons folliculine, comme une GAP pour RagC [
9
]. Visualiser le ballet des interactions et des réarrangements structuraux qui régulent le complexe mTORC1 est une étape importante pour comprendre son fonctionnement biologique mais aussi ses dysfonctionnements au cours des maladies. La cristallographie aux rayons X et la cryo-microscopie électronique (cryo-EM) constituent deux approches puissantes pour cela. La cristallographie permet d’obtenir des informations d’une grande précision à l’échelle atomique, et constitue de ce fait une approche de choix pour observer le détail des structures. La nécessité d’obtenir des cristaux peut cependant s’avérer une limitation importante. C’est particulièrement le cas lorsque les complexes sont de grande taille ou que leurs composants présentent une flexibilité structurale, ce qui est souvent le cas dans des complexes périmembranaires finement régulés, tels que mTORC1. Depuis quelques années, d’immenses progrès méthodologiques et instrumentaux ont été réalisés en cryo-EM, une révolution qui a été saluée par le prix Nobel de Chimie attribué en 2017 à Jacques Dubochet, Joachim Frank, et Richard Henderson. Ils permettent aujourd’hui de déterminer la structure d’assemblages complexes avec une très bonne résolution. Ces deux méthodes ont été combinées au cours des trois dernières années pour déterminer de nombreuses structures de composants de la machinerie mTORC1
(
Figure 1B
)
[
10
-
16
]. Ces structures éclairent de nombreux aspects de la régulation du complexe mTORC1 par les GTPases Rag et RheB, et de la régulation de ces GTPases elles-mêmes.
|
Les GTPases Rag au cœur de la régulation de mTORC1
Les GTPases Rag sont des GTPases atypiques, constituées d’un domaine GTPase, apparenté aux petites GTPases de la superfamille de l’oncogène p21Ras, suivi d’un domaine structural C-terminal connu sous le nom de domaine
Roadblock
. La structure de l’hétérodimère RagA/RagC a été déterminée dans un grand nombre de conditions : par cristallographie, sous forme Rag GTP/RagC-GDP isolée [
14
], et par cryo-EM, associé à l’adaptateur RAPTOR isolé [
12
], au sein du complexe pré-activé lié à la sous-unité kinase mTOR [
14
], et en complexe avec les GAP GATOR1 [
11
] et folliculine [
13
,
15
]. Ces structures montrent que RagA et RagC forment un hétérodimère par l’intermédiaire de leurs domaines
Roadblock
C-terminaux, avec une interface insensible à la nature des nucléotides, alors que leurs domaines GTPase ont des structures et des écartements différents en fonction du nucléotide
(
Figure 2A
)
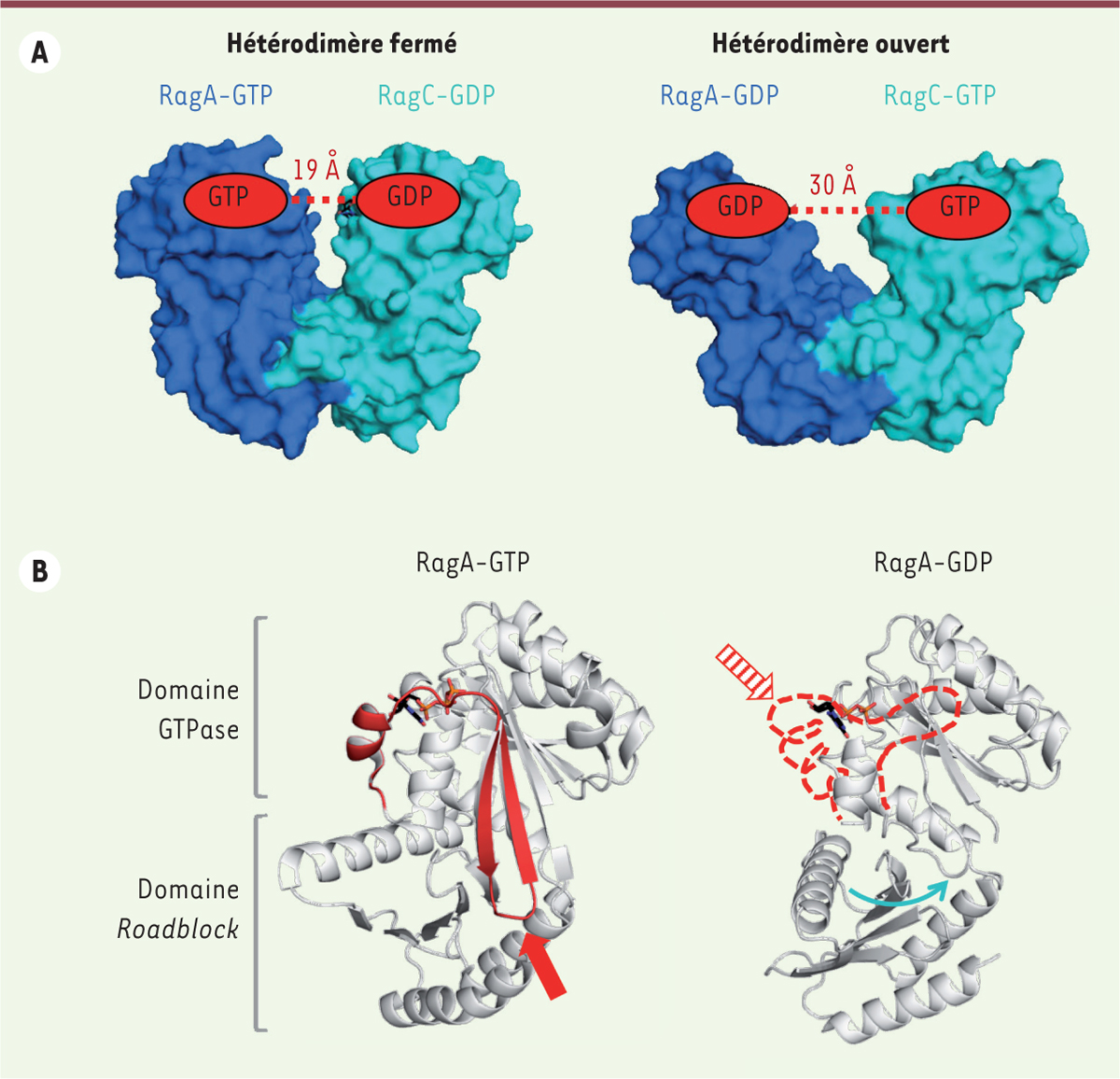
. Ces différences de conformation et d’écartement sont importantes pour l’assemblage du complexe mTORC1 actif. Notamment, plusieurs régions impliquées dans la reconnaissance du nucléotide alternent entre une conformation assez rigide dans la forme associée au GTP, ce qui la rend favorable aux interactions protéine-protéine, et une conformation très flexible dans la forme associée au GDP, qui ne leur est pas favorable. La forme active RagA-GTP/RagC-GDP est la plus fermée. Cela permet de former un contact bien ajusté avec l’adaptateur RAPTOR et de recruter ainsi mTOR à la membrane. En revanche, la forme inactive RagA-GDP/RagC-GTP est largement ouverte, empêchant la formation d’une interaction stable.
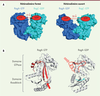 | Figure 2.
L’état nucléotidique des GTPases Rag contrôle l’assemblage du complexe mTORC1. A.
La largeur de l’ouverture entre les domaines GTPases au sein de l’hétérodimère RagA/RagC dépend des nucléotides GDP et GTP. La forme la plus fermée fixe l’adaptateur RAPTOR, ce qui conduit au recrutement de mTOR à la membrane du lysosome, alors que la forme ouverte défavorise cette interaction.
B.
Dans la forme liée au GTP (PDB 6S6A, [
14
]), la région switch 1 interagit fortement avec le nucléotide et la région interswitch traverse le domaine GTPase entre le site nucléotidique et le domaine
Roadblock
(en rouge). Dans la forme liée au GDP (PDB 6ULG, [
13
]), ces deux régions sont très flexibles et désordonnées dans la structure (en pointillé rouge). Cette transition entre un état structuré, qui pousse sur le domaine
Roadblock
(flèche rouge pleine), et une forme flexible, qui relâche la pression (flèche rouge hachurée), explique comment le nucléotide agit à distance pour modifier la position du domaine
Roadblock
par rapport au domaine GTPase (flèche bleue).
|
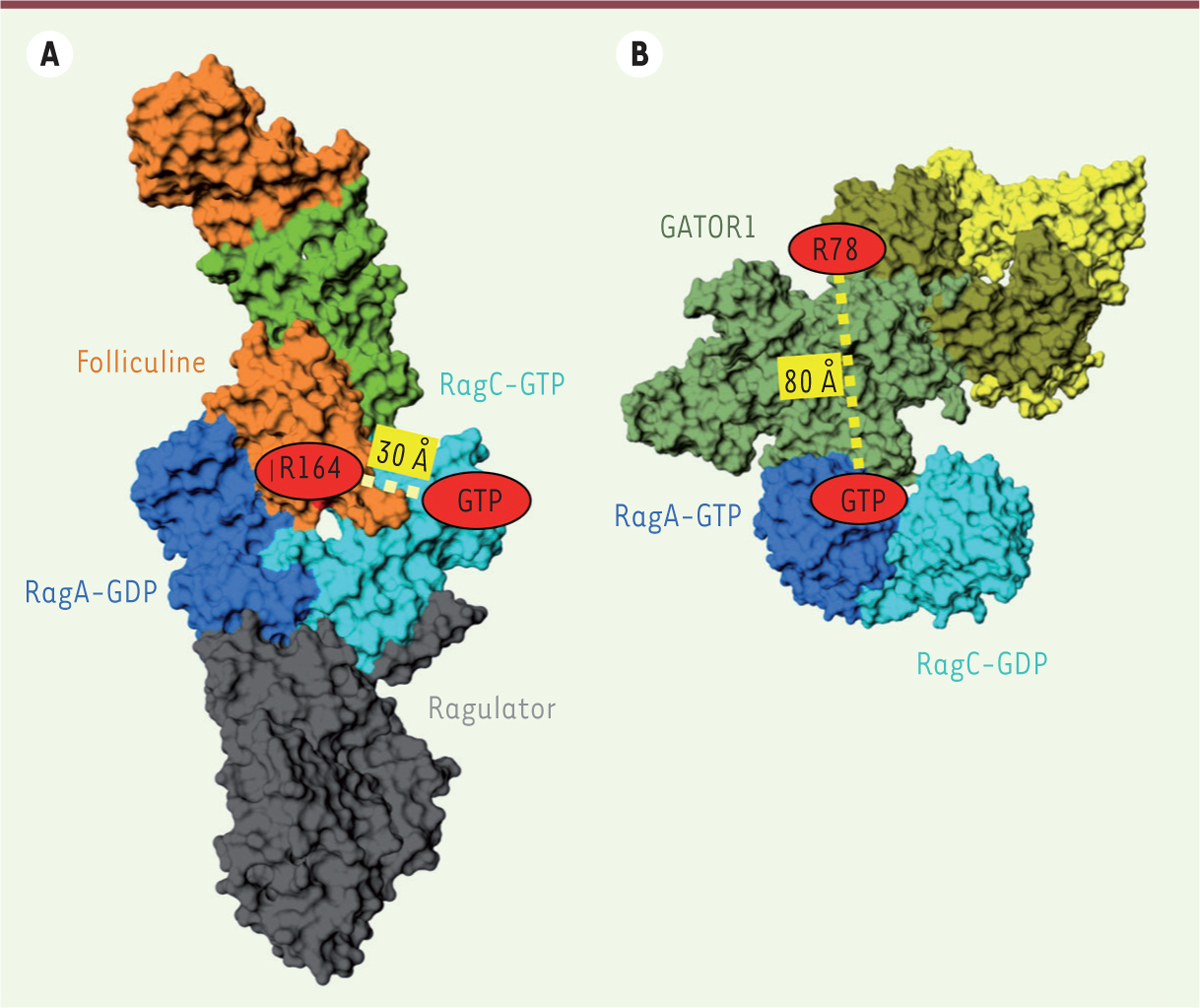
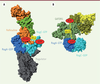 | Figure 3.
Les conformations des complexes Rag-GAP ne sont pas compatibles avec une activité GAP.
L’arginine 164 de la folliculine (PDB 6ULG, [
13
]) (A) et l’arginine 78 de GATOR1 (PDB 6CES, [
11
]) (B), qui sont nécessaires à ces GAP pour stimuler l’hydrolyse du GTP, sont trop éloignées pour interagir avec le GTP, suggérant que ces complexes ne correspondent pas à la conformation compétente.
|
L’écartement des domaines GTPases est déterminé par leur position relative par rapport aux domaines
Roadblock
. Une question intrigante est donc de savoir comment les domaines GTPase ajustent leurs positions par rapport aux domaines
Roadblock
, alors que le site nucléotidique n’est pas en contact direct avec ces domaines. La réponse se trouve probablement dans un mécanisme allostérique (à distance) qui a été bien décrit pour les petites GTPases de la famille Arf, auxquelles le domaine GTPase des Rag est plus particulièrement apparenté [
17
,
18
]. Les GTPases Arf possèdent en effet un élément structural qui traverse le domaine GTPase et permet au site nucléotidique de « dialoguer » avec la partie située à l’opposé dans la structure [
19
]. Cet élément fonctionne comme un « bouton-pression », avec une position rétractée et une position exposée en fonction du nucléotide fixé, le GDP ou le GTP. La comparaison des différentes structures permet de suggérer qu’un tel élément structural existe également dans le domaine GTPase de RagA et RagC, et qu’il interagit avec le domaine
Roadblock
[
12
,
14
]. Ainsi, dans sa position exposée, cet élément exerce une pression sur le domaine
Roadblock
et peut ainsi modifier la position relative des deux domaines
(
Figure 2B
)
. Un même mécanisme allostérique pourrait ainsi être mobilisé par le Ragulator pour exercer son activité GEF sur RagA, alors que celui-ci interagit avec le domaine
Roadblock
à distance du site du nucléotide [
7
,
20
,
21
]. Cependant, le mécanisme utilisé par le Ragulator reste mystérieux. Les structures montrent en effet que son interface avec les domaines
Roadbloack
est identique quels que soient les nucléotides, et cela même dans des structures qui ne contiennent que les domaines
Roadblock
[
20
,
22
]. Autre possibilité, l’échange GDP/GTP de RagA pourrait être constitutif et d’autres types de régulateurs pourraient intervenir pour l’inhiber plutôt que pour le stimuler, un mécanisme déjà proposé pour la région cytoplasmique de SLC38A9 (
sodium-coupled neutral amino acid transporter 9
), dont la structure liée à RagA-GTP/RagC-GDP vient d’être décrite [
16
].
|
La régulation des GTPases Rag par leurs GAP n’a pas encore livré tous ses secrets
Une autre régulation de l’hétérodimère RagA/RagC est celle exercée par les GAP : GATOR1 hydrolyse le GTP sur RagA et favorise ainsi l’inactivation de mTORC1 par dissociation du complexe ; la folliculine hydrolyse le GTP sur RagC et est nécessaire à l’activation de mTORC1. Les structures des complexes RagC-GDP/RagA-GTP/GATOR1 [
11
] et RagA-GDP/RagC-GTP/folliculine/Ragulator [
13
,
15
] ont récemment été déterminées par cryo-EM. Ces deux GAP, chacune composée de plusieurs sous-unités, ne présentent aucune ressemblance structurale entre elles, et fixent l’hétérodimère RagA/RagC de façon très différente
(
Figures 3A,B
)
. Par exemple, la folliculine s’insère dans la cavité entre les deux domaines GTPases, alors que GATOR1 forme une interaction latérale avec RagA. Étonnamment, ni la folliculine ni GATOR1 ne positionnent d’éléments à proximité des sites nucléotidiques susceptibles de favoriser l’hydrolyse du GTP. C’est le cas de nombreuses GAP des petites GTPases qui stimulent l’hydrolyse du GTP en insérant une arginine dans le site nucléotidique [
6
], une arginine nécessaire à l’activité GAP a été identifiée dans chacune de ces deux GAP [
11
,
13
,
23
]. Cependant, aussi bien pour GATOR1 (Arg 78) que pour la folliculine (Arg 164), cette arginine est située beaucoup trop loin du GTP pour pouvoir intervenir directement dans la réaction
(
Figures 3A, B
)
. Ce paradoxe suggère que les modes d’interaction de GATOR1 et de la folliculine observés en cryo-EM ne correspondent pas aux structures compétentes pour l’activité GAP. Le corollaire est que ces deux régulateurs doivent avoir un second mode d’interaction, très différent, qui leur permet d’agir comme des GAP. À l’appui de cette hypothèse, GATOR1, qui n’a pas d’activité GAP
in vitro
, retrouve cette activité à la suite de mutations empêchant le mode d’interaction qui a été visualisé dans la structure [
13
]. Les interactions de GATOR1 et de la folliculine avec les GTPases Rag observées par cryo-EM pourraient donc être responsables de l’inhibition de l’activité GAP. Le fait que seule cette conformation soit observée en cryo-EM reste cependant mystérieux. Nous suggérons une alternative non exclusive, par analogie avec d’autres GTPases, dont les GAP agissent via des mécanismes allostériques, comme les protéines G hétérotrimériques et leurs GAP de la famille RGS [
24
], ou comme la petite GTPase de la bactérie
Myxococcus xanthus
, MglA, et sa GAP, MglB [
25
]. Dans ces systèmes, l’arginine nécessaire à l’hydrolyse du GTP est localisée dans le domaine GTPase, et la GAP interagit à distance pour favoriser son bon positionnement. RagA et RagC possèdent des arginines au sein de leur site nucléotidique. Il serait intéressant d’explorer le rôle de ces arginines dans l’hydrolyse du GTP.
|
Assemblage et régulation du complexe mTORC1 à la membrane du lysosome : vers la reconstitution du puzzle
Pour achever son activation, le complexe mTORC1, préalablement associé au lysosome grâce au module Rag, recrute la petite GTPase RheB, qui est localisée sous forme active (liée au GTP) à la surface du lysosome. La structure cristallographique de la sous-unité kinase mTOR isolée [
26
], et les structures en cryo-EM de mTOR associé à RagA-GTP/RagC-GDP/RAPTOR [
12
] et à Rheb-GTP/RAPTOR [
10
], fournissent des informations importantes sur le mécanisme d’action de RheB. Elles montrent que le module Rag n’est pas suffisant pour activer mTOR, et que cette activation est contrôlée par RheB-GTP. RheB-GTP interagit avec les domaines non catalytiques de mTOR, ce qui induit un changement de leur position par rapport au domaine catalytique et renforce les interactions avec RAPTOR. Ce remodelage structural se propage jusqu’au site catalytique, où il induit de subtils réarrangements des résidus catalytiques qui peuvent expliquer comment RheB stimule l’activité kinase de façon allostérique.
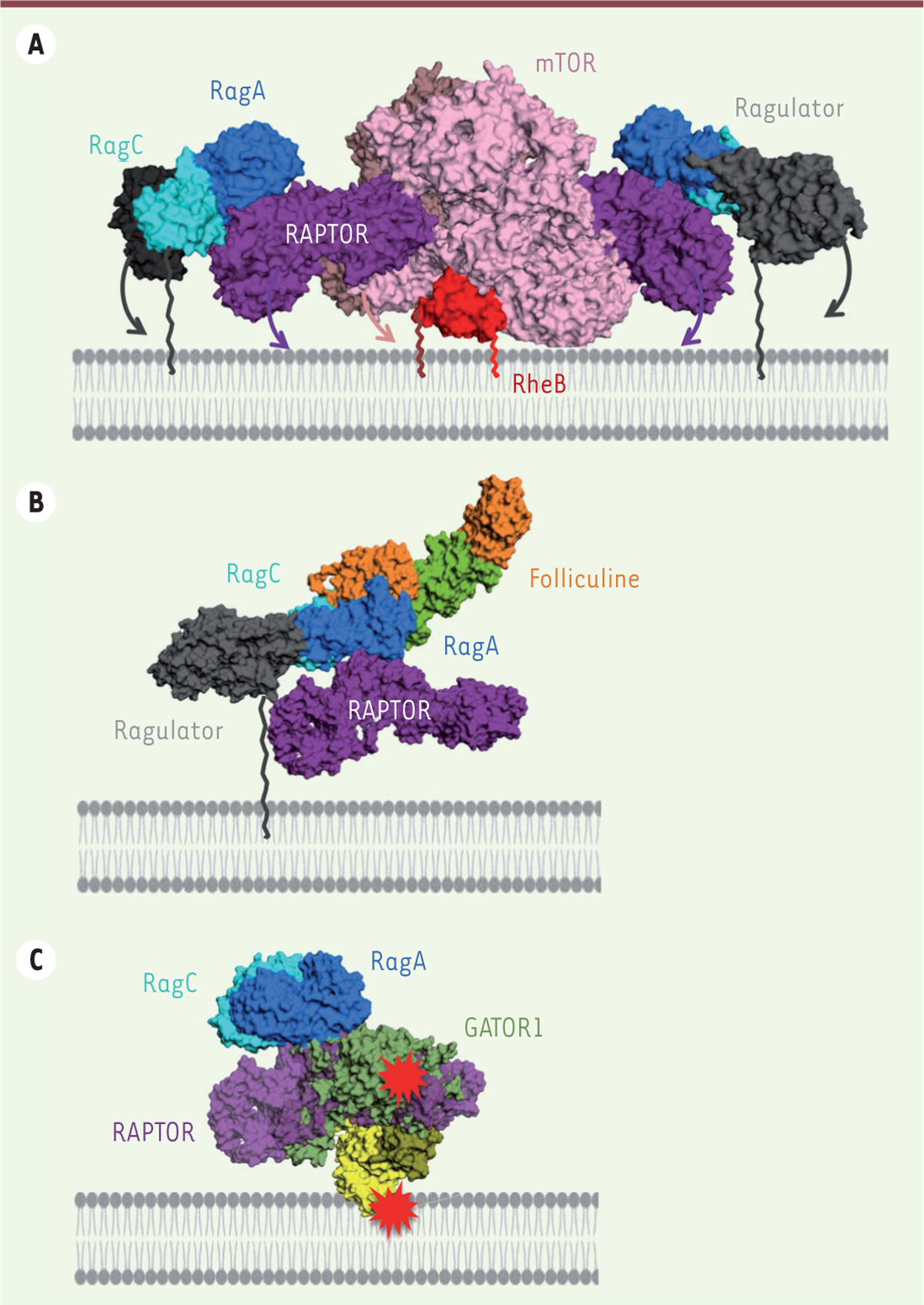
Toutes les structures partielles du complexe mTORC1 que nous venons de décrire
(
Figure 1
)
partagent des sous-unités communes, qui peuvent donc être superposées entre elles pour assembler un modèle quasi-atomique du complexe mTORC1 activé
(
Figure 4A
)
[
14
]. Quelles peuvent être les implications possibles de ce modèle dans un contexte membranaire ? Grâce aux quatre ancrages lipidiques (deux dans Ragulator et deux dans RheB), ce modèle peut être orienté de façon relativement précise par rapport à la surface de la membrane lysosomiale
(
Figure 4A
)
[
14
]. Il est frappant de constater que le complexe expose une surface très étendue et relativement plane vers la membrane. Il est donc probable qu’en plus des ancrages lipidiques, le complexe mTORC1 activé forme de multiples interactions directes avec la membrane. L’exemple des régulateurs de petites GTPases a montré que leurs interactions à la périphérie de la membrane induisent très souvent des changements de conformation qui modifient et régulent leur activité [
6
,
27
]. Il est donc tout à fait plausible que la surface de la membrane fasse plus que servir de plate-forme à l’assemblage du complexe mTORC1 actif, et contribue aussi à induire des changements conformationnels plus importants que ceux observés par cristallographie ou par cryo-EM en l’absence de la membrane. Ainsi, la membrane pourrait par exemple « pousser » sur les domaines non-catalytiques de la sous-unité kinase mTOR ou « tirer » sur le Ragulator pour induire des réarrangements structuraux amplifiant l’activité kinase (
Figure 4A
, flèches). Les structures déterminées en cryo-EM suggèrent qu’il existe une certaine flexibilité au sein de la sous-unité kinase [
10
], ce qui pourrait être compatible avec ces mouvements et les favoriser.
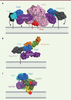 | Figure 4.
Architecture et régulation de mTOC1 à la membrane du lysosome : de nouvelles perspectives ouvertes par l’élucidation des structures. A.
Modèle quasi-atomique du complexe mTORC1 activé, à la membrane du lysosome, reconstitué à partir de structures partielles du complexe [
10
,
14
]. Le complexe étant un dimère, il est ancré à la membrane par quatre modifications lipidiques (indiquées par des lignes brisées). La surface de mTORC1 qui fait face à la membrane est étendue et assez plane, ce qui suggère que le complexe pourrait former des interactions directes avec la membrane. Ces interactions pourraient à leur tour induire des mouvements au sein du complexe (flèches), amplifiant ainsi l’activation.
B.
La superposition du complexe Ragulator/Rag/RAPTOR (PDB 6U62, [
12
]) avec le complexe Rag/folliculine (PDB 6ULG, [
13
]) montre que la folliculine est compatible avec l’assemblage du complexe mTORC1 à la membrane, suggérant la possibilité d’un effet « feedforward » au cours duquel tous les composants sont présents simultanément.
C.
La superposition du complexe Ragulator/Rag/RAPTOR avec le complexe Rag/GATOR1 (PDB 6CES [
11
]) montre que la fixation de GATOR1 n’est pas compatible avec celle de RAPTOR, ni potentiellement avec l’association de l’hétérodimère Rag avec la membrane, posant la question de l’environnement dans lequel GATOR1 exerce son action. Les régions en conflit stérique sont indiquées par le symbole rouge.
|
Des études récentes ont montré que plusieurs protéines kinases dimériques, telles que le récepteur de l’EGF (
epidermal growth factor
) [
28
] ou, plus récemment, la protéine kinase B-Raf [
29
], adoptent des structures asymétriques au sein du dimère, et que cette asymétrie joue un rôle important dans les mécanismes de leur autorégulation. Un autre exemple récent d’asymétrie structurale, mais aussi fonctionelle, est celui d’ARNO, un GEF pour les GTPases Arf, qui forme un dimère et doit être associé à la membrane pour activer la GTPase. L’analyse en microscopie électronique montre qu’ARNO a une structure flexible et asymétrique [
30
]. La reconstitution de son activité GEF dans des membranes artificielles révèle qu’il n’interagit avec la membrane que par un seul monomère à la fois, ce qui pourrait permettre au second monomère d’explorer plus facilement la membrane à la recherche d’une nouvelle molécule Arf à activer [
30
]. Une telle asymétrie est possible dans le complexe mTORC1, par exemple si une seule sous-unité kinase est recrutée à la membrane par le module Rag et activée par RheB-GTP, l’autre sous-unité kinase restant dans une conformation inactive. Les structures de mTOR inactif [
14
] et actif [
10
] seraient compatibles avec une organisation asymétrique hémi-activée au prix de réarrangements structuraux minimes, ce qui pourrait correspondre à un état d’activité intermédiaire de mTORC1.
Finalement, par analogie avec les petites GTPases, pour lesquelles l’influence des membranes est de mieux en mieux comprise [
6
,
27
], il est plausible que les membranes contribuent directement à la régulation des GEF (Ragulator) et GAP (GATOR1 et folliculine) des GTPases Rag. On peut ainsi faire plusieurs observations en considérant l’hétérodimère RagA/RagC en complexe avec ses GAP dans le contexte de la membrane. La première est que le mode d’interaction de la folliculine, qui inactive RagC et contribue donc, en principe, à activer mTORC1, est compatible avec le complexe activé, mais aussi avec l’environnement membranaire
(
Figure 4B
)
. Bien que de nombreuses questions restent posées quant au mode effectif d’interaction de la folliculine, comme nous l’avons discuté plus haut, cette observation suggère un mécanisme de type
feedforward
1
, dans lequel la folliculine favoriserait directement la formation du complexe actif. Inversement, GATOR1, qui inactive RagA et donc mTORC1, en compétition directe avec l’adaptateur RAPTOR, percuterait probablement la membrane
(
Figure 4C
)
. Ainsi, ce mode d’interaction ne semble possible que si l’hétérodimère RagA/RagC est à la fois dissocié de RAPTOR/mTOR et éloigné de la membrane, impliquant que le processus d’inactivation pourrait déjà avoir été déclenché par d’autres mécanismes au moment où la GAP intervient.
|
Les multiples données structurales obtenues récemment permettent désormais de visualiser en détail la structure de chaque composant du complexe mTORC1, et de comprendre la façon dont ces composants s’organisent entre eux et comment l’information se propage de façon allostérique au sein de cet immense complexe protéique. Elles constituent ainsi un socle de connaissances sans précédent pour comprendre la transmission de signaux en amont et en aval de mTORC1, par exemple par les senseurs d’acides aminés ou par les régulateurs de RheB. Certains mystères n’ont pas encore livré leurs secrets ; il reste notamment à élucider la façon dont les GTPases Rag sont régulées par leurs GEF et leurs GAP. Dans le futur, la prise en compte de la machinerie mTORC1 dans un environnement membranaire, inspirée par des systèmes apparentés, comme les petites GTPases ou d’autres kinases, devrait ouvrir de nouvelles questions passionnantes auxquelles il sera important d’apporter des réponses pour comprendre comment mTORC1 fonctionne dans un contexte physiologique [
14
]. L’une des approches possibles, qui s’est révélée remarquablement fructueuse pour les petites GTPases, serait notamment de reconstituer la machinerie mTORC1 dans des membranes artificielles afin d’évaluer l’impact de la membrane sur ses activités régulatrices et catalytiques. Les réponses à ces questions seront d’un grand intérêt pour comprendre les mécanismes défectueux et les effets des mutations dans les maladies, et identifier les talons d’Achille qui peuvent être ciblés dans des approches thérapeutiques.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Ce travail a été financé par le CNRS, l’École normale supérieure Paris-Saclay et par un contrat de l’Agence Nationale de la Recherche obtenu par J.C. (numéro 18-CE11-0013-01).
|
Footnotes |
1.
Sabatini
DM
.
Twenty-five years of mTOR: uncovering the link from nutrients to growth.
.
Proc Natl Acad Sci USA.
2017;
;
114
:
:11818.
–
11825
.
2.
Kim
DH
,
Sarbassov
DD
,
Ali
SM
,
et al.
GbetaL, a positive regulator of the rapamycin-sensitive pathway required for the nutrient-sensitive interaction between raptor and mTOR.
.
Mol Cell.
2003;
;
11
:
:895.
–
904
.
3.
Sancak
Y
,
Bar-Peled
L
,
Zoncu
R
,
et al.
Ragulator-Rag complex targets mTORC1 to the lysosomal surface and is necessary for its activation by amino acids.
.
Cell.
2010;
;
141
:
:290.
–
303
.
4.
Kim
DH
,
Sarbassov
DD
,
Ali
SM
,
et al.
mTOR interacts with raptor to form a nutrient-sensitive complex that signals to the cell growth machinery.
.
Cell.
2002;
;
110
:
:163.
–
175
.
5.
Nicastro
R
,
Sardu
A
,
Panchaud
N
,
De Virgilio
C
.
The architecture of the Rag GTPase signaling network.
.
Biomolecules.
2017;
;
7
:
:48.
.
6.
Cherfils
J
,
Zeghouf
M
.
Regulation of small GTPases by GEFs, GAPs, and GDIs.
.
Physiol Rev.
2013;
;
93
:
:269.
–
309
.
7.
Bar-Peled
L
,
Schweitzer
LD
,
Zoncu
R
,
Sabatini
DM
.
Ragulator is a GEF for the rag GTPases that signal amino acid levels to mTORC1.
.
Cell.
2012;
;
150
:
:1196.
–
1208
.
8.
Bar-Peled
L
,
Chantranupong
L
,
Cherniack
AD
,
et al.
A Tumor suppressor complex with GAP activity for the Rag GTPases that signal amino acid sufficiency to mTORC1.
.
Science.
2013;
;
340
:
:1100.
–
1106
.
9.
Tsun
ZY
,
Bar-Peled
L
,
Chantranupong
L
,
et al.
The folliculin tumor suppressor is a GAP for the RagC/D GTPases that signal amino acid levels to mTORC1.
.
Mol Cell.
2013;
;
52
:
:495.
–
505
.
10.
Yang
H
,
Jiang
X
,
Li
B
,
et al.
Mechanisms of mTORC1 activation by RHEB and inhibition by PRAS40.
.
Nature.
2017;
;
552
:
:368.
–
373
.
11.
Shen
K
,
Huang
RK
,
Brignole
EJ
,
et al.
Architecture of the human GATOR1 and GATOR1-Rag GTPases complexes.
.
Nature.
2018;
;
556
:
:64.
–
69
.
12.
Rogala
KB
,
Gu
X
,
Kedir
JF
,
et al.
Structural basis for the docking of mTORC1 on the lysosomal surface.
.
Science.
2019;
;
366
:
:468.
–
475
.
13.
Shen
K
,
Rogala
KB
,
Chou
HT
,
et al.
Cryo-EM Structure of the human FLCN-FNIP2-Rag-Ragulator complex.
.
Cell.
2019;
;
179
:
:1319.
–
29e8
.
14.
Anandapadamanaban
M
,
Masson
GR
,
Perisic
O
,
et al.
Architecture of human Rag GTPase heterodimers and their complex with mTORC1.
.
Science.
2019;
;
366
:
:203.
–
210
.
15.
Lawrence
RE
,
Fromm
SA
,
Fu
Y
,
et al.
Structural mechanism of a Rag GTPase activation checkpoint by the lysosomal folliculin complex.
.
Science.
2019;
;
366
:
:971.
–
977
.
16.
Fromm
SA
,
Lawrence
RE
,
Hurley
JH
.
Structural mechanism for amino acid-dependent Rag GTPase nucleotide state switching by SLC38A9.
.
Nat Struct Mol Biol.
2020;
;
27
:
:1017.
–
20
.
17.
Klinger
CM
,
Spang
A
,
Dacks
JB
,
Ettema
TJ
.
Tracing the archaeal origins of eukaryotic membrane-trafficking system building blocks.
.
Mol Biol Evol.
2016;
;
33
:
:1528.
–
1541
.
18.
Cherfils
J
.
Encoding Allostery in mTOR Signaling: The Structure of the Rag GTPase/Ragulator Complex.
.
Mol Cell.
2017;
;
68
:
:823.
–
824
.
19.
Pasqualato
S
,
Renault
L
,
Cherfils
J
.
Arf, Arl, Arp and Sar proteins: a family of GTP-binding proteins with a structural device for front-back communication.
.
EMBO Rep.
2002;
;
3
:
:1035.
–
1041
.
20.
Su
MY
,
Morris
KL
,
Kim
DJ
,
et al.
Hybrid structure of the RagA/C-Ragulator mTORC1 activation complex.
.
Mol Cell.
2017;
;
68
:
:835.
–
46e3
.
21.
Shen
K
,
Sabatini
DM
.
Ragulator and SLC38A9 activate the Rag GTPases through noncanonical GEF mechanisms.
.
Proc Natl Acad Sci USA.
2018;
;
115
:
:9545.
–
9550
.
22.
de Araujo
MEG
,
Naschberger
A
,
Furnrohr
BG
,
et al.
Crystal structure of the human lysosomal mTORC1 scaffold complex and its impact on signaling.
.
Science.
2017;
;
358
:
:377.
–
381
.
23.
Shen
K
,
Valenstein
ML
,
Gu
X
,
Sabatini
DM
.
Arg-78 of Nprl2 catalyzes GATOR1-stimulated GTP hydrolysis by the Rag GTPases.
.
J Biol Chem.
2019;
;
294
:
:2970.
–
2975
.
24.
Tesmer
JJ
,
Berman
DM
,
Gilman
AG
,
Sprang
SR
.
Structure of RGS4 bound to AlF4–activated G(i alpha1): stabilization of the transition state for GTP hydrolysis.
.
Cell.
1997;
;
89
:
:251.
–
261
.
25.
Galicia
C
,
Lhospice
S
,
Varela
PF
,
et al.
MglA functions as a three-state GTPase to control movement reversals of Myxococcus xanthus.
.
Nat Commun.
2019;
;
10
:
:5300.
.
26.
Yang
H
,
Rudge
DG
,
Koos
JD
,
et al.
mTOR kinase structure, mechanism and regulation.
.
Nature.
2013;
;
497
:
:217.
–
223
.
27.
Peurois
F
,
Peyroche
G
,
Cherfils
J
.
Small GTPase peripheral binding to membranes: molecular determinants and supramolecular organization.
.
Biochem Soc Trans.
2019;
;
47
:
:13.
–
22
.
28.
Kovacs
E
,
Zorn
JA
,
Huang
Y
,
et al.
A structural perspective on the regulation of the epidermal growth factor receptor.
.
Annu Rev Biochem.
2015;
;
84
:
:739.
–
764
.
29.
Kondo
Y
,
Ognjenovic
J
,
Banerjee
S
,
et al.
Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases.
.
Science.
2019;
;
366
:
:109.
–
115
.
30.
Das
S
,
Malaby
AW
,
Nawrotek
A
,
et al.
Structural organization and dynamics of homodimeric cytohesin family Arf GTPase exchange factors in solution and on membranes.
.
Structure.
2019;
;
27
:
:1782.
–
97e7
.
|