| |
| Med Sci (Paris). 37(3): 230–233. doi: 10.1051/medsci/2021007.Les virus géants et l’origine des ARN polymérases des eucaryotes Patrick Forterre1,2* and Morgan Gaia3** 1Département de microbiologie, Institut Pasteur
,
25 rue du Docteur-Roux
,
75015Paris
,
France
.
2Institut de biologie intégrative de la cellule (I2BC), Département de microbiologie, CEA, CNRS, Université Paris-Saclay
,
91198Gif-sur-Yvette
,
France
.
3Génomique Métabolique, Genoscope, Institut François Jacob, CEA, CNRS, Univ. Évry, Université Paris-Saclay
,
91057Évry
,
France
.
MeSH keywords: DNA-directed RNA polymerases, Cellules eucaryotes, Virus géants, enzymologie, génétique |
La découverte en 2003 du premier virus géant, le
Mimivirus
, dont le génome a une taille de 1,2 Mb, a soulevé de très nombreuses questions, en particulier sur l’origine de ces virus et leur place dans le monde vivant [
1
, [
2
]. Bien que ces virus puissent sembler fondamentalement différents des autres, ce n’est pas le cas :
Mimivirus
et les autres virus géants font partie d’un même phylum groupant plusieurs familles de virus à ADN, les
Nucleocytoviricota
, communément appelés NCLDV (
nucleocytoplasmic large DNA viruses
) [
3
]. Ces virus sont tous de grande taille, les plus petits correspondant à la famille du virus de la variole, dont le génome est « seulement » dix fois plus petit que celui des géants. Les différentes familles de NCLDV infectent les eucaryotes les plus divers (animaux, algues, protistes), ce qui suggère une origine très ancienne dans l’évolution des espèces. Nous avons récemment pu le confirmer en réalisant des analyses phylogénétiques approfondies de ces virus [
4
]. De plus, en analysant leurs ARN polymérases (ARN Pol) et les ARN Pol cellulaires, notre étude a montré que deux des trois ARN Pol des eucaryotes, les ARN pol I et II, sont probablement d’origine virale.
|
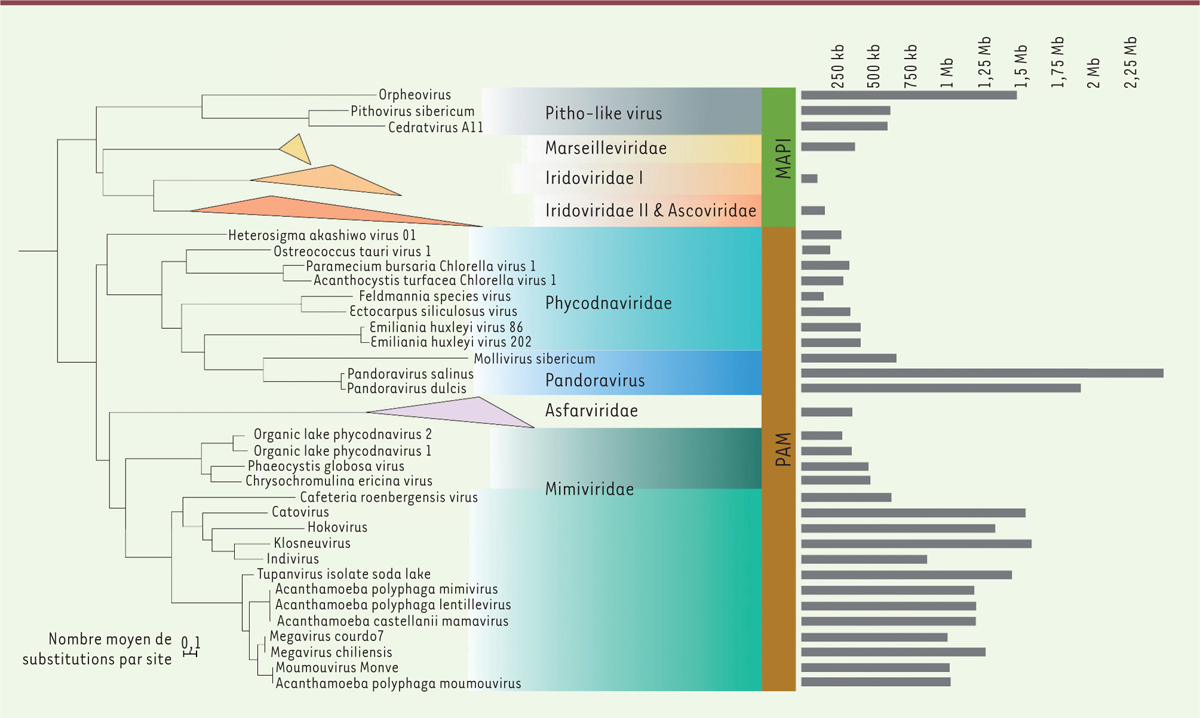
Une phylogénie de référence pour les NCLDV
L’analyse phylogénétique de huit gènes marqueurs conservés chez presque tous les NCLDV a permis d’organiser les différentes familles de NCLDV en deux grands groupes, que nous avons désignés d’après les initiales de ces familles : les groupes MAPI (pour
Marseilleviridae, Ascoviridae
, Pitho-like viruses,
Iridoviridae
) et PAM (pour
Phycodnaviridae
,
Asfarviridae
,
Mimiviridae
) [
4
]
(
Figure 1
).
Nous avons préféré exclure la famille des
Poxviridae
, comprenant les virus de la variole et de la vaccine, car les protéines de ces virus évoluent trop rapidement pour pouvoir les placer avec certitude dans une phylogénie. Nos analyses préliminaires avaient d’ailleurs suggéré que leur inclusion pouvait être source d’artéfacts.
 | Figure 1.
Phylogénie des virus Nucleocytoviricota (NCLDV) fondée sur une analyse de la concaténation de 8 gènes marqueurs, présents chez la plupart des NCLDV, codant les deux grandes sous-unités de l’ARN polymérase, l’ADN polymérase, l’ADN primase/hélicase, la protéine majeure de capside, l’ATPase d’encapsidation, et les facteurs de transcription TF2S et VLTF3.
Les tailles moyennes des génomes des virus appartenant aux différentes familles de NCLDV sont indiquées à droite. MAPI : groupe de virus NCLDV comportant les
Marseilleviridae,
les
Ascoviridae
, les
Pitho-like viruses
, et les
Iridoviridae
; PAM : groupe de virus NCLDV comportant les
Phycodnaviridae
, les
Asfarviridae
, et les
Mimiviridae
.
|
|
Trois apparitions indépendantes du gigantisme chez les NCLDV
Notre étude a montré que le gigantisme était apparu au moins trois fois indépendamment au cours de l’évolution des NCLDV : les
Mimiviridae
et les Pandoravirus (dont les génomes sont encore deux fois plus grands que ceux des Mimivirus et qui font partie des
Phycodnaviridae
) sont devenus géants indépendamment au cours de l’évolution des PAM, tandis qu’
Orpheovirus
(du groupe des Pitho-like viruses), un autre virus géant, serait apparu au cours de l’évolution des MAPI
(
Figure 1
)
. Les virus géants ne sont donc pas les descendants de cellules qui seraient devenues des virus, mais ont évolué à partir de virus plus petits, peut-être en même temps que la taille de leurs hôtes augmentait. Ces derniers, des protistes unicellulaires de grande taille, tels que les amibes du genre
Acanthamoeba,
ont en effet l’habitude de phagocyter des bactéries pour se nourrir. En acquérant une taille sensiblement identique à celle d’une petite bactérie, les virus géants pourraient tromper leurs proies qui, agissant en prédateurs, vont les ingérer et se faire infecter par la même occasion [
5
]. Ces virus chercheraient donc à « mimer » une bactérie, justifiant le nom de Mimivirus donné au premier d’entre eux, ainsi que le nom
Imitervirales
attribué récemment par l’ICTV (
international committee on taxonomy of viruses
) à l’ordre dans lequel se trouve la famille des
Mimiviridae
. Tandis que les bactéries ou les archées parasites intracellulaires évoluent toujours par régression, il semble donc que les virus, bien qu’étant des parasites obligatoires, peuvent parfois évoluer en devenant plus complexes. Toutes les familles et sous-familles de NCLDV possèdent en effet de nombreux gènes qui leur sont spécifiques et qui sont sans doute apparus après leur diversification, comme cela a été montré dans le cas des Pandoravirus [
6
]. Cette complexification des virus, pourtant parasites, pourrait venir du fait qu’ils ne se contentent pas d’exploiter les cellules qu’ils infectent, mais les transforment de l’intérieur en cellules virales pouvant parfois bénéficier de l’apport de nouveaux gènes codés dans le génome du virus et qui vont augmenter la productivité et l’efficacité du cycle viral.
L’origine des NCLDV reste mystérieuse. La plupart de ces virus possèdent des protéines de capside et des ATPases utilisées pour l’encapsidation qui sont homologues aux protéines équivalentes présentes chez plusieurs familles de petits virus à ADN infectant les trois domaines du vivant (archées, bactéries, et eucaryotes). Ils ont donc été récemment classés avec tous ces virus par l’ICTV dans un même « domaine viral » (
realm
en Anglais), celui des
Varidnaviria
[
3
]. Si l’on considère que l’histoire des virions représente celle des virus, les
Varidnaviria
descendraient donc tous d’un ancêtre commun de type viral très ancien.
|
Deux des trois ARN polymérases des eucaryotes auraient une origine virale
En plus de ces gènes impliqués dans la formation des virions, tous les NCLDV partagent un petit groupe de protéines impliquées dans la réplication et la transcription de leurs génomes qui ont évolué en parallèle de façon verticale (c’est-à-dire héritées), ce qui nous a permis d’établir la phylogénie de la Figure 1 [
4
]. En particulier, la plupart des génomes des NCLDV codent leur propre ARN Pol, qui est homologue des ARN Pol cellulaires, ce qui permet de produire des arbres phylogénétiques comprenant à la fois ces virus, les archées, les bactéries, et les eucaryotes. Une telle analyse n’en est pas moins délicate : alors qu’une première étude des ARN Pol avait situé les NCLDV dans un groupe monophylétique séparé des domaines cellulaires, et conduisant à la notion de quatrième domaine du vivant [
7
], deux publications successives avaient critiqué ces résultats [
8
, [
9
]. En utilisant les mêmes données, mais en appliquant des modèles d’évolution plus sophistiqués, les auteurs de ces études obtenaient des phylogénies dans lesquelles les ARN Pol des NCLDV émergeaient des eucaryotes à différentes positions. Selon eux, les NCLDV ne formaient donc pas un quatrième domaine, mais auraient recruté les gènes codant leurs ARN Pol à partir de leurs hôtes plusieurs fois indépendamment. Toutes ces phylogénies étaient toutefois assez mal résolues, et systématiquement fondées sur l’analyse de l’une des deux grandes sous-unités de l’ARN Pol seulement.
Nous avions montré en 2017 qu’il était possible d’obtenir une phylogénie universelle du vivant très robuste en concaténant les deux sous-unités des ARN Pol des archées, des bactéries et des eucaryotes [
10
]. Nous avons donc décidé d’y ajouter les ARN Pol des NCLDV afin de compléter notre arbre phylogénétique. De plus, dans la plupart des études menées jusque-là, seule l’ARN Pol II des eucaryotes avait été utilisée comme marqueur évolutif de ce domaine, alors que toutes les cellules eucaryotes possèdent au moins trois ARN Pol : l’ARN Pol I, qui transcrit principalement les gènes codant les ARN ribosomiques, l’ARN Pol II, qui produit les ARN messagers (et est souvent considérée comme étant l’homologue des ARN Pol des archées et des bactéries), et l’ARN Pol III, qui transcrit principalement les gènes codant les ARN de transfert. L’inclusion de ces trois ARN Pol des eucaryotes et des séquences d’ARN Pol issues des NCLDV dans notre analyse phylogénétique nous a permis de proposer un scénario évolutif différent des précédents [
4
].
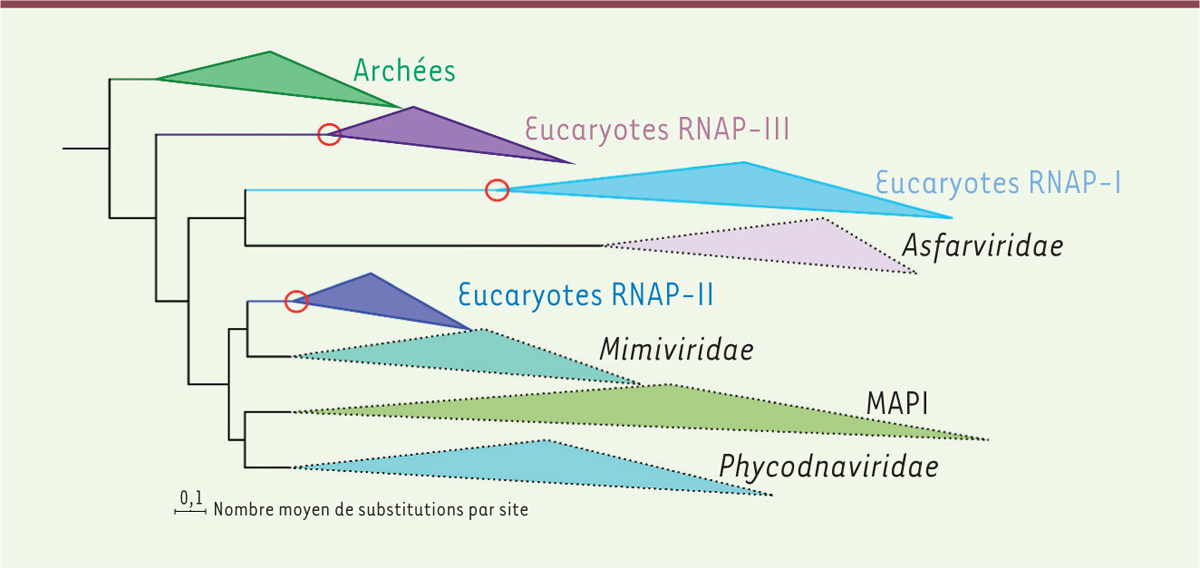
Étonnamment, l’ARN Pol des eucaryotes restée la plus proche de celles des archées n’était pas l’ARN Pol II, mais l’ARN Pol III. La présence des trois ARN Pol des eucaryotes dans notre jeu de données permettait de virtuellement situer trois fois dans notre arbre phylogénétique la position relative du dernier ancêtre commun des eucaryotes (
last eukaryotic common ancestor
, LECA)
(
Figure 2
)
. En effet, puisque ces trois ARN Pol sont présentes chez tous les eucaryotes actuels, elles devaient être déjà présentes chez LECA. Or, les ARN Pol des NCLDV émergeaient à des positions différentes entre les trois groupes d’ARN Pol des eucaryotes, ce qui indiquait que les NCLDV avaient commencé à diverger entre eux avant l’apparition de LECA. Nos résultats confirmaient donc l’ancienneté des NCLDV. Toutefois, les NCLDV ne formaient pas un groupe à part (« un quatrième domaine » du vivant), mais nos résultats montraient également que plusieurs familles et groupes monophylétiques conservaient les mêmes positions relatives que dans la topologie obtenue avec l’ensemble des marqueurs conservés chez les NCLDV : il apparaissait que c’étaient deux des ARN Pol des eucaryotes qui branchaient entre les NCLDV plutôt que l’inverse. Ainsi, les NCLDV auraient bien recruté initialement leur ARN Pol à partir d’un hôte cellulaire. Toutefois, contrairement à l’hypothèse qui proposait de multiples recrutements à partir de différents groupes d’eucaryotes modernes, ce recrutement n’aurait eu lieu qu’une seule fois, à partir d’un ancêtre très lointain de tous les eucaryotes actuels. La position de l’ARN Pol III, la plus proche des séquences d’archées, à la base des eucaryotes et des NCLDV suggérait que cette ARN Pol, non seulement était l’homologue des ARN Pol des archées et des bactéries, mais était également davantage similaire à celle recrutée initialement par l’ancêtre des NCLDV. Toutefois, outre la position des ARN Pol III, les ARN Pol I et II émergeaient au milieu des NCLDV. Nous avons conclu que ces dernières avaient dû être recrutées par les ancêtres des eucaryotes modernes à partir des NCLDV. La présence des trois ARN Pol des eucaryotes indiquant la position relative de LECA, il est possible de conclure que tous ces évènements ont eu lieu entre les ancêtres des NCLDV et les proto-eucaryotes, c’est-à-dire la lignée cellulaire précédant LECA, qui marque l’émergence des eucaryotes modernes.
 | Figure 2.
Représentation schématique de la phylogénie des archées, eucaryotes, et virus Nucleocytoviricota (NCLDV) fondée sur une analyse de la concaténation des deux grandes sous-unités des ARN polymérases des archées, des cellules eucaryotes (triangles en trait plein), et des NCLDV (triangles en pointillé).
L’arbre phylogénétique a été « enraciné » en utilisant les archées comme groupe extérieur. Les trois cercles rouges indiquent les trois positions de LECA, à la base des trois clades correspondant aux trois ARN polymérases (RNAP) des eucaryotes. MAPI : groupe de virus NCLDV comportant les
Marseilleviridae,
les
Ascoviridae
, les
Pitho-like viruses
, et les
Iridoviridae
.
|
Ces résultats remettent donc en question la vision traditionnelle sur l’origine des trois ARN Pol des eucaryotes. On pensait jusque-là qu’elles étaient apparues par duplication de gènes chez les proto-eucaryotes. Or notre analyse montre que ce n’est pas le cas, et que les NCLDV ont joué un rôle majeur dans la formation de la cellule eucaryote moderne. Il est impressionnant de penser que l’ARN Pol II, l’enzyme qui transcrit en ARN messagers tous nos gènes codant des protéines, est d’origine virale. |
L’hypothèse d’une origine virale du noyau des cellules eucaryotes à nouveau sur le devant de la scène
Chez les bactéries, on a récemment découvert des virus qui provoquent la formation d’un noyau au sein duquel le génome viral est répliqué [
11
]. Cette observation renforce l’hypothèse selon laquelle le noyau des cellules eucaryotes aurait évolué à partir des usines virales des NCLDV (pour revue, voir [
12
]). Ces usines virales, qui se développent dans le cytoplasme pour la production de virions, présentent en effet des similarités de taille et de structure avec le noyau des cellules eucaryotes. Dans le cadre de cette hypothèse, nos résultats suggèrent que plusieurs NCLDV ont pu, à différentes périodes de l’évolution, apporter des éléments qui ont progressivement conduit à l’émergence du noyau cellulaire tel que nous le connaissons. Plus généralement, on peut penser que la coévolution et la « course aux armements » entre NCLDV et proto-eucaryotes a dû jouer un rôle important dans la complexification croissante de ces derniers entre le dernier ancêtre commun aux archées et aux eucaryotes et LECA.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
La Scola
B
,
Audic
S
,
Robert
C
,
et al.
A giant virus in amoebae. Science.
2003;
;
299
:
:2033.
.
2.
Raoult
D
,
Forterre
P
.
Redefining viruses: lessons from mimivirus.
.
Nat Rev Microbiol.
2008;
;
6
:
:315.
–
319
.
3.
Koonin
EV
,
Dolja
VV
,
Krupovic
M
,
et al.
Global organization and proposed megataxonomy of the virus world.
.
Microbiol Mol Biol Rev.
2020;
;
84
:
:e00061.
–
19
.
4.
Guglielmini
J
,
Woo
AC
,
Krupovic
M
,
et al.
Diversification of giant and large eukaryotic dsDNA viruses predated the origin of modern eukaryotes.
.
Proc Natl Acad Sci USA.
2019;
;
116
:
:19585.
–
19592
.
5.
Gujgo
E
,
Kartenbeck
J
,
Lien
P
,
et al.
Amoebal pathogen mimivirus infects macrophages through phagocytosis.
.
PLoS Pathog.
2008
;
10.1371/journal.ppat.1000087
6.
Legendre
M
,
Fabre
E
,
Poirot
O
,
et al.
Diversity and evolution of the emerging Pandoraviridae family.
.
Nat Commun.
2018;
;
9
:
:2285.
.
7.
Colson
P
,
de Lamballerie
X
,
Fournous
G
,
et al.
Reclassification of giant viruses composing a fourth domain of life in the new order Megavirales.
.
Intervirology.
2012;
;
55
:
:321.
–
332
.
8.
Williams
TA
,
Embley
TM
,
Heinz
E
.
Informational gene phylogenies do not support a fourth domain of life for nucleocytoplasmic large DNA viruses.
.
PLoS One.
2011;
;
6
:
:e21080.
.
9.
Moreira
D
,
López-García
P
.
Evolution of viruses and cells: do we need a fourth domain of life to explain the origin of eukaryotes?
.
Philos Trans R Soc B Biol Sci.
2015;
;
370
:
:20140327.
.
10.
Da Cunha
V
,
Gaia
M
,
Gadelle
D
,
et al.
Lokiarchaea are close relatives of Euryarchaeota, not bridging the gap between prokaryotes and eukaryotes.
.
PLoS Genet.
2017;
;
13
:
:e1006810.
.
11.
Chaikeeratisak
V
,
Nguyen
K
,
Khanna
K
,
et al.
Assembly of a nucleus-like structure during viral replication in bacteria.
.
Science.
2017;
;
355
:
:194.
–
197
.
12.
Forterre
P
,
Gaïa
M
.
Giant viruses and the origin of modern eukaryotes.
.
Curr Opin Microbiol.
2016;
;
31
:
:44.
–
49
.
|