| |
| Med Sci (Paris). 36: 38–41. doi: 10.1051/medsci/2020182.
Profilage
in silico
des inhibiteurs de protéine kinases
Victor Reys1* and Gilles Labesse1** 1CNRS UMR 5048 - Inserm U1054 - Université de Montpellier
,
29 rue de Navacelles
,
34090Montpellier Cedex
,
France |
Des mutations ou réarrangements de l’ADN chromosomique sont fréquemment à l’origine de dysfonctionnements cellulaires divers pouvant mener à l’apparition d’un cancer. En effet, les mutations peuvent drastiquement modifier la quantité ou l’activité de certaines protéines liées à la division cellulaire, la différenciation cellulaire ou l’apoptose, et donc modifier le comportement des cellules au sein d’un organisme. Parmi les protéines ayant un fort impact sur la vie cellulaire, les protéine kinases en constituent une famille importante, puisqu’on en recense actuellement 535 chez l’homme [1]. Leur fonction est de moduler l’activité d’autres protéines, notamment d’autres protéine kinases, ou également des phosphatases, très majoritairement par phosphorylation de leurs cibles. Le dérèglement des cascades de régulation où sont impliquées les protéine kinases amplifie des signaux aberrants, pouvant stimuler la multiplication anarchique des cellules impactées, et donc l’apparition d’un cancer. Cette particularité fonctionnelle fait des protéine kinases des cibles thérapeutiques de choix.
L’histoire des traitements du cancer par inhibiteur de protéine kinases débute véritablement en 2001, avec la mise sur le marché de l’imatinib (Gleevec
®
) [2]. Depuis, une recherche très active a abouti à la mise sur le marché de plusieurs dizaines de médicaments ciblant cette famille de protéines [3]. Néanmoins, ces médicaments restent difficiles à concevoir et entraînent régulièrement des effets secondaires, principalement liés aux similarités entre leurs cibles thérapeutiques. À l’inverse, cette similarité favorise la poly-pharmacologie, destinée à cibler simultanément plusieurs protéine kinases impliquées dans la même cascade ou la même régulation cellulaire. Dans les deux cas, une connaissance fine de la spécificité des candidats-médicaments est requise.
|
Données expérimentales L’analyse des séquences d’ADN des gènes humains a permis en 2002 d’établir une première liste de 518 protéine kinases qui constitue ce que l’on appelle maintenant le kinome humain [4]. Depuis, cette liste a été augmentée et comprend désormais 535 protéines, qui ont été classées par groupes, familles et sous-familles. Elles possèdent un même repliement et pour la très grande majorité (sauf une cinquantaine de pseudo-kinases) un site de liaison de l’ATP (adénosine triphosphate).
Les deux dernières décennies ont permis de résoudre la structure d’environ 250 de ces protéines, principalement par la diffraction aux rayons X. L’analyse de ces structures a confirmé la similarité globale de ces enzymes, mais a aussi révélé certaines particularités (organisation des domaines associés de type SH2 ([
Src Homology 2
] ou SH3 [
Src Homology 3
], multimérisation…). Ces études ont permis la caractérisation fine de leurs interactions avec de petites molécules à visée thérapeutique en vue d’une optimisation rationnelle de ces interactions.
Modélisation du kinome
Grâce à ces données expérimentales, il est de nos jours possible de modéliser de manière fiable la très grande majorité des protéine kinases humaines. Une version 3D du kinome est ainsi mise à jour chaque année avec le pipeline @TOME [5], développé par le CBS (Centre de biologie structurale de Montpellier). Cette modélisation permet d’explorer les différentes conformations qu’est susceptible d’adopter chaque protéine kinase et d’identifier ensuite certains des ligands comme des candidats potentiels pour le développement de nouveaux inhibiteurs [6]. Cette première étape de modélisation peut permettre de mieux caractériser le site de liaison de l’ATP et d’identifier par exemple les pseudo-kinases ou les substitutions spécifiques d’acides aminés trouvées dans une protéine kinase particulière (ou dans une famille de protéine kinases). Ces particularités sont importantes pour envisager la conception d’inhibiteurs spécifiques. L’ensemble des modèles produits est d’ores et déjà accessible à la communauté scientifique (
http://atome.cbs.cnrs.fr/kinome
).
|
Les inhibiteurs de protéine kinases La majeure partie des inhibiteurs de kinases développés de nos jours sont des inhibiteurs compétitifs de l’ATP. Ils occupent la place de l’ATP au sein de la cible et empêche ainsi son hydrolyse et le transfert d’un phosphate sur une protéine substrat. L’utilisation d’inhibiteurs compétitifs dans le traitement du cancer a d’abord été controversée. Du fait du grand nombre de protéine kinases, de leur importante similarité de séquences (généralement 40-70 % et jusqu’à 90 %), de structure et d’activité, un manque important de spécificité était attendu.
Ce n’est que grâce à la résolution de structures cristallographiques (ex. : complexe Abl
1
-imatinib) que l’on a pu identifier certaines conformations particulières qui ne sont pas toutes prises par l’ensemble des protéine kinases humaines, et qui, de ce fait, permettent d’envisager la conception de médications hautement spécifiques. Ainsi, l’imatinib n’inhibe fortement que 4 protéine kinases, dont Abl, mais, par exemple, pas Src [une autre protéine kinase découverte dans un sarcome (src), agissant comme un proto-oncogène] qui lui est pourtant identique à 70 %. Cette information structurale aide la recherche et la conception de nouveaux inhibiteurs. Toutefois, il reste de nombreuses protéine kinases orphelines et de nombreux cancers sans traitement efficace.
Ainsi, de nombreux criblages fonctionnels à large échelle [3, 7] ont été effectués pour identifier de nouveaux composés chimiques inhibant spécifiquement une ou quelques protéine kinases d’intérêt. Ces données ont permis de mettre en évidence des inhibitions relativement spécifiques pour des châssis moléculaires particuliers. La dérivation de ces petites molécules pour optimiser leur spectre d’activité mais aussi leurs propriétés pharmaceutiques est, depuis, un domaine très actif. Toutefois, la synthèse organique de ces composés et le criblage systématique ne peuvent être envisagés pour tous les composés chimiques théoriquement accessibles. De même, toutes les protéines ne peuvent être produites et testées. Néanmoins, les informations fonctionnelles acquises pour des milliers de composés et des centaines de protéine kinases complètent déjà utilement les données structurales plus parcellaires. Leur combinaison ouvre des perspectives intéressantes pour une optimisation plus rationnelle des inhibiteurs de protéine kinases. |
Criblage intégré du kinome De nombreux outils informatiques sont actuellement développés pour permettre de tirer parti de ces informations structurales et fonctionnelles. Les uns proviennent du domaine de la bio-informatique dite structurale et les autres de la chémo-informatique. Jusqu’à récemment, ces différents outils étaient rarement combinés, mais les progrès de ces techniques informatiques et la grande masse de données militent pour leur plus grande intégration. La fouille intense des données (que l’on peut considérer comme une intelligence artificielle) ouvre aussi de nouvelles possibilités pour extraire des informations plus pertinentes. Parmi les stratégies modernes de développement d’un médicament, l’amarrage moléculaire et l’analyse de similarité des molécules chimiques sont des options de choix pour les premières étapes de définition d’inhibiteurs spécifiques. Ces deux méthodes virtuelles reposent sur des hypothèses différentes :
-
l’amarrage moléculaire recherche des molécules se positionnant adéquatement dans le site actif d’une cible,
-
la similarité moléculaire recherche des molécules de structures chimiques semblables aux composés connus.
Connaissant la structure tridimensionnelle d’une cible thérapeutique, il est possible de prédire les « poses »
2
probables de petites molécules (amarrage) et d’en déduire une affinité théorique (principalement l’enthalpie d’interaction). On peut ainsi classer les complexes
a priori
stables et trier ainsi les ligands potentiels. Cette information peut permettre de sélectionner les composés prometteurs et d’en guider l’optimisation. Toutefois, cette méthode souffre encore de nombreux faux négatifs et faux positifs. Ce criblage virtuel est souvent suivi d’une évaluation par un expert, étape longue et fastidieuse. Surtout, ces défauts limitent le criblage systématique de grandes chimiothèques sur l’ensemble des protéine kinases humaines. Cependant, les données récemment acquises (quelques milliers de structures de complexes) permettent la mise en place de pipelines d’analyse faisant appel à l’intelligence artificielle pour mieux détecter les complexes valides et prédire plus finement les affinités correspondantes. C’est dans cette perspective que nous développons une nouvelle version de notre serveur @TOME, combinant maintenant la modélisation comparative des protéine kinases et le criblage virtuel pour accélérer ces analyses.
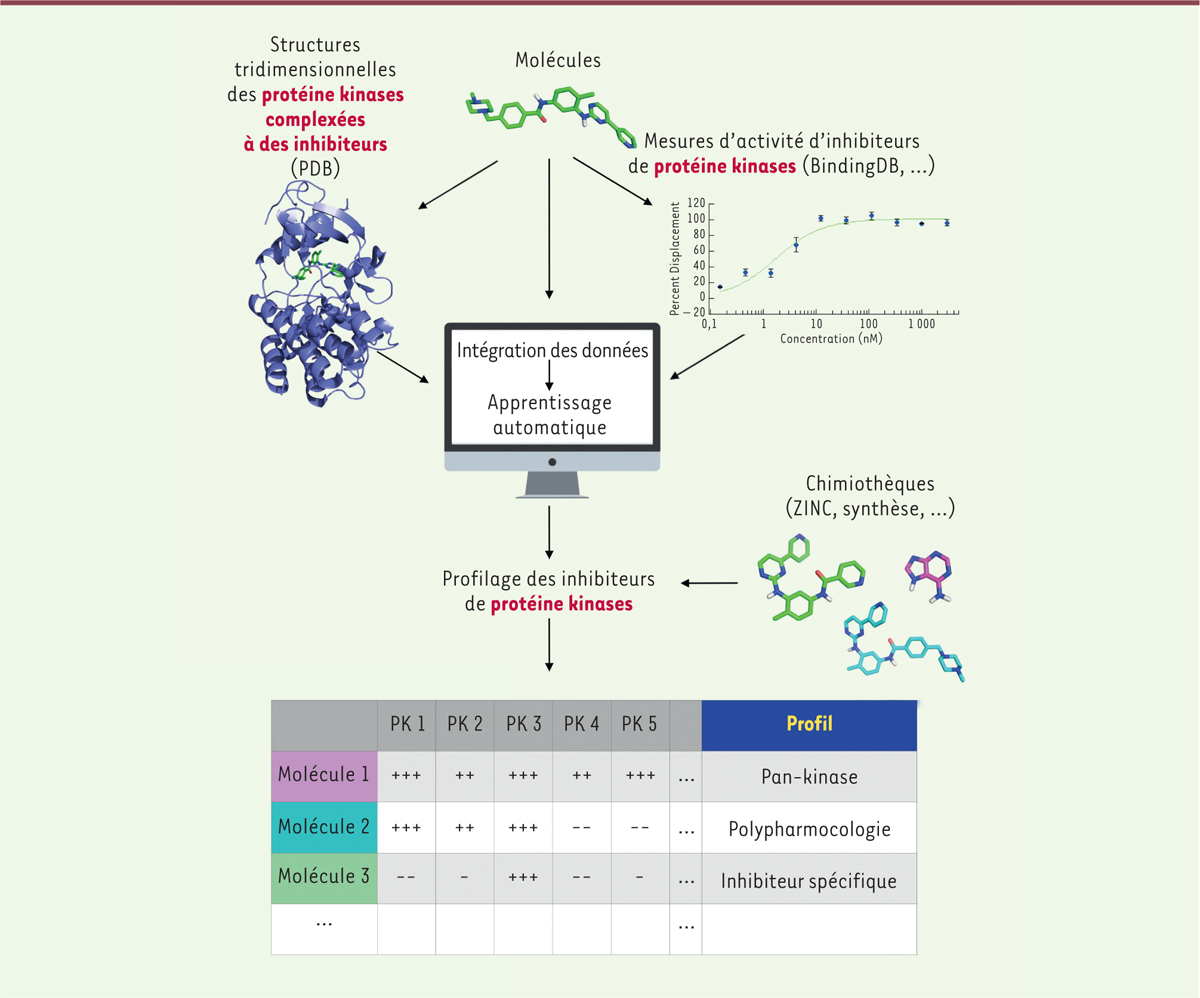
Les criblages expérimentaux (ci-dessus) sont reproduits
in silico
pour permettre l’apprentissage des paramètres en vue d’automatiser l’ensemble de ces étapes. De bonnes corrélations avec les données expérimentales suggèrent que l’on pourra prochainement estimer un profil d’activité pour de nombreux composés chimiques sur la majorité du kinome humain
(
Figure 1
)
.
 | Figure 1.
Le nombre des données structurales des protéine kinases et des données fonctionnelles obtenues par des criblages expérimentaux d’inhibiteurs croît depuis une vingtaine d’années.
Les avancées techniques informatiques permettent l’intégration de ces différentes données afin d’en extraire des paramètres prédictifs. L’immense masse de données disponibles, combinée avec l’intelligence artificielle, permet des prédictions d’affinités et de « poses » au sein d’une protéine de façon précise. La construction d’outils de profilage des inhibiteurs de protéine kinases pour le kinome humain est la prochaine étape qui augmentera considérablement la vitesse de découverte de solutions thérapeutiques pour le traitement des cancers.
|
La comparaison des structures chimiques des petites molécules permet aussi d’extrapoler les propriétés des unes aux autres de manière très complémentaire. Toutefois cette approche requiert une grande masse de données expérimentales portant sur l’affinité ou l’activité. Or celles-ci étaient rarement accessibles aux laboratoires publics. Cet état de fait évolue et l’analyse de larges campagnes de criblage expérimental ouvre la voie à l’utilisation de telles méthodes comparatives par la recherche académique. Il s’agit d’optimiser les outils de comparaison et notamment de choisir les caractéristiques chimiques à comparer. L’utilisation de méthodes consensus permet d’étendre la pertinence et la robustesse de ces comparaisons. Toutefois, cette recherche de similarité implique de se focaliser sur des séries chimiques déjà étudiées, contrairement à la méthode d’amarrage. À l’inverse, la similarité moléculaire permet des prédictions fonctionnelles plus précises. Par ailleurs, des molécules semblables adoptent régulièrement des modes d’amarrages similaires au sein de cibles similaires. Nous avons donc récemment développé un outil de comparaison des structures chimiques des petites molécules pour l’évaluation des prédictions d’amarrage. Nous allons prochainement tester systématiquement cet outil pour filtrer les criblages virtuels par amarrage en vue d’améliorer leurs prédictions. Des outils d’apprentissage machine permettront d’automatiser grandement ces tris. Enfin, l’ajout de données fonctionnelles devrait permettre d’optimiser simultanément la prédiction des affinités et des « poses » amarrées [8]. |
L’ensemble des données expérimentales (structure, activité) récemment acquises et l’avancée des techniques informatiques permet d’envisager le profilage des inhibiteurs de protéine kinases humaines dans un proche avenir. En effet, il devrait être prochainement possible de prédire le mode d’amarrage et l’affinité d’un composé pour une protéine kinase donnée et guider ainsi la conception de candidats médicaments de manière plus rationnelle. Nous espérons que ces approches intégrées permettront d’aider la découverte de nouvelles solutions thérapeutiques anti-cancéreuses. |
Cette recherche est effectuée dans le cadre d’un contrat doctoral avec l’Université de Montpellier et financée par la Ligue Contre le Cancer (thèse de doctorat de Victor Reys).
|
Nous remercions la Ligue contre le Cancer pour sa proposition de soumission à médecine/sciences à l’occasion de la Journée « Jeunes et Chercheurs » du 19 octobre 2020.
|
Footnotes |
1.
Wilson
LJ
,
Linley
A
,
Hammond
DE
,
et al.
New perspectives, opportunities, and challenges in exploring the human protein Kinome.
.
Cancer Res.
2018;
;
78
:
:15.
–
29
.
2.
Lyseng-Williamson
K
,
Jarvis
B
.
Imatinib. Drugs.
2001;
;
61
:
:1765.
–
1776
.
3.
Klaeger
S
,
Heinzlmeir
S
,
Wilhelm
M
,
et al.
The target landscape of clinical kinase drugs.
.
Science.
2017;
;
358
:
:eaan4368.
.
4.
Manning
G
,
Whyte
DB
,
Martinez
R
,
et al.
The protein kinase complement of the human genome.
.
Science.
2002;
;
298
:
:1912.
–
1934
.
5.
Pons
JL
,
Labesse
G
.
@Tome-2: a new pipeline for comparative modeling of protein-ligand complexes.
.
Nucleic Acids Res.
2009;
;
37
:
:W485.
–
W491
.
6.
Martin
L
,
Catherinot
V
,
Labesse
G
.
kinDock: a tool for comparative docking of protein kinase ligands.
.
Nucleic Acids Res.
2006;
;
34
:
:W325.
–
W329
.
7.
Metz
JT
,
Johnson
EF
,
Soni
NB
,
et al.
Navigating the kinome.
.
Nat Chem Biol.
2011;
;
7
:
:200.
–
202
.
8.
Schneider
M
,
Pons
JL
,
Bourguet
W
,
Labesse
G
.
Towards accurate high-throughput ligand affinity prediction by exploiting structural ensembles, docking metrics and ligand similarity.
.
Bioinformatics.
2020;
;
36
:
:160.
–
8
.
|