| |
| Med Sci (Paris). 36(12): 1218–1220. doi: 10.1051/medsci/2020234.
La bactérie
Listeria
module la signalisation de l’interféron en sécrétant une protéine qui se lie à l’ARN
Alessandro Pagliuso1* and Pascale Cossart2** 1Université Paris-Saclay, INRAE, AgroParisTech, Institut Micalis, Équipe d’épigénétique et de microbiologie cellulaire
,
78350Jouy-en-Josas
,
France
.
2Département de biologie cellulaire et infection, Unité des interactions bactéries-cellules, Institut Pasteur
,
25-28 rue du Docteur Roux
,
75015Paris
,
France
.
MeSH keywords: Protéines bactériennes, Protéine-58 à domaine DEAD, Interactions hôte-pathogène, Humains, Interféron de type I, Listeria monocytogenes, ARN bactérien, Protéines de liaison à l'ARN, Récepteurs immunologiques, Transduction du signal, Virulence, composition chimique, métabolisme, pathogénicité |
Au cours de leur co-évolution avec leur hôte, les bactéries pathogènes ont développé une variété impressionnante de stratégies pour prendre le contrôle des voies de signalisation et éviter les mécanismes de défense des cellules cibles. Afin d’établir une infection efficace, les bactéries intracellulaires pathogènes utilisent des facteurs de virulence – également appelés protéines effectrices – qui sont sécrétés dans le cytoplasme des cellules de l’hôte. Ces effecteurs bactériens se localisent dans des compartiments cellulaires spécifiques, où ils exercent leur fonction principalement en se liant à des protéines de la cellule hôte, qu’ils modifient. Au cours de la dernière décennie, plusieurs mécanismes d’action ont été décrits pour les effecteurs bactériens sécrétés, allant de la suppression de la réponse immunitaire innée, l’évasion de l’autophagie, l’inhibition de l’apoptose, à la subversion du trafic membranaire et la manipulation de la dynamique du cytosquelette. La plupart des effecteurs bactériens décrits jusqu’à présent ont été décrits comme interagissant avec des constituants protéiques de l’hôte. Seuls quelques effecteurs interagissaient avec les acides nucléiques, et plus particulièrement avec l’ADN [
1
]. Des effecteurs bactériens sécrétés capables de se lier à l’ARN n’ont pas encore été décrits [
2
].
|
Zea, le premier effecteur bactérien sécrété ciblant l’ARN
Listeria monocytogenes
est l’agent causal de la listériose, une maladie d’origine alimentaire. Cette bactérie est non seulement devenue un modèle pour comprendre les interactions hôte-pathogène, mais c’est aussi un formidable outil pour disséquer des mécanismes fondamentaux de biologie cellulaire et d’immunologie [
3
]. Le pouvoir pathogène de
Listeria
repose sur la production d’un arsenal de facteurs de virulence qui permettent à cette bactérie de pénétrer à l’intérieur d’une grande variété de types de cellules et de s’y répliquer à l’abri du système immunitaire. Au cours des dernières décennies, en utilisant des approches de génétique classique ou de post-génomique comparative, notre laboratoire a contribué à la caractérisation de nombreux facteurs de virulence de ce pathogène. Il s’agit de protéines de surface ou de protéines sécrétées [
4
]. La protéine caractérisée le plus récemment est la protéine Zea, un petit polypeptide basique (point isoélectrique [pI] = 8,4) de 177 acides aminés, ne présentant pas de similarité avec des protéines de fonction connue [
5
]
(
Figure 1
)
. Nous nous sommes intéressés à cette protéine car elle était présente dans l’espèce pathogène
L. monocytogenes
, mais absente de l’espèce non pathogène
Listeria innocua
, ce qui suggérait qu’elle contribuait à la virulence de l’espèce pathogène. De plus, elle était prédite comme sécrétée par la bactérie en raison de la présence d’un peptide signal à son extrémité N-terminale, et pouvait donc interagir avec des cibles des cellules eucaryotes. Afin de déterminer le rôle de Zea dans le processus infectieux, nous avons produit une souche mutante de
L. monocytogenes
dépourvue de cette protéine. Nous avons constaté que les bactéries déficientes en Zea colonisaient mieux le foie et la rate des souris inoculées, ce qui indiquait que Zea est un facteur qui atténue la virulence de
Listeria
. Pour comprendre la fonction de cette protéine, nous avons analysé sa structure cristallographique aux rayons X, qui avait été déposée avant notre étude dans la banque de données des protéines
(
Figure 1
)
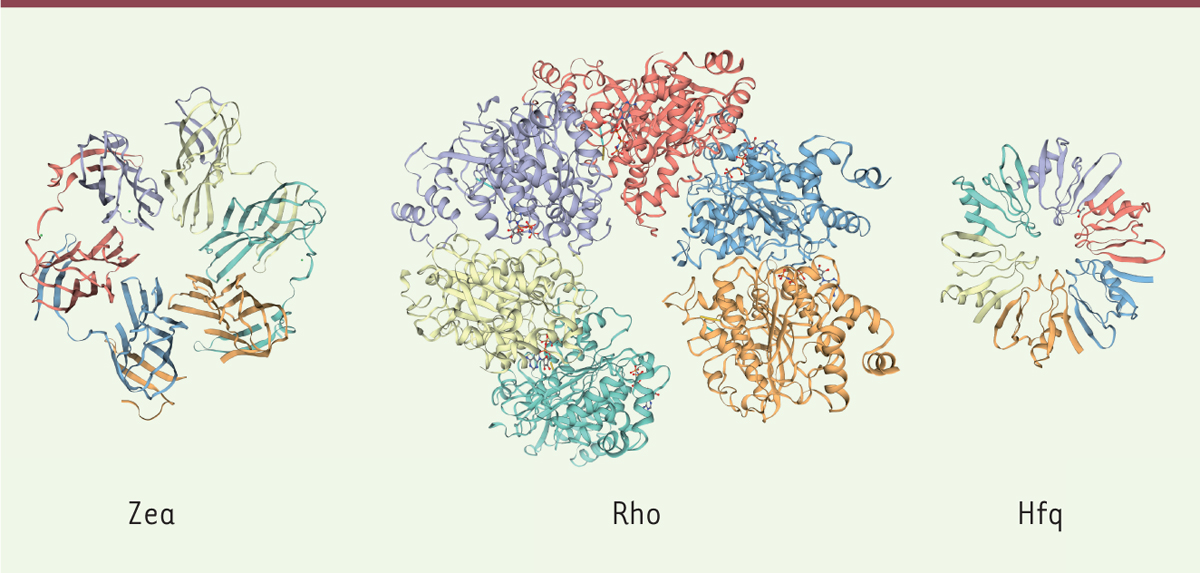
. Bien que Zea n’ait pas de domaine structural connu, il est apparu qu’elle se repliait en un homo-hexamère d’architecture similaire à celle des protéines du cytosol bactérien connues pour se lier à l’ARN, telles que Rho et Hfq
(
Figure 1
)
. Il était alors tentant de supposer que Zea était également une protéine de liaison à l’ARN, la première décrite comme sécrétée par une bactérie. Nous avons d’abord vérifié que Zea était secrétée, et avons montré que Zea était en effet présente dans le milieu de croissance des cultures de
Listeria
en utilisant des anticorps spécifiques dirigés contre la protéine. Nous avons ensuite testé si Zea pouvait se lier à de l’ARN bactérien en purifiant la protéine présente dans le milieu extracellulaire par immunoprécipitation avec des anticorps anti-Zea, puis en séquençant l’ARN co-purifié. Nous avons ainsi pu identifier plusieurs transcrits de gènes de
Listeria
associés à Zea. Un test de liaison
in vitro
a confirmé que Zea pouvait se lier directement à l’ARN de
Listeria
. De même, après surexpression de Zea, nous avons mis en évidence une accumulation de ses ARN cibles dans le milieu de croissance, qui est probablement la conséquence de leur stabilisation suite à leur liaison à la protéine. Il est intéressant de noter que l’analyse de la séquence de la protéine Zea n’a pas révélé la présence de motifs caractéristiques de liaison à l’ARN, ce qui suggérait que Zea se lie à l’ARN de manière « non canonique », comme cela a été montré récemment pour plusieurs protéines de mammifères [
6
]. En effet, le développement de nouvelles techniques fondées sur la liaison covalente des complexes protéines-ARN sous l’action des rayons ultra-violets (UV), suivie de la capture par affinité de l’ARN poly(A)
+
et de l’identification par spectrométrie de masse des protéines co-éluées, a révélé un grand nombre de protéines de mammifères ayant des capacités de liaison à l’ARN insoupçonnées jusqu’alors [
6
]. Grâce à ces approches globales, le recensement des protéines de liaison à l’ARN chez les mammifères s’est accéléré ces dernières années, et des protéines avec des domaines de liaison à l’ARN « non canoniques » ont été identifiées. Malheureusement, cette méthode ne peut pas être appliquée aux procaryotes car l’ARN bactérien ne possède pas la queue poly(A) qui est utilisée pour purifier les complexes ARN-protéines. Cette limitation a ralenti l’identification de protéines bactériennes de liaison à l’ARN. Cependant, une telle approche pourrait théoriquement être appliquée aux cellules infectées par les bactéries afin d’identifier des protéines de liaison à l’ARN sécrétées par les bactéries et ciblant l’ARN de l’hôte. Aucun effecteur bactérien ciblant l’ARN de la cellule infectée n’ayant encore été identifié, elle pourrait mettre en évidence une stratégie, non encore décrite, utilisée par une bactérie pathogène pour « dominer » son hôte par la manipulation du métabolisme de l’ARN de la cellule hôte.
 | Figure 1.
Représentation de la structure tridimensionnelle de la protéine bactérienne hexamérique sécrétée Zea et de deux autres protéines bactériennes hexamériques non sécrétées se liant à l’ARN, Rho et Hfq.
Chaque couleur représente un monomère différent dans la structure polymérique.
|
|
Un nouveau mécanisme de contrôle de la production d’interféron lors d’une infection bactérienne
Deux études avaient précédemment montré que, lors d’une infection par
Listeria
, de l’ARN bactérien est sécrété dans le cytoplasme des cellules de l’hôte, où il est alors détecté par le facteur RIG-I (
retinoic acid-inducible gene I protein
), une protéine de défense des cellules de mammifères qui « surveille » l’invasion du cytoplasme en détectant l’ARN viral ou bactérien [
7
,
8
]. La liaison de l’ARN de
Listeria
à RIG-I déclenche une cascade de signalisation qui conduit à la production d’interféron (IFN) de type I, une cytokine antivirale majeure, mais qui est bénéfique à la réplication de
Listeria
au cours de certaines phases de l’infection [
9
]. Comme la protéine Zea est sécrétée et se lie à l’ARN de
Listeria
, nous nous sommes demandés si elle était impliquée dans le contrôle de la production d’interféron dépendant de RIG-I. Nous avons infecté des cellules, soit avec une souche de
Listeria
de type sauvage, soit avec une souche qui surexprime Zea. Étonnamment, les bactéries surexprimant Zea ont été capables de déclencher une réponse IFN plus forte que les bactéries de type sauvage, ce qui indique que Zea peut stimuler la production d’IFN. De plus, en éliminant RIG-I des cellules, nous avons montré que la stimulation de l’expression de l’IFN induite par Zea était dépendante de la présence de cette protéine. Comment Zea active-t-elle la signalisation impliquant RIG-I ? Par une expérience de liaison
in vitro
, nous avons découvert que Zea était capable de se lier à RIG-I. Toutefois, la liaison de Zea à RIG-I n’était pas suffisante pour induire la production d’IFN, ce qui suggérait que la présence d’ARN de
Listeria
était également nécessaire pour déclencher cette réponse de la cellule infectée. En accord avec cette hypothèse, nous avons montré que, lorsque les ARN liés à Zea étaient produits
in vitro
puis introduits à l’intérieur des cellules par transfection, ils étaient capables de déclencher la production d’IFN. Enfin, en extrayant RIG-I et ses partenaires ARN des cellules infectées par
Listeria
, nous avons découvert que Zea et RIG-I se liaient à un sous-ensemble similaire d’ARN de
Listeria
. Collectivement, ces résultats suggèrent que pendant l’infection par
Listeria
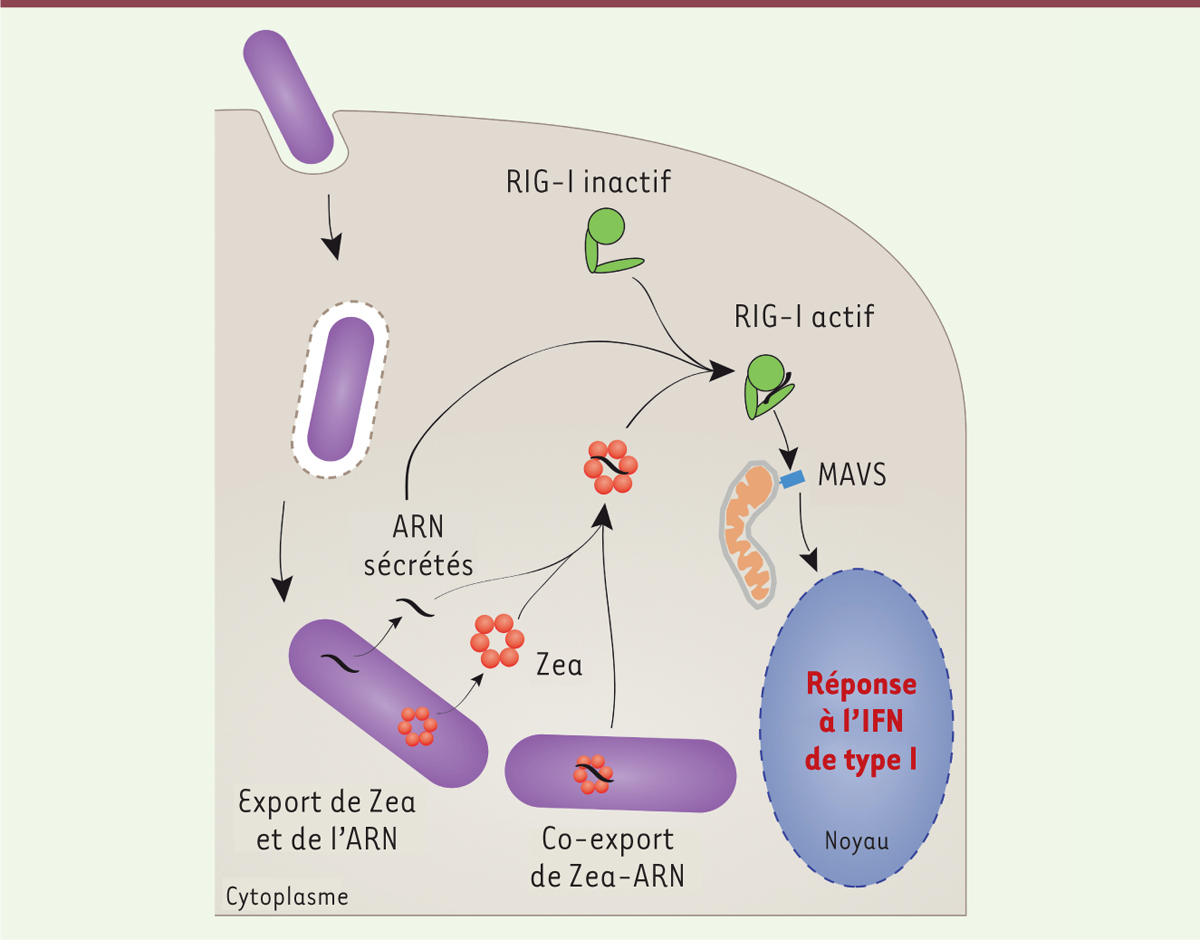
, les complexes Zea-ARN ont accès au cytosol de l’hôte et induisent une production d’IFN en se liant et en stimulant l’activité de RIG-I
(
Figure 2
)
.
 | Figure 2.
Modèle actuel rendant compte de la stimulation de la réponse à l’interféron de type I induite par la protéine Zea.
On ignore si Zea et ses ARN cibles sont exportés conjointement par
Listeria
ou s’ils ne se lient qu’après avoir été exportés individuellement par la bactérie. RIG-1 :
retinoic acid-inducible gene I protein
; MAVS : protéine de signalisation antivirale mitochondriale ; IFN : interféron.
|
|
Nos travaux ont permis d’identifier la première protéine bactérienne sécrétée se liant à l’ARN, Zea, qui contrôle la virulence de
Listeria
. Ils ont ainsi révélé un nouveau type d’acteurs dans le dialogue hôte-pathogène. Zea se lie à l’ARN extracellulaire de
Listeria
, et nous avons montré que les complexes Zea-ARN sont impliqués dans la modulation de la production d’IFN dépendante de RIG-I pendant l’infection par cette bactérie. Nous avons également contribué à l’identification des molécules d’ARN de
Listeria
qui sont importantes pour activer la signalisation impliquant RIG-I, donc la réponse de l’hôte. Il est à noter que nous avons trouvé des protéines orthologues de Zea dans d’autres espèces bactériennes appartenant aux genres
Bacillus, Clostridium et Acinetobacteri
a. Certaines de ces bactéries n’ont aucune virulence, et se trouvent principalement dans l’environnement ainsi que dans le microbiote intestinal humain. Le concept d’ARN social, c’est-à-dire d’ARN exporté par une bactérie pour communiquer ou influencer le comportement d’un organisme cible, a été proposé en 2017 [
10
], mais il existait peu de preuves de son existence réelle avant l’identification de Zea. Si les orthologues de Zea s’avèrent être également des protéines bactériennes sécrétées se liant à l’ARN, le concept d’ARN social prendra tout son sens en pointant une nouvelle facette de la biologie de la communication bactéries-hôtes fondée sur la sécrétion bactérienne d’ARN et de ses ligands protéiques. Une fois de plus, l’étude de
Listeria
aura permis d’ouvrir un nouveau champ d’étude et de commencer à l’explorer.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Bierne
H
,
Pourpre
R
.
Bacterial factors targeting the nucleus: the growing family of nucleomodulins.
.
Toxins (Basel).
2020;
;
12
.
2.
Tawk
C
,
Sharan
M
,
Eulalio
A
,
Vogel
J
.
A systematic analysis of the RNA-targeting potential of secreted bacterial effector proteins.
.
Sci Rep.
2017;
;
7
:
:9328.
.
3.
Radoshevich
L
,
Cossart
P
.
Listeria monocytogenes: towards a complete picture of its physiology and pathogenesis.
.
Nat Rev Microbiol.
2018;
;
16
:
:32.
–
46
.
4.
Glaser
P
,
Frangeul
L
,
Buchrieser
C
,
et al.
Comparative genomics of Listeria species.
.
Science.
2001;
;
294
:
:849.
–
852
.
5.
Pagliuso
A
,
Tham
TN
,
Allemand
E
,
et al.
An RNA-binding protein secreted by a bacterial pathogen modulates RIG-I signaling.
.
Cell Host Microbe.
2019;
;
26
:
(823–35)
:e11.
.
6.
Hentze
MW
,
Castello
A
,
Schwarzl
T
,
Preiss
T
.
A brave new world of RNA-binding proteins.
.
Nat Rev Mol Cell Biol.
2018;
;
19
:
:327.
–
341
.
7.
Frantz
R
,
Teubner
L
,
Schultze
T
,
et al.
The secRNome of Listeria monocytogenes harbors small noncoding RNAs that are potent inducers of beta interferon.
.
mBio.
2019;
;
10
.
8.
Abdullah
Z
,
Schlee
M
,
Roth
S
,
et al.
RIG-I detects infection with live Listeria by sensing secreted bacterial nucleic acids.
.
EMBO J.
2012;
;
31
:
:4153.
–
4164
.
9.
Witte
CE
,
Archer
KA
,
Rae
CS
,
et al.
Innate immune pathways triggered by Listeria monocytogenes and their role in the induction of cell-mediated immunity.
.
Adv Immunol.
2012;
;
113
:
:135.
–
156
.
10.
Braukmann
F
,
Jordan
D
,
Miska
E
.
Artificial and natural RNA interactions between bacteria and C. elegans.
.
RNA Biol.
2017;
;
14
:
:415.
–
420
.
|