| |
| Med Sci (Paris). 36(11): 976–979. doi: 10.1051/medsci/2020152.Identification des cellules dendritiques inflammatoires par analyse multi-omique Charles-Antoine Dutertre1,2,3* and Florent Ginhoux1,3,4** 1Singapore immunology network, A*STAR, 8A Biomedical grove, Immunos building
,
Singapore138648
,
Singapour
.
2Program in emerging infectious disease, Duke-NUS medical school
,
8 College road
,
Singapore169857
,
Singapour
.
3Translational immunology institute, Singhealth/Duke-NUS academic medical centre, the Academia
,
20 College road, Discovery tower level 8
,
Singapore169856
,
Singapour
.
4Shanghai institute of immunology, Department of immunology and microbiology
,
Shanghai
,
Chine
.
MeSH keywords: Algorithmes, Séparation cellulaire, Cellules dendritiques, Analyse de profil d'expression de gènes, Génomique, Humains, Inflammation, Antigènes CD14, Lupus érythémateux disséminé, Protéomique, méthodes, métabolisme, anatomopathologie, génétique, immunologie |
Les phagocytes mononucléés circulants regroupent des populations de cellules dendritiques (CD) et de monocytes ayant des fonctions et des phénotypes chevauchants, rendant leur discrimination encore imprécise. Une meilleure définition phénotypique de ces cellules est nécessaire pour étudier précisément leur fonction en situation normale ou dans différents contextes pathologiques tels que les maladies inflammatoires ou auto-immunes [
1
,
2
]. Les CD sont divisées en trois principales sous-populations, les CD conventionnelles 1 et 2 (cDC1 et cDC2), spécialisées dans la présentation d’antigènes aux lymphocytes T respectivement CD8
+
et CD4
+
, et les CD plasmacytoïdes (pDC) spécialisées dans la sécrétion d’interféron de type 1 (IFN-I) [
3
]. De récentes études ont montré que les pDC murines sont dérivées de progéniteurs lymphoïdes, remettant en cause leur appartenance à la famille des CD [
4
]. Enfin, nous avons récemment décrit une population de cellules progénitrices circulantes ayant la capacité de se différencier en cDC1 et en cDC2 [
5
].
Alors que les cDC1 expriment spécifiquement certains marqueurs (CADM1, XCR1, CLEC9A) permettant leur délimitation précise, les cDC2 sont très hétérogènes et ont un phénotype chevauchant souvent celui des monocytes, en particulier dans certains tissus en situation inflammatoire, où des cellules dérivées de monocytes peuvent adopter un phénotype très proche de celui des cDC2 [
6
]. Les monocytes sont également divisés en sous-populations définies en fonction de leur expression du récepteur pour la région Fc des immunoglobulines G (RFcγIIIA, CD16) et de CD14, un corécepteur du lipopolysaccharide bactérien (LPS). Ainsi, les monocytes classiques (cMo, CD14
++
CD16
-
), intermédiaires (iMo, CD14
++
CD16
+
) et non-classiques (ncMo, CD14
+
CD16
++
) constituent les trois sous-populations de monocytes circulants [
7
]. Parmi les phagocytes mononucléés, les monocytes ont été longtemps considérés comme étant les seuls à exprimer ces deux protéines membranaires. Cependant, cette restriction est remise en question car les cMo et les cDC2 (qui contiennent deux sous-populations, les DC2 et les DC3) [
8
] forment un continuum phénotypique avec des cellules au phénotype intermédiaire, exprimant à la fois des marqueurs de CD et de monocytes.
|
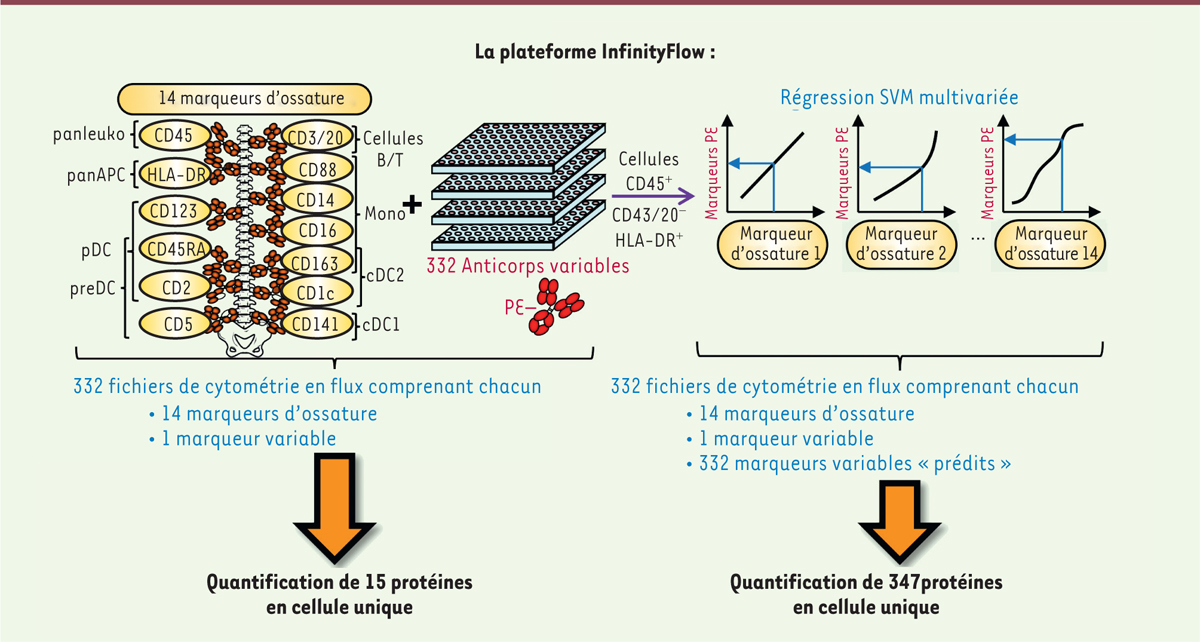
Les analyses protéomiques et transcriptomiques en cellule unique délimitent les populations de phagocytes mononucléés circulants, et mettent en évidence une population de cDC2 CD14+
Afin de mieux caractériser et délimiter les CD et les monocytes circulants, nous avons réalisé une analyse de l’expression de 332 protéines membranaires par cytométrie en flux (FACS ; 332 fichiers). Aidé par de l’apprentissage automatique (machine learning), la plateforme InfinityFlow permet de prédire l’expression de ces 332 protéines et de les combiner pour obtenir une matrice de données contenant 347 dimensions d’expression protéique
(
Figure 1
)
. Une réduction de dimensionnalité (DimRed) des 332 protéines prédites utilisant l’algorithme tSNE a permis de classer les phagocytes mononucléés circulants, et de définir de nouveaux marqueurs spécifiquement exprimés par les différentes sous-populations
(
Figure 2A
)
. Une analyse comparative des cDC2 et des monocytes classiques (cMo) a permis de définir des marqueurs améliorant leur discrimination phénotypique. La combinaison de ces nouveaux marqueurs discriminants par cytométrie conventionnelle permet une délimitation précise des populations de cDC2 et de cMo, et indique que les cellules intermédiaires (CD1c
+
CD14
+
) sont phénotypiquement apparentées aux cDC2.
 | Figure 1.
La plateforme InfinityFlow.
Les cellules du sang périphérique d’un donneur unique ont été analysées par cytométrie en flux. Toutes les cellules ont été marquées avec des anticorps détectant 14 protéines dites « d’ossature ». Les cellules ont ensuite été divisées pour réaliser 332 marquages utilisant chacun un anticorps différent conjugue à un fluorochrome appelé « PE ». Après extraction des cellules CD45
+
CD3/20
-
HLA-DR
+
(phagocytes mononucléés), une analyse en régression multivariée (SVM) a été réalisée afin de prédire l’expression des 332 marqueurs « variables ». Cette approche permet ainsi d’obtenir des fichiers d’expression de 347 protéines en cellule unique (figure adaptée de [
2
]).
|
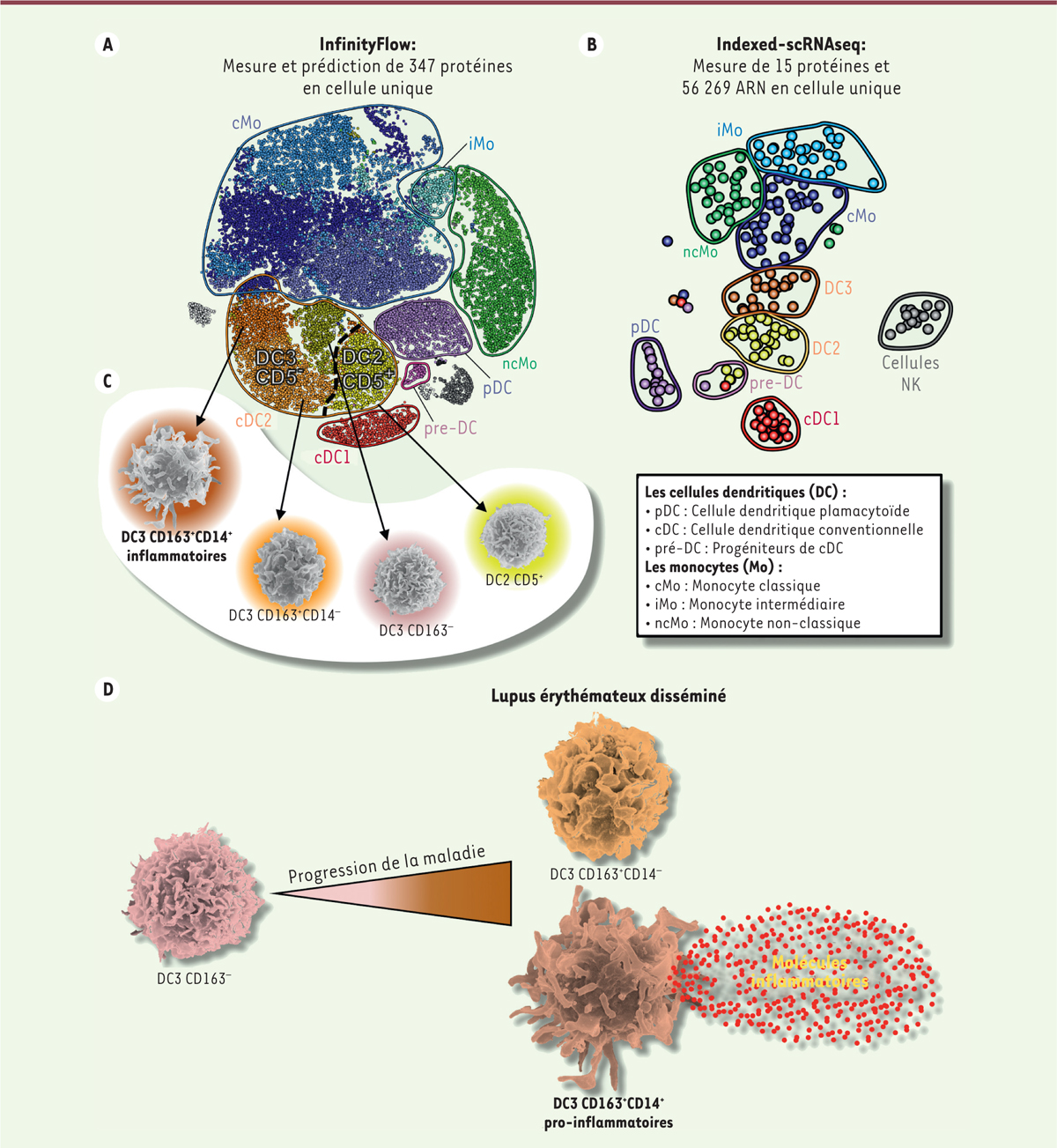
 | Figure 2.
Analyse multi-omique des phagocytes mononucléés circulants humains. A-B.
Analyse de l’expression de
(A)
protéines utilisant le plateforme InfinityFlow ou de
(B)
15 protéines combinées à l’intégralité des ARN messagers (transcriptome) en cellule unique des phagocytes mononucléés CD45
+
CD3/20
-
HLA-DR
+
. Visualisation des cellules uniques par réduction de dimensionnalité et regroupement non-supervisé, utilisant respectivement les algorithmes tSNE et Phenograph.
C.
Images de microscopie électronique des sous-population de cDC2 (DC2 et DC3) et de monocytes classiques.
D.
Schéma montrant chez des patients atteints de lupus érythémateux disséminé la conversion des DC3 CD163
-
en DC3 CD163
+
, parmi lesquelles, les DC3 CD163
+
CD14
+
pourraient participer à la pathologie, en produisant d’importantes quantités de molécules inflammatoires (figure adaptée de [
2
]).
|
Les phagocytes mononucléés circulants ont ensuite été caractérisés par une analyse transcriptomique en cellule unique, combinée avec une indexation-FACS (mesure de l’expression de protéines membranaires pour chaque cellule triée ;
Figure 2B
). L’analyse tSNE de leur profil transcriptomique a permis de définir l’ensemble des populations de CD, de monocytes, et a mis en évidence une population contaminante de cellules NK. L’utilisation de l’indexation de marqueurs protéiques a permis de définir les cDC2 CD1c
+
, les monocytes CD14
+
, ainsi que la population intermédiaire CD1c
+
CD14
+
. Cette population intermédiaire est entièrement contenue dans le groupe de cDC2 défini par l’analyse des transcrits, confirmant, au niveau transcriptomique, que ces cellules sont apparentées aux cDC2.
Les cDC2 incluent donc une population de cellules exprimant CD14, un marqueur classiquement associé aux monocytes, et l’analyse de leur hétérogénéité a permis de définir deux sous-populations principales, les DC2 CD5
+
et les DC3 CD5
-
, qui sont très hétérogènes et comprennent les DC3 CD14
+
(
Figure 2C
)
. L’analyse par microscopie électronique des sous-populations de cDC2 (DC2 et DC3) indique que les DC3 CD14
+
sont les cellules les plus grandes et les plus granuleuses, ce qui suggère qu’elles sont dans un état plus activé
(
Figure 2C
)
.
|
Les DC3 CD14+ sont des cellules dendritiques inflammatoires impliquées dans la pathogenèse du lupus érythémateux disséminé
Une étude avait mis en évidence une population de cellules CD1c
+
CD14
+
inflammatoires dans les liquides d’ascite de patientes atteintes d’un cancer du sein métastatique, mais ces cellules avaient alors été décrites comme étant dérivées de monocytes [
9
]. L’analyse de la signature transcriptomique de ces cellules inflammatoires d’ascite carcinomateuse indique que les DC3 CD14
+
sont les seuls phagocytes mononucléés du sang (circulantes) à exprimer ces gènes inflammatoires, permettant donc d’appeler ces cellules « DC3 CD14
+
inflammatoires circulantes ». L’analyse transcriptomique des sous-populations de DC indique que les DC3 inflammatoires sont enrichies en transcrits impliqués dans des modules de maturation, mais aussi en transcrits impliqués dans le lupus érythémateux disséminé (
systemic lupus erythematosus
, SLE ).
L’analyse de groupes de patients présentant un SLE ou une sclérodermie systémique (
systemic sclerosis
, SSc), une autre maladie auto-immune, a permis d’observer une augmentation significative de la proportion de DC3, spécifiquement dans le sang de patients SLE, par rapport aux leucocytes mononucléés CD45
+
, mais aussi par rapport aux cDC2 totales (DC2 & DC3)
(
Figure 2D
)
. De plus, leur fréquence est corrélée significativement avec le score de gravité de la maladie (
Systemic lupus erythematosus disease activity index
, SLEDAI). L’analyse transcriptomique des DC2 et DC3 de donneurs sains et de patients SLE a permis de montrer une réduction de l’état d’activation des DC2, accompagnée d’une forte activation des DC3 chez les patients, indiquant l’importance de l’analyse séparée des DC2 et DC3, qui jusque alors étaient regroupées en une seule population de cDC2.
Afin d’évaluer l’implication fonctionnelle des DC3, en particulier des DC3 CD14
+
inflammatoires, dans la pathogenèse du SLE, nous avons cultivé les sous-populations de DC2 et DC3 de donneurs sains en présence de sérum de donneurs sains ou de patients SLE présentant une maladie active ou inactive
(
Figure 2D
)
. 55 médiateurs solubles ont ensuite été mesurés dans les surnageants de culture, et les données obtenues analysées par l’algorithme UMAP (
Uniform Manifold Approximation and Projection
) [
10
]. Dans cette analyse UMAP, où chaque point correspond à un surnageant de culture, les surnageants des DC3 CD14
+
inflammatoires sont regroupés dans une même zone de l’espace indiquant un programme sécrétoire spécifique de ces cellules. Dans l’espace UMAP, les surnageants issus de DC3 CD14
+
inflammatoires cultivées avec du sérum de patients SLE actifs sont regroupés, indiquant que ces cellules voient leur programme sécrétoire spécifiquement activé dans un environnement de patients atteints de SLE actif. Lorsqu’elles sont cultivées en présence de sérum de patients atteints de SLE actif, les DC3 CD14
+
inflammatoires sécrètent une grande variété de médiateurs solubles, dont bon nombre ont été préalablement décrits comme étant impliqués dans la pathogenèse du SLE.
Ces travaux montrent l’importance d’étudier les DC3 inflammatoires dans d’autres situations pathologiques, dans la circulation, mais aussi dans les tissus, et de mieux définir les mécanismes contrôlant l’activation et l’induction de ces cellules. De telles recherches devraient permettre de développer de nouvelles stratégies thérapeutiques, ciblant les DC3 inflammatoires, dans des maladies inflammatoires et auto-immunes. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Tan-Garcia
A
,
Wai
LE
,
Zheng
D
,
et al.
Intrahepatic CD206
+
macrophages contribute to inflammation in advanced viral-related liver disease
.
.
J Hepatol.
2017;
;
63
:
:490.
–
500
.
2.
Dutertre
CA
,
Becht
E
,
Irac
SE
,
et al.
Single-cell analysis of human mononuclear phagocytes reveals subset-defining markers and identifies circulating inflammatory dendritic cells.
.
Immunity.
2019;
;
51
:
:573.
–
589
.
3.
Dutertre
CA
,
Wang
LF
,
Ginhoux
F
.
Aligning bona fide dendritic cell populations across species.
.
Cell Immunol.
2014
:
:3.
–
10
.
4.
Dress
RJ
,
Dutertre
CA
,
Giladi
A
,
et al.
Plasmacytoid dendritic cells develop from Ly6D
+
lymphoid progenitors distinct from the myeloid lineage
.
.
Nat Immunol.
2019;
;
20
:
:852.
–
864
.
5.
See
P
,
Dutertre
CA
,
Chen
J
,
et al.
Mapping the human DC lineage through the integration of high-dimensional techniques.
.
Science.
2017;
;
356
:
:6342.
.
6.
Guilliams
M
,
Dutertre
CA
,
Scott
CL
,
et al.
Unsupervised high-dimensional analysis aligns dendritic cells across tissues and species.
.
Immunity.
2016;
;
45
:
:669.
–
684
.
7.
Dutertre
CA
,
Amraoui
S
,
DeRosa
A
,
et al.
Pivotal role of M-DC8
+
monocytes from viremic HIV-infected patients in TNFα overproduction in response to microbial products
.
.
Blood.
2012;
;
120
:
:2259.
–
2268
.
8.
Villani
AC
,
Satija
R
,
Reynolds
G
,
et al.
Single-cell RNA-seq reveals new types of human blood dendritic cells, monocytes, and progenitors.
.
Science.
2017;
;
356
:
:6335.
.
9.
Segura
E
,
Touzot
M
,
Bohineust
A
,
et al.
Human inflammatory dendritic cells induce Th17 cell differentiation.
.
Immunity.
2013;
;
38
:
:336.
–
348
.
10.
Becht
E
,
McInnes
L
,
Healy
J
,
et al.
Dimensionality reduction for visualizing single-cell data using UMAP.
.
Nat Biotechnol.
2018;
;
37
:
:38.
–
44
.
|