| |
| Med Sci (Paris). 36(8-9): 813–816. doi: 10.1051/medsci/2020146.ADN et portait-robot : où en est-on ? Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université /EFS/CNRS ; CoReBio PACA, case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Adulte, Ethnies, Génétique légale, Études d'associations génétiques, Humains, Nouveau-né, Modes de transmission héréditaire, Mâle, Phénotype, Polymorphisme de nucléotide simple, Portraits comme sujet, Pigmentation de la peau, Jumeaux, génétique, méthodes, tendances |
Selon une impression assez répandue, et dont les grands médias se font parfois l’écho, « définir un portrait-robot à partir d’une trace ADN, c’est possible » comme l’affirmait Europe 1 en janvier 2015. Il est vrai que notre ADN définit assez précisément notre apparence physique, comme le montre la très forte ressemblance des jumeaux monozygotes même lorsqu’ils ont été séparés à la naissance (Figure 1). Mais sommes-nous capables de déduire ce phénotype à partir de l’analyse d’ADN ? Ce sujet avait fait l’objet d’une « Chronique génomique » il y a près de dix ans [1] (→) et il semble intéressant de voir ce qu’il en est aujourd’hui. On peut en effet penser qu’avec l’impressionnante avancée des techniques de génomique, le séquençage de l’ADN de centaines de milliers de personnes et la constitution de grandes bases informatiques réunissant données génomiques, médicales et morphologiques, la prédiction du phénotype d’une personne à partir de l’analyse de son ADN aura fait de grands progrès. (→) Voir le Forum de D. Casane et al., m/s n° 3, mars 2019, page 245  | Figure 1. Les frères Jim Lewis et Jim Springer, séparés à l’âge de quatre semaines et réunis pour la première fois 39 ans plus tard (CBS news, 12 août 2014). |
|
Origine géographique et groupes d’ascendance : utile mais peu précis La détermination des ascendances d’une personne grâce à l’analyse de son profil génétique (un demi-million de Snip1, déterminés grâce à un microarray) et à sa comparaison avec une série de profils-type a effectivement beaucoup gagné en précision et en fiabilité. Sans prendre pour argent comptant les affirmations d’une entreprise comme 23andMe
2, qui donne sur son site Internet l’exemple de l’analyse d’une certaine Jacqueline qui s’avérerait avoir 50,1 % d’ascendance européenne (dont 7,0 % française et allemande et 0,2 % scandinave), le tout au terme d’une étude facturée 99 dollars américains et sans aucune indication de l’intervalle de confiance, on peut reconnaître que de telles analyses révèlent de manière fiable la ou les principales composantes de l’ascendance d’une personne. On pourra alors indiquer que cet individu doit avoir un « type » européen, africain ou asiatique, ou éventuellement une combinaison de ces caractères. C’est une information importante mais, vue la variété phénotypique à l’intérieur de chacun des groupes d’ascendance, cela ne définit pas précisément son apparence, surtout dans le cas de plus en plus fréquent d’ascendances multiples. |
La pigmentation, sujet des principales avancées On pouvait déjà, il y a une dizaine d’années, avoir une idée de la couleur des yeux et de celle des cheveux grâce à l’analyse de six Snip pour les yeux et onze pour les cheveux [1], aboutissant à un système d’identification appelé IrisPlex [2] puis HIrisPlex [3]. Ces systèmes se composent en fait d’une liste de Snip à analyser (détermination des allèles présents dans l’ADN étudié) et d’un algorithme permettant à partir du jeu d’allèles trouvé, d’évaluer la probabilité pour différentes classes de couleur des yeux ou des cheveux. Notons que ces Snip sont tous situés dans des gènes plus ou moins impliqués dans le phénotype, et donc repèrent probablement la cause biologique de ces différences de pigmentation – et non des polymorphismes qui leur seraient simplement associés. Ces travaux, qui font l’objet d’une attention soutenue de la part des autorités de police, ont été activement poursuivis et ont fait/font l’objet de nombreux articles dans une revue spécialisée, Forensic Sciences International : Genetic, qui a publié une revue générale sur le sujet en 2015 [4]. Le dernier article paru, dont les auteurs seniors (Susan Walsh et Manfred Kayser) étaient à l’origine des systèmes IrisPlex et HIrisPlex, décrit un système appelé HIrisPlex-S qui inclut une prédiction de la couleur de la peau [5]. Comme indiqué précédemment [1, 6], cette caractéristique est nettement plus difficile à étudier (et donc à prédire) du fait de sa forte corrélation avec les groupes d’ascendance qui induit de nombreuses associations avec des Snip liés à cette ascendance et non de manière causale à la couleur de la peau. Il a donc fallu des études approfondies effectuées en étudiant 77 polymorphismes présents dans 37 gènes déjà plus ou moins associés à la pigmentation de la peau [6], et une validation sur 1 539 personnes appartenant à 59 populations, pour arriver à un jeu de 17 Snip et un algorithme associé permettant la prédiction de la couleur de la peau, répartie en cinq catégories [5]. Le système indique les probabilités respectives pour les différentes catégories (total égal à 1), ce qui permet de nuancer les résultats (Figure 2).
 | Figure 2.Prédiction de la couleur de la peau. À gauche : probabilités des différentes catégories ; à droite : photographie de l’intérieur de l’avant-bras de la personne concernée. Il faut combiner les deux valeurs notables (0,299 pour « intermé-diaire » et 0,693 pour « foncé » pour estimer la teinte effective (extrait partiel et modifié de la figure 6 de [ 5]). |
Au total, ce nouveau système HIrisPlex-S autorise donc une prédiction assez fiable de l’ensemble des caractères de pigmentation d’un individu à partir de l’analyse de son ADN pour un total de 41 Snip3, moyennant l’application d’un algorithme à ces résultats. Il faut d’ailleurs noter que les auteurs de ces travaux sont universitaires (Pays-Bas et États-Unis) et que leur système est totalement ouvert, utilisable par tous via un site Internet très clair et associé à un manuel d’utilisation détaillé (https://hirisplex.erasmusmc.nl/). À une époque où le moindre résultat susceptible d’une application fait immédiatement l’objet d’une création de start-up, la chose est assez rare pour devoir être soulignée. |
Forme du visage : la bouteille à l’encre Yeux, cheveux, teint : ce sont là des éléments importants pour la reconnaissance faciale, mais la forme du visage, ses traits, la bouche, le nez, les oreilles… sont aussi importants, sinon plus. Et ils sont largement déterminés par notre ADN, comme le montre la ressemblance des jumeaux homozygotes (Figure 1). Mais le nombre de gènes impliqués dans la forme du visage est très grand, et même la définition quantitative de cette forme reste problématique. Le sexe et l’ascendance permettent une première approche, mais c’est loin d’être suffisant. Certes, les techniques de reconnaissance faciale sont efficaces et sont largement utilisées tant pour autoriser l’accès à un téléphone portable que (en Chine au moins) pour la surveillance de la population – mais elles comparent un visage à un visage de référence, ou à tous ceux qui sont contenus dans une base de données, elles ne le prédisent pas à partir d’un ensemble d’informations génétiques. Et elles reposent sur des techniques d’intelligence artificielle et d’apprentissage dont le fonctionnement exact reste obscur. Un article de Craig Venter et de son entreprise Human Longevity qui prétendait réussir à identifier des personnes à partir de leur séquence d’ADN [7] a été sévèrement critiqué [8] et n’a pas eu de suite. Une approche originale récemment publiée [9] inverse le processus habituel : au lieu de chercher à prédire un visage à partir de l’ADN, on établit le profil ADN correspondant à chacun de nombreux visages de référence, et on compare ensuite le profil de l’ADN étudié à tous ces profils, aboutissant ainsi à quelques visages de référence qui doivent ressembler à celui qui correspond à cet ADN. Cette approche semble assez prometteuse, mais doit être poursuivie et largement testée pour savoir si elle est réellement utilisable. En tout état de cause, la déduction de la forme du visage et des traits reste l’enjeu majeur pour aboutir à une prédiction précise, et force est de constater que ce sujet n’a pas beaucoup avancé au cours de la dernière décennie. Les approches d’intelligence artificielle et d’apprentissage profond permettront-elles de dépasser ce blocage ? |
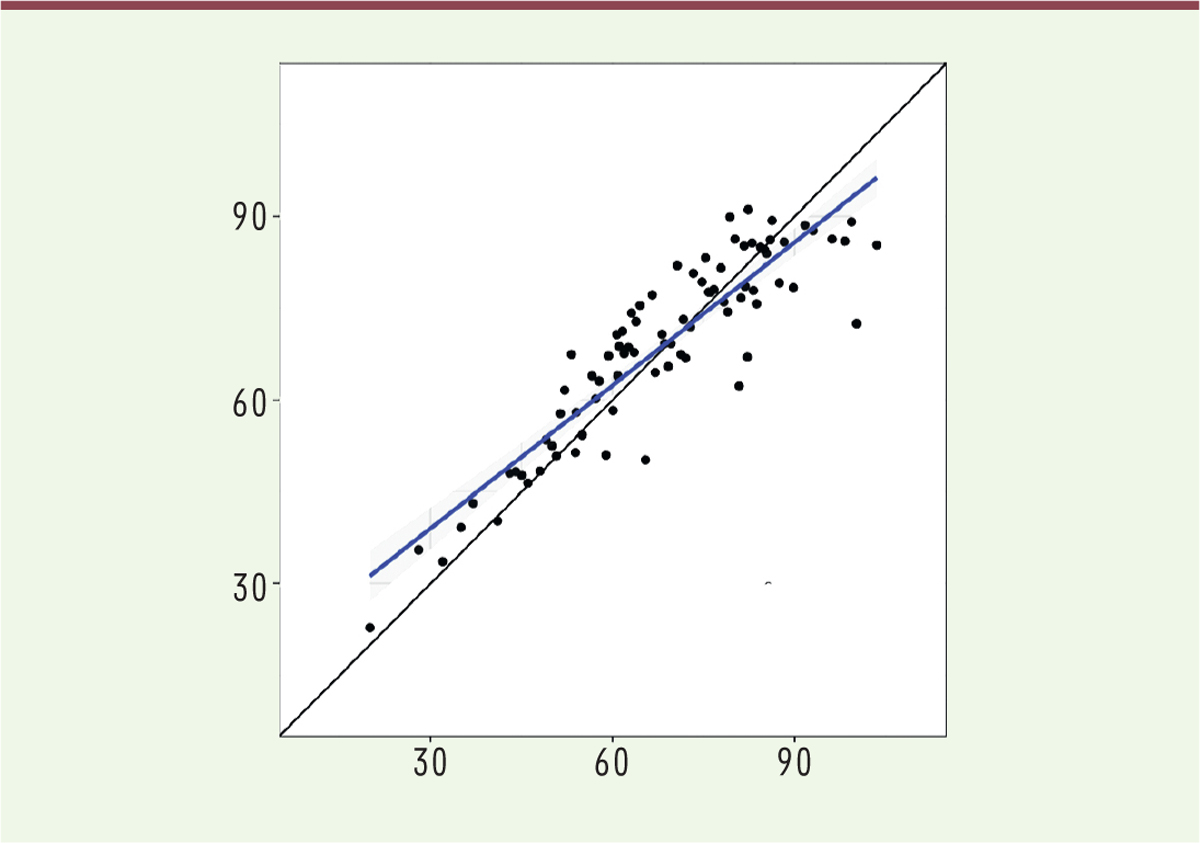
L’âge est à l’évidence un déterminant important pour l’apparence d’une personne. On pourrait penser qu’a priori il y a peu de chances de pouvoir l’évaluer à partir de l’ADN, molécule « immortelle » présente à l’identique dans (presque) toutes les cellules de l’organisme. Pourtant une méthode, déjà proposée il y a dix ans [1], reposait sur la fréquence des cercles d’excision des gènes du récepteur de l’antigène des lymphocytes T (TCR) et semblait assez prometteuse. Il ne semble pas que cette approche ait été poursuivie : mesurer ce paramètre dans des échantillons d’ADN de scène de crime était sans doute acrobatique du point de vue technique. Plus récemment, il a été montré que des modifications épigénétiques de l’ADN (la méthylation de la cytosine dans des dinucléotides CpG) étaient fortement corrélées à l’âge, et une équipe italienne [10] a pu mettre au point un test fondé sur l’analyse de huit doublets CpG qui, avec une analyse informatique appropriée, permet de déterminer l’âge à partir de l’ADN avec une marge d’erreur de l’ordre de quatre années (Figure 3).
 | Figure 3.Âge prédit (ordonnée) en fonction de l’âge réel (abscisse) pour 83 individus non utilisés pour paramétrer l’algorithme. En bleu, la droite de régression linéaire. On voit que les deux âges sont bien corrélés avec relativement peu de déviations (extrait partiel et modifié de la figure 4 de [ 10]). |
Ce test apporte donc une méthode assez précise et techniquement accessible (séquençage bisulfite4 de seulement huit doublets CpG) pour estimer l’âge d’une personne à partir de son ADN. Reste qu’elle a été étalonnée uniquement sur une population italienne (330 personnes non apparentées) et qu’il va falloir déterminer si elle reste valable pour d’autres populations. Notons aussi que cinq des huit doublets analysés se situent dans un seul gène, ELOVL2 (elongation of very long chain fatty acids-like 2), sur le rôle duquel les bases de données sont très peu bavardes – il serait impliqué dans la synthèse d’acides gras à très longue chaîne. Quoiqu’il en soit, il existe maintenant, sous réserve de réplication dans d’autres populations, une technique simple et rapide pour évaluer l’âge d’une personne d’après l’analyse de son ADN. |
Des progrès encore limités, une utilisation variable, des questions qui restent Finalement, si l’on compare la situation actuelle à celle qui prévalait lors de la précédente chronique sur ce sujet [1], les progrès semblent relativement modestes. Si l’origine géographique, les caractères de pigmentation et sans doute l’âge peuvent être déduits des analyses, les traits, la forme du visage, aspect essentiel de notre individualité, restent peu accessibles (bien que génétiquement déterminés) en raison de leurs complexités phénotypique et génétique. On est encore loin de pouvoir réellement obtenir un portrait-robot précis et ressemblant à partir d’un ADN « anonyme », quoiqu’en disent les médias – et contrairement à une impression sans doute majoritaire. La première approximation de l’aspect d’une personne peut cependant s’avérer très utile. Les approches présentées dans cette chronique ont, par exemple, permis d’avoir une idée du visage de nos lointains ancêtres, comme l’a montré la reconstruction du visage d’un Denisovien grâce à une analyse de l’ADN de Néandertal – aidée d’une bonne dose d’imagination [11]5. Elles peuvent aussi servir pour l’identification de restes humains après une catastrophe aérienne ou un attentat. Mais l’utilisation la plus fréquente et la plus médiatisée est celle que peut en faire la police, lorsqu’elle dispose d’ADN provenant de la scène de crime mais que le profil génétique classique (l’empreinte ADN reposant sur un peu plus d’une vingtaine de fragments dans une région hypervariable de l’ADN et qui est aussi caractéristique d’une personne que ses empreintes digitales) ne correspond à rien dans le FNAEG (fichier national automatisé des empreintes génétiques) français ou son équivalent hors de France [13] (→). (→) Voir la Synthèse de F. Iseni et J.N. Tournier, page 797de ce numéro L’intention est bonne, mais il s’agit là d’une analyse intrusive repérant de nombreux caractères de la personne, ce qui impose un encadrement très strict de la pratique – d’autant plus que la police, comme tout un chacun, aura tendance à s’exagérer la précision et la fiabilité des résultats : « l’ADN ne ment jamais »… De fait, les pratiques sont très différentes selon les nations. Aux États-Unis, ce forensic phenotyping est largement utilisé, de manière variable selon les États ; en France, il est théoriquement interdit puisque « Les empreintes génétiques […] ne peuvent être réalisées qu’à partir de segments d’acide désoxyribonucléique non codants, à l’exception du segment correspondant au marqueur du sexe ». Une décision de la Cour de Cassation en 2014 a semblé remettre en cause l’interdiction, mais cette dernière a été réaffirmée, comme l’indique le Tableau de l’Article A38 du code de procédure pénale, modifié par arrêté du 10 août 2015 (Livre IV, Titre XX) (https ://www.legifrance.gouv.fr/) [12]. La situation est très variable en Europe, allant d’une interdiction stricte en Allemagne à une autorisation large au Royaume-Uni, plus encadrée aux Pays-Bas, et à de nombreuses situations ambiguës [12]. On peut supposer que la technique est, en fait, assez largement utilisée par les services de police pour orienter la recherche des auteurs d’un crime, même si elle n’est pas admise comme preuve devant un tribunal. La tentation est sans doute forte d’utiliser cet outil pour faire avancer une enquête, même s’il n’est pas officiellement reconnu, et d’autant plus qu’on lui prête une précision et une fiabilité qu’il ne possède pas (encore ?). Pour conclure, on peut noter que la prédiction du phénotype d’une personne à partir de son ADN a fait des progrès notables au cours des dernières années, mais qu’elle n’a pas encore atteint la précision que l’on associe à l’image courante d’un « portrait-robot », et ceci principalement en raison de la difficulté à prédire les traits et la forme du visage. C’est d’un certain point de vue une bonne chose, car cela permet de faire avancer la réflexion et de mettre en place les garde-fous nécessaires pour éviter le fichage généralisé des citoyens que pratiquent déjà certaines nations. ‡ |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Footnotes |
1.
Jordan
B.
Nous avons les moyens de faire parler votre ADN . Med Sci (Paris). 2011; ; 27 : :667.–670. 2.
Walsh
S
,
Liu
F
,
Ballantyne
KN
, et al. IrisPlex: a sensitive DNA tool for accurate prediction of blue and brown eye colour in the absence of ancestry information . Forensic Sci Int Genet. 2011; ; 5 : :170.–180. 3.
Walsh
S
,
Liu
F
,
Wollstein
A
, et al. The HIrisPlex system for simultaneous prediction of hair and eye colour from DNA . Forensic Sci Int Genet. 2013; ; 7 : :98.–115. 4.
Kayser
M.
Forensic DNA phenotyping: predicting human appearance from crime scene material for investigative purposes . Forensic Sci Int Genet. 2015; ; 18 : :33.–48. 5.
Chaitanya
L
,
Breslin
K
,
Zuñiga
S
, et al. The HIrisPlex-S system for eye, hair and skin colour prediction from DNA: introduction and forensic developmental validation . Forensic Sci Int Genet. 2018; ; 35 : :123.–135. 6.
Walsh
S
,
Chaitanya
L
,
Breslin
K
, et al. Global skin colour prediction from DNA . Hum Genet. 2017; ; 136 : :847.–863. 7.
Lippert
C
,
Sabatini
R
,
Maher
MC
, et al. Identification of individuals by trait prediction using whole-genome sequencing data . Proc Natl Acad Sci USA. 2017; ; 114 : :10166.–10171. 8.
Reardon
S.
Geneticists pan paper that claims to predict a person’s face from their DNA . Nature News. 2017; ; 549 : :139.–140. 9.
Sero
D
,
Zaidi
A
,
Jiarui
L
, et al. Facial recognition from DNA using face-to-DNA classifiers . Nat Commun. 2019; ; 10 : :2557.. 10. Montesanto A, D’Aquila P, Lagani V, et al. A new robust epigenetic model for forensic age prediction . J Forensic Sci. 2020 May 26. doi: 10.1111/1556-4029.14460. 11.
Gokhman
D
,
Mishol
N
,
de Manuel
M
, et al. Reconstructing denisovan anatomy using DNA methylation maps . Cell. 2019; ; 179 : (180–92)
:e10.. 13.
Laurent
FX
,
Vibrac
G
,
Rubio
A
, et al. Les nouvelles technologies d’analyses ADN au service des enquêtes judiciaires . Med Sci (Paris). 2017; ; 33 : :971.–978. |