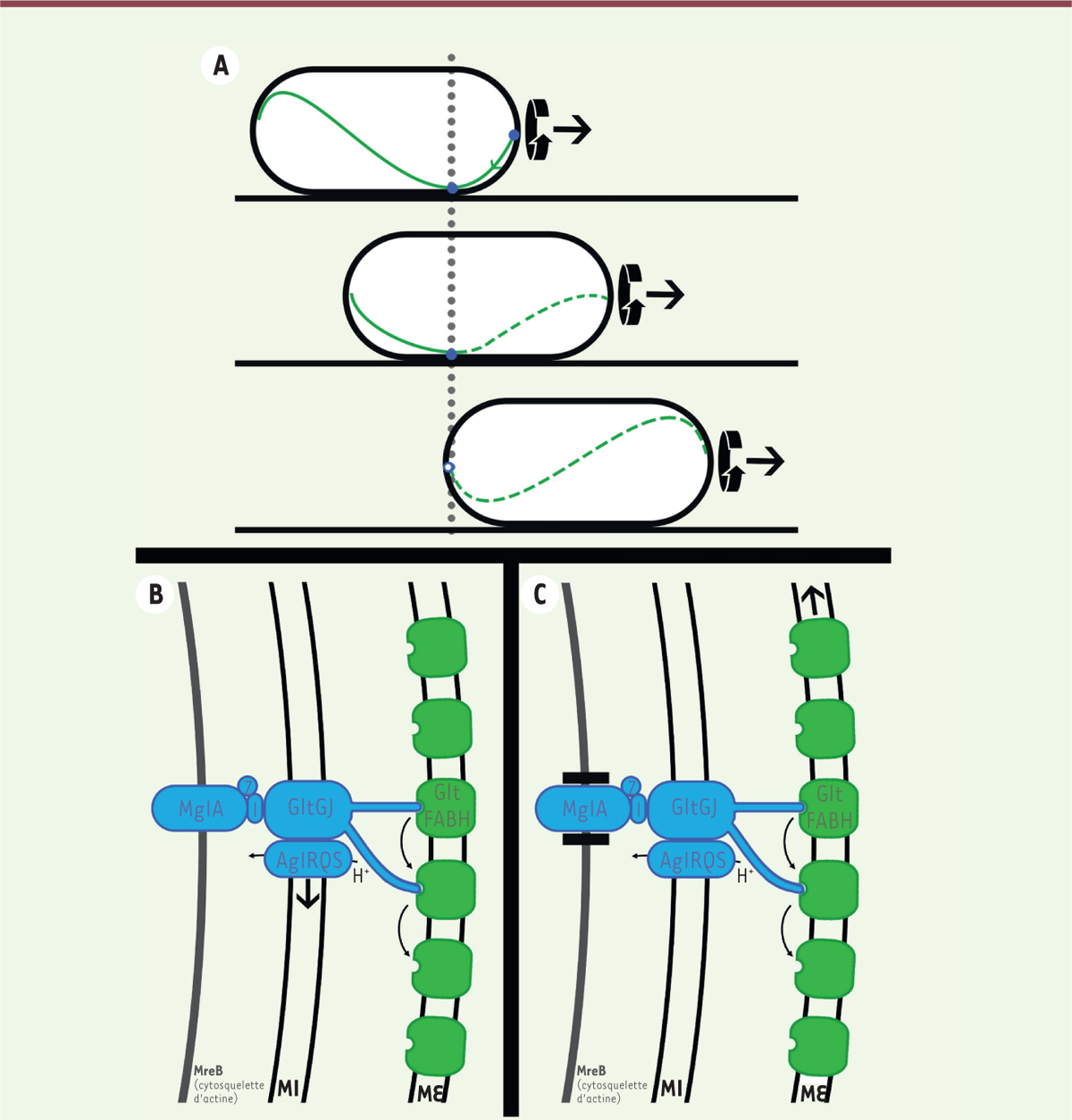
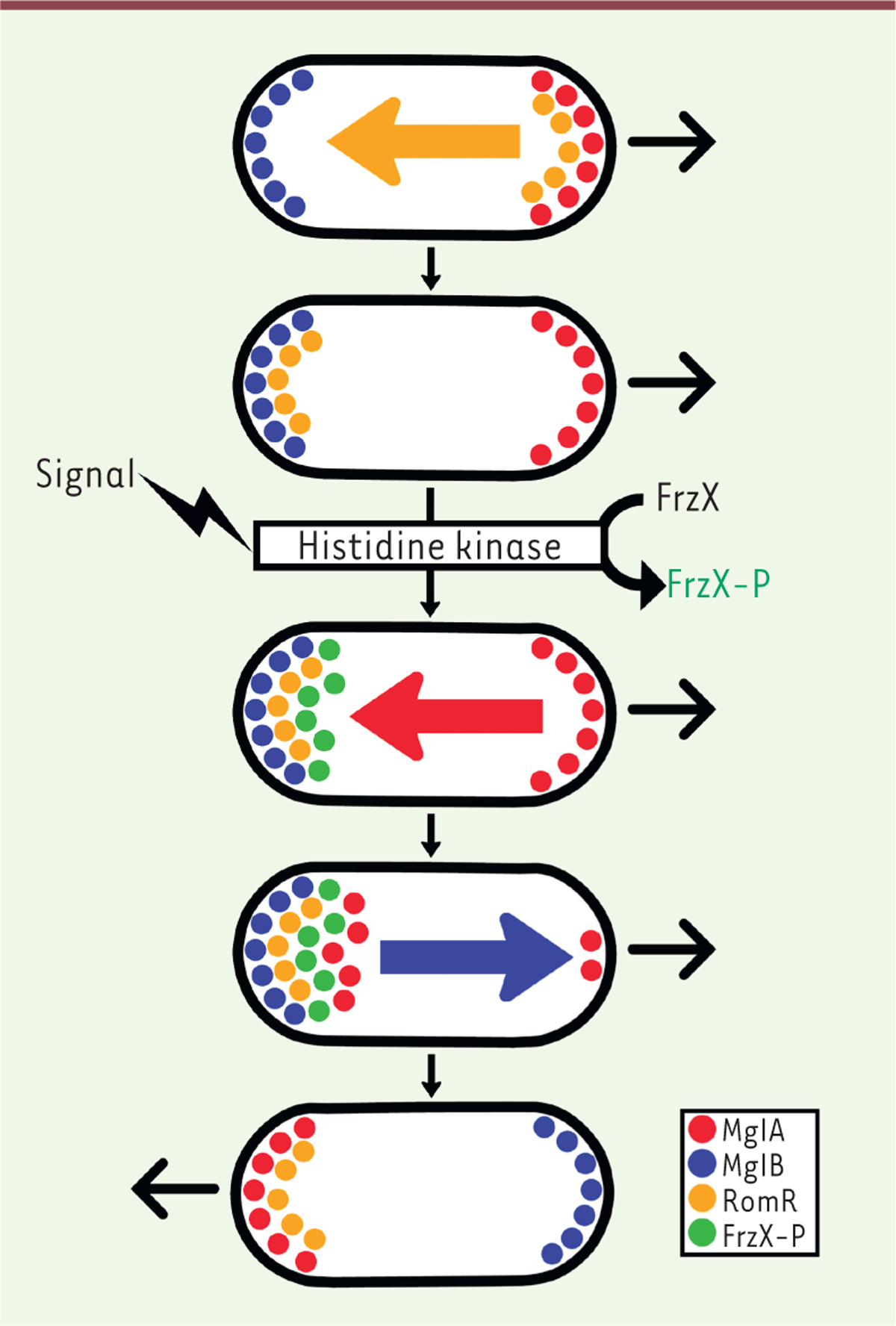
|
Entretien avec Tâm Mignot mené par les auteurs de la Nouvelle

Tâm Mignot est directeur du Laboratoire de Chimie Bactérienne (LCB), une
unité mixte de recherche du CNRS et de l’université d’Aix-Marseille au
sein de laquelle il dirige l’équipe « Biologie cellulaire de la motilité
bactérienne ». Son groupe est composé d’une vingtaine de scientifiques,
comprenant des chercheurs, des ingénieurs, des techniciens de
laboratoire et des doctorants. Tâm Mignot est également éditeur
scientifique pour le journal eLife. Il a reçu plusieurs
distinctions, dont le prix Bettencourt Schueller et la médaille de
bronze du CNRS en 2011. Quel est votre parcours ?
Tâm Mignot
J’ai grandi dans la région de Nice et j’ai passé un bac scientifique, le
bac C (en 1993). Mais je n’étais bon ni en maths ni en physique. Je me
suis donc orienté vers la biologie, en DEUG de sciences biologiques (en
1996) qui débouchait sur une licence de biochimie. Ensuite, j’ai fait
une maîtrise de biochimie et je me suis vraiment intéressé à la
microbiologie grâce à un cours de microbiologie que j’ai trouvé
fascinant. Je suis donc allé faire un DEA de microbiologie à l’Institut
Pasteur (en 1998) où j’ai trouvé un environnement qui m’a plu. J’ai donc
fait une thèse de microbiologie. J’ai travaillé sur la bactérie
responsable de l’anthrax (maladie du charbon) et sur la régulation des
gènes qui permettent de modifier l’enveloppe bactérienne au cours de
l’infection. J’ai soutenu ma thèse en 2002. Je suis ensuite parti en
postdoc à l’université de Californie, à Berkeley, et je suis tombé par
hasard sur un laboratoire qui étudiait une bactérie fascinante qui était
Myxococcus xanthus. J’ai donc travaillé sur cette
bactérie lors de mon postdoc ; c’était l’époque où l’on commençait les
approches cellulaires dans les bactéries. On voyait les cellules changer
de direction, mais personne n’avait encore montré qu’il existait des
systèmes qui contrôlaient la localisation des protéines, qui les
relocalisaient d’un pôle à l’autre. J’ai mis en place les expériences
qui permettaient de voir : 1) la réversion et, en faisant ces
manipulations, de voir 2) les points d’adhérences focales pour la
première fois. Mon postdoctorat a duré quatre ans. Puis, je suis rentré
en France où j’ai proposé au CNRS de me recruter sur un projet où
j’allais poursuivre la caractérisation de ces phénomènes. J’ai pris mes
fonctions de chargé de recherche en 2007 au Laboratoire de Chimie
Bactérienne (LCB) où j’ai monté une équipe. En 2014, j’ai été promu
directeur de recherche et je suis devenu le directeur du LCB en
2018. Qu’est-ce qui vous a mené vers la science ? TM : Je ne sais pas trop, je pense que j’étais plutôt
littéraire. En terminale, au lycée, j’ai eu un cours sur le code
génétique et là, j’ai été fasciné par son expression, l’ADN, le côté
incroyablement prédictif du code. Et c’est le moment où j’ai décidé que
j’allais faire de la biologie moléculaire. Jusqu’à la maîtrise, ce
n’était pas complètement clair mais à la sortie, les choses ont été
assez précises : je voulais continuer dans cette voie, j’allais faire
une thèse, un postdoctorat, puis tenter de me faire recruter. Quelle est votre plus grande fierté, réussite en
science ? TM : Je pense sans arrogance que j’ai élucidé un nouveau
mécanisme de mouvement dans la cellule, et c’était quelque chose
d’énorme car c’est très rare d’avoir la chance de pouvoir expliquer un
mécanisme aussi complexe. Évidemment, il y a des trous et il y a
certainement des choses fausses dans ce qu’on propose. Mais le modèle,
le principe global, je pense, est correct. Il a tenu. Quand j’ai proposé
les sites d’adhérences focales en postdoctorat, il y avait beaucoup de
scepticisme. De constater que 1) d’autres laboratoires le
reproduisaient, et 2) de trouver des molécules, un moteur, des
adhésines, et qu’on arrivait à construire une machine qui résistait à
l’analyse, alors notre hypothèse de départ était suffisamment
« costaude » pour arriver à travailler dessus. Et, finalement, arriver à
publier dans Nature en 2016 le mécanisme qui est
maintenant ancré dans les bouquins, j’avoue que c’est une sacrée fierté
au niveau professionnel. En sachant qu’il y a eu beaucoup de doutes,
mais j’étais toujours très confiant, parce que si je vois ces foyers
fixes dans une cellule en mouvement c’est qu’ils sont en mouvements
opposés, c’est de la logique pure. Et s’ils sont en mouvement opposés
alors forcément, ils sont propulseurs. Cela m’a donné une très grosse
base de confiance sur la solidité du projet. Ça va être la fin d’un
chapitre pour moi, on ne va pas aller beaucoup plus loin, maintenant on
va aller vers les mouvements de groupe. Quelle est la chose la plus étonnante que vous ayez
découverte ? TM : Ça, je l’ai fait en thèse où j’ai réalisé que l’on doit
regarder les choses avec la plus grande ouverture d’esprit possible.
Cela m’est arrivé à deux reprises dans ma carrière. Pendant ma thèse, je
regardais, par exemple, l’expression d’un gène et il y avait deux
protéines qui semblaient faire la même chose, et on se demandait quels
étaient leurs rôles respectifs. Donc j’étais parti sur l’étude de la
régulation de ces protéines et je voyais qu’il y avait des effets de
régulation, je voyais des choses intéressantes mais cela ne m’expliquait
toujours rien par rapport à ces protéines. Puis un jour, je regarde ces
deux courbes et c’était hyper simple : il y en avait une exprimée à un
moment puis l’autre exprimée après. Et d’un coup ça me frappe : elles
font la même chose mais ne sont pas exprimées au même moment, c’est
qu’il y en une qui remplace l’autre. Qu’importe le mécanisme et son
importance, l’essentiel est surtout de réaliser que j’avais eu ces
données pendant trois mois sous les yeux et soudain en regardant les
courbes, je change d’état d’esprit : je ne regarde plus avec en tête la
question initiale, et, d’un coup, cela m’apparaît… c’est là que ma thèse
a commencé. Donc, parfois il faut sortir du cadre, se poser une autre
question, regarder ce qu’on a sous les yeux et c’est là où l’on peut
découvrir des choses. Il faut être préparé à cela.
|