| |
| Med Sci (Paris). 36(4): 412–415. doi: 10.1051/medsci/2020066.Diversité des mécanismes de transcription des virus
géants Manon Dassa-Valzer,1*a Romain Debiton,1*b Margaux Gibert,1*c Alexandre Lutz,1*d and Amel Latifi2e 1Master 2 Microbiologie Intégrative et Fondamentale, Aix
Marseille Université, Marseille, France 2Aix Marseille Université, CNRS, LCB UMR,
7283, IMM, Marseille,
France MeSH keywords: Variation génétique, Génome viral, Virus géants, Mimiviridae, Phylogenèse, Transcription génétique, Assemblage viral, classification, génétique |
Avec son génome de 1,2 Mb et sa capside de 450 nm, Mimivirus, le premier virus géant
identifié en 2003 [1], a marqué
une rupture dans le monde microbien. Les techniques de filtration utilisées pour
l’isolement des virus avaient en effet occulté l’existence des virus géants dont la
taille des capsides entraînait leur rétention par les filtres. Depuis la découverte de
Mimivirus, de nombreux autres virus géants ont été identifiés. Ils possèdent des
capsides allant de 300 nm à 2 µm et des génomes à ADN double brin variant entre 288 kb
et 2,5 Mb. Bien qu’ils possèdent parfois un génome plus grand que certaines bactéries,
ce sont des parasites obligatoires d’organismes eucaryotes unicellulaires [2]. Leurs hôtes sont retrouvés dans
tous les écosystèmes, y compris certains environnements extrêmes comme les cheminées
hydrothermales, ou des environnements plus communs comme les sols, le pergélisol, les
eaux ou encore le microbiote intestinal. Bien que des virus géants puissent être
véhiculés chez l’homme, leur présence ne s’accompagne pas d’une pathogénicité. Étant
donné leur grande diversité, notamment au niveau de leurs génomes et de leurs cycles
infectieux, il est difficile d’avoir une classification universelle. La diversité
génomique est illustrée par une forte représentation de gènes ne possédant aucune
homologie ni avec les gènes d’autres familles de virus ni avec le monde cellulaire, mais
souvent conservés au sein d’une même famille de virus. Ces gènes sont connus sous le
terme de gènes orphelins. Il existe différents mécanismes d’entrée et de sortie des
virions, qui aboutissent tous au transfert du contenu de la particule virale dans le
cytoplasme de l’hôte et à la libération de particules virales dans le milieu. Les
différences inter-virus au niveau des cycles infectieux sont liées à la dépendance au
noyau cellulaire pour les mécanismes de réplication de l’ADN et de transcription des
gènes. Cet article résume les différents mécanismes de transcription connus à ce jour à
travers trois exemples majeurs. |
Mimivirus, le géant de l’autonomie La famille des Mimiviridae est la première famille de virus géants
identifiée [11,12] (→).
(→) Voir la Nouvelle de M. Bekliz et al.,
m/s n° 10, octobre 2016, page 818, et la Synthèse de
J.M. Claverie et C. Abergel, m/s n° 12, décembre 2016,
page 1087
Ils sont capables de réaliser leurs cycles infectieux de façon quasi-autonome, pour
les fonctions nucléaires, dans leur hôte cellulaire. En effet, immédiatement après
l’infection, les Mimiviridae initient la transcription de leur
génome. Ceci est possible car ils possèdent tous les gènes permettant la synthèse
d’une machinerie de transcription autonome (ARN polymérase virale). Ils utiliseront
ensuite la machinerie de traduction de l’hôte pour traduire leurs ARN messagers. Ils
sont de plus capables d’embarquer cette machinerie dans leurs capsides. Ceci va
permettre la transcription des gènes précoces immédiatement après l’infection
(Figure 1). Ces
derniers gènes présentent un promoteur unique, conservé, différent des promoteurs
cellulaires et de ceux utilisés par les gènes tardifs [3]. En plus d’être autonomes pour la
transcription, ces virus le sont également pour la maturation de leurs ARNm (voir
Encadré « maturation des ARN messagers ») grâce à leur capacité
d’intégrer les facteurs de maturation dans leur capside. En 2009, l’analyse des
génomes de ces virus a permis de proposer un mécanisme selon lequel un ARN
palindromique gouverne l’arrêt de la transcription et la maturation des transcrits
(règle de l’épingle à cheveux) [4]. Ce mécanisme, caractérisé en 2015, est assuré par la polyA
polymérase virale qui présente une structure originale et la capacité unique
d’ajouter de longues queues polyA au niveau des structures en épingle à cheveux
[5]. Ceci augmente la
stabilité des ARNm viraux, retardant leur dégradation, favorisant ainsi la
traduction virale par rapport à la traduction cellulaire. Ce sont ces spécificités
qui vont différencier les Mimiviridae des autres virus présentés
dans cet article.
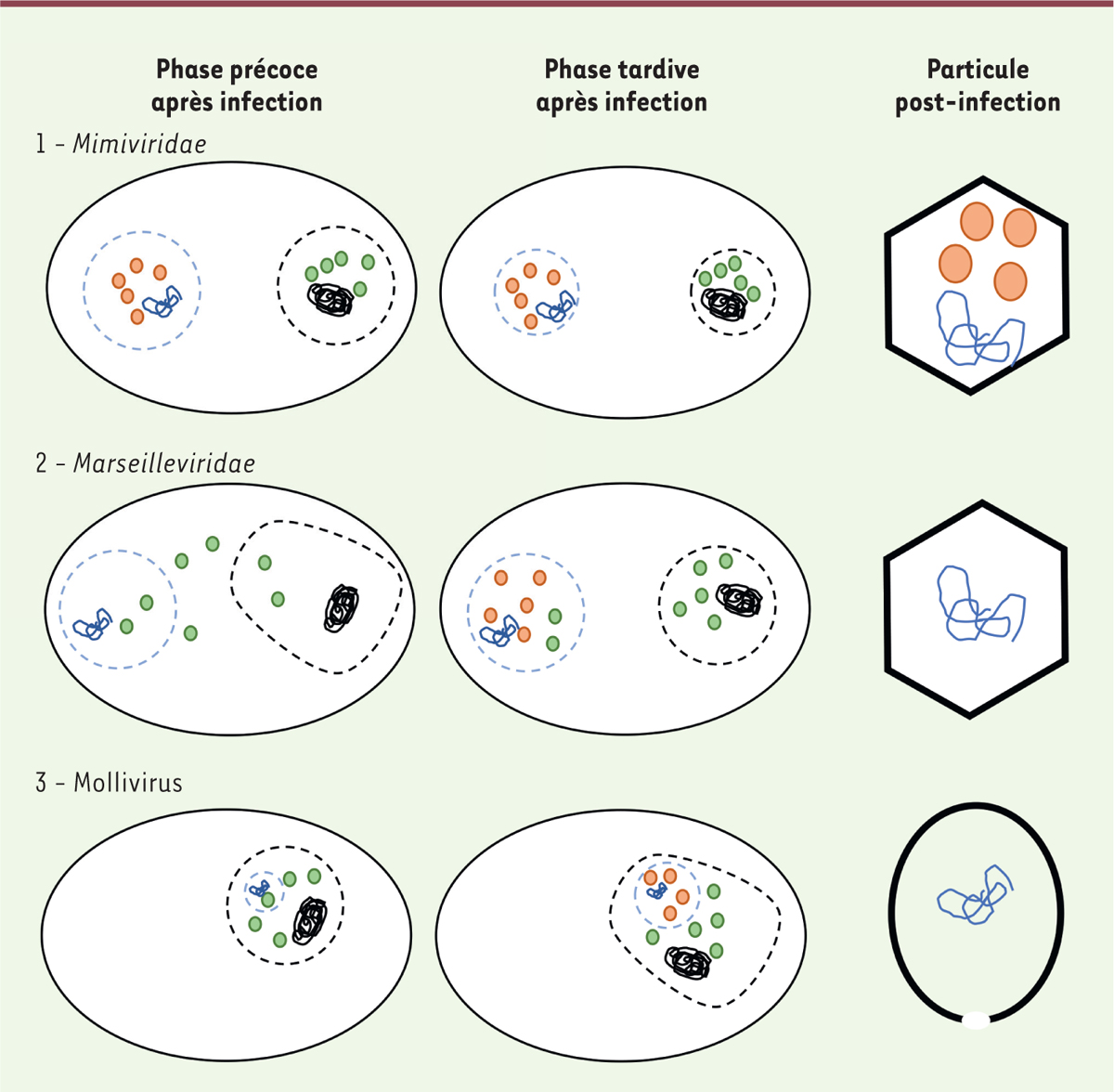
 | Figure 1. Représentation schématique de différents mécanismes de
transcription chez les virus géants. 1. Le virus
commence la transcription de ses gènes précoces dans l’usine virale en
utilisant sa propre machinerie. Dans la phase tardive, le virus va
continuer son cycle infectieux de façon autonome, pour finalement
libérer des particules virales contenant son génome ainsi que sa propre
machinerie de transcription. 2. Le virus
va recruter la machinerie de transcription de l’hôte, en la faisant
migrer du noyau vers l’usine virale. Ceci a pour effet de déstructurer
le noyau de l’hôte. Par la suite, le virus utilise sa propre machinerie
de transcription néosynthétisée pour la transcription des gènes tardifs.
Finalement, il libèrera des particules virales contenant son génome,
mais sera incapable d’encapsider sa propre machinerie.
3. L’usine virale va se former
dans le noyau de l’hôte et détournera la machinerie de transcription de
ce dernier. En phase tardive, le virus continue son cycle infectieux
dans le noyau mais en utilisant sa propre machinerie néosynthétisée,
conduisant à la déstructuration du noyau. La capside virale libérée
contient le génome viral. Traits pleins noirs : ADN de l’hôte. Traits
pointillés noirs : noyau de l’hôte. Traits pleins bleus : ADN viral.
Traits pointillés bleus : usine virale. Ronds verts : machinerie de
transcription de l’hôte. Ronds orange : machinerie de transcription
virale. |
|
Mollivirus sibericum, le géant casanier Mollivirus sibericum, virus géant isolé dans le pergélisol sibérien, est un exemple
intéressant de virus détournant la transcription de son hôte en installant son usine
virale au sein du noyau cellulaire. Une approche de protéomique a permis de
décrypter toutes les étapes du cycle de ce virus, ce qui a permis d’avoir une vision
globale de cette infection [6]. Cette étude a montré que, bien que codant pour une ARN polymérase,
celle-ci ne se retrouve pas dans la capside. Lors de la phase précoce, la machinerie
cellulaire permet la production de l’ARN polymérase virale, qui prend le relais pour
le reste du cycle infectieux. La microscopie à fluorescence a également montré le
transport de l’ADN viral vers le noyau cellulaire dès le début de l’infection.
Toutefois le mécanisme précis du transfert de celui-ci à travers le cytoplasme vers
le noyau cellulaire reste encore à élucider. Une fois le matériel viral introduit
dans le noyau, ce dernier se déstructure partiellement, permettant ainsi l’accès de
l’ADN viral à la polymérase cellulaire (Figure
1). Ce mécanisme est également partagé avec les virus de la
famille des Pandoraviridae[7,8],
pourtant très éloignés de Molliviridae. Ces virus s’appuient ainsi
sur la machinerie de transcription cellulaire en la détournant à leur profit. Les cycles infectieux des deux familles virales présentés ci-dessus possèdent ainsi
des niveaux de dépendance nucléaire opposés, mais il existe d’autres virus ayant un
niveau de dépendance intermédiaire. |
Marseilleviridae, le géant indécis Les Marseilleviridae ont été considérés pendant longtemps comme des
virus exclusivement cytoplasmiques parce qu’ils codent une machinerie de
transcription complète. Or l’analyse du protéome des virions a révélé l’absence
d’ARN polymérase, ce qui impose l’utilisation de la machinerie cellulaire, au moins
pour les gènes précoces [9].
En effet, une étude par microscopie électronique et microscopie à fluorescence a
montré, en 2017, que, lors de la phase précoce, le noyau de la cellule hôte se
déforme pour permettre la migration de protéines du noyau de la cellule hôte vers
l’usine virale. Ce processus se fait sans la libération de l’ADN de l’hôte,
éliminant l’hypothèse d’une dégradation totale du noyau. Le modèle proposé pour ces
virus est qu’ils seraient capables de recruter la machinerie de transcription de
leur hôte, en l’extrayant du noyau et en la faisant migrer vers l’usine virale
formée dans le cytoplasme (Figure
1). Ce phénomène leur permettrait d’assurer la transcription des
gènes précoce, notamment les gènes codant pour la machinerie de transcription et de
maturation des ARNm viraux. La migration des protéines nucléaires et la localisation
cytoplasmique de l’usine virale reflètent un mécanisme de dépendance au noyau
intermédiaire à ceux vus précédemment. |
Entretien avec Chantal Abergel mené par les auteurs de la
Nouvelle

Chantal Abergel est chercheure en microbiologie avec un tropisme particulier
pour la virologie. Suite à son implication dans la découverte et la
caractérisation d’un nouveau groupe de virus, les « virus géants » en 2003,
elle consacrera le reste de sa carrière à l’étude de cette nouvelle famille
émergente. Elle est la co-fondatrice du laboratoire « Information Génomique
et Structurale » (IGSMarseille Luminy) au sein duquel elle exerce en tant
que directrice de recherche. Elle a reçu la médaille d’argent du CNRS en
2014 et le prix « La Recherche » en 2015, ainsi que la distinction
d’Officier de l’Ordre National du Mérite en 2019.
Quel est votre parcours académique ?
Chantal Abergel
Parcours universitaire à l’université de Marseille-Luminy jusqu’en Master 1.
Après, j’ai quitté pour un moment le cursus car je voulais partir en
Amérique du Sud pour faire mon Master 2, mais la personne que je devais
rencontrer à Genève pour démarrer ce Master 2 n’est jamais venue… J’ai donc
interrompu mon cursus à ce moment. J’ai travaillé au Service de santé des
armées en parasitologie en cherchant à acquérir des connaissances dans ce
domaine. Cela m’a permis d’intégrer un Master l’année d’après dans un
laboratoire qui travaillait sur les calculs pancréatiques. Malheureusement,
c’était la première année de cette formation et le responsable avait oublié
de demander les bourses de thèses… Il a donc fallu que je recherche une
thèse. Par chance, une bourse CIFRE co-financée par Matra espace était
proposée par un laboratoire qui travaillait sur la cristallisation des
protéines. Ayant des connaissances dans ce domaine, j’ai obtenu cette
bourse. Le projet de thèse portait sur l’étude des paramètres critiques à la
cristallisation des protéines, dont la microgravité. J’ai donc fait une
thèse en science des matériaux et j’ai pu faire voler des cristaux dans
l’espace ! Après ma thèse, je suis partie cinq ans en post-doctorat aux NIH
(National Institutes of Health) à Bethesda, aux États-Unis.
Comment a été créé le laboratoire Information Génomique et
Structurale ?
CA : De retour en France, avec Jean-Michel Claverie,nous avons
eu l’opportunité en 1995 de créer un laboratoire de bio-informatique et de
biologie structurale : l’IGS. Nous nous sommes alors intéressés à des
projets qui étaient centrés sur l’analyse et la comparaison des génomes. On
a donc démarré un petit programme de génomique structurale qui portait sur
la production des protéines et leur caractérisation structurale. Nous avons
également eu un financement important en proposant un projet pour rechercher
de nouvelles cibles à de nouveaux antibiotiques en comparant les génomes de
bactéries pathogènes à ceux des bactéries non pathogènes.
Comment l’IGS s’est-il orienté vers les virus géants ?
CA : L’expertise du laboratoire en génomique a fait que de
nombreuses personnes nous ont contactés pour faire du séquençage et de
l’annotation de génomes, notamment des génomes bactériens. Parmi eux, Didier
Raoult, qui était un spécialiste des bactéries parasites, a demandé à
collaborer avec le laboratoire sur cette thématique. Il s’avère qu’aux
alentours des années 2000, un échantillon est arrivé d’Angleterre dans le
laboratoire de Didier Raoult dans le but de caractériser l’organisme
pathogène infectant les amibes. Notre laboratoire a été impliqué sur
l’aspect génomique de ce travail et c’est nous qui avons procédé à
l’annotation du génome. Et c’est là que nous avons découvert Mimivirus. Cela
a été une fascination immédiate à cause de toutes les surprises que
présentaient Mimivirus. On a décidé de ne pas lâcher les virus géants et on
a basculé vers un développement de nouvelles méthodes qui nous permettaient
de les étudier. Et à partir de là, nous avons non seulement continué à
travailler sur Mimivirus mais nous avons également cherché à isoler d’autres
virus géants pour démontrer qu’ils étaient ubiquitaires, qu’on les avait
juste occultés à cause d’un processus de filtration utilisé depuis longtemps
et imposé par des traditions historiques
Actuellement, qu’est-ce qui est fait dans votre laboratoire
?
CA : Nous essayons de faire le plus de choses possibles.
C’est-à-dire qu’on utilise les techniques et les méthodologies qui existent
à l’heure actuelle pour essayer d’avoir le plus de réponses sur les cycles
infectieux de ces virus, sur leur mode de fonctionnement, le rôle des
protéines de ces virus et l’interaction qui existe entre ces virus et la
cellule, voire même dans des systèmes plus complexes (virus géant,
virophage, transpoviron). On utilise toutes les techniques d’imagerie
possibles. On essaye également d’implémenter les mutations dans la cellule
pour pouvoir réaliser des études fonctionnelles sur les gènes essentiels
pour ces virus. Nous faisons de la résolution de structure, en tentant de
comprendre la fonction de chacune de ces protéines avec des questions plus
vastes qui sont de rétablir les voies métaboliques. Enfin, nous essayons
d’établir des liens entre les protéines qui sont codées dans le génome de
ces virus. Globalement, nous essayons de répondre, par toutes les techniques
possibles et imaginables, à tout un panel de questions biologiques.
Qu’est-ce qui vous intéresse le plus dans l’étude des virus géants ?
Est-ce que c’est d’établir toute la mécanistique que va avoir un virus
pour son cycle infectieux ou est-ce la découverte d’un nouveau virus
?
CA : Les deux. Mais plus que ça, il y a une question de fond qui
est « Quelle est la place des virus dans l’évolution ? Quelle est la place
des virus par rapport au monde cellulaire ? ». Et ces virus géants nous ont
permis de nous introduire et de nous positionner avec ce genre de questions
dans le domaine de la virologie. Redéfinir la notion de virus, les replacer
dans un contexte évolutif par rapport au monde cellulaire.
Est-ce vous qui avez eu l’idée d’aller chercher les virus géants à
tel ou tel endroit, ou ce sont les recherches qui vous orientent sur le
prochain site ?
CA : L’idée du pergélisol, c’est Jean-Michel Claverie qui l’a
eue. Un article publié dans PNAS montrait qu’il était possible de réactiver
une plante à partir d’un fragment de fruit congelé dans le pergélisol. S’il
était possible de réactiver un organisme aussi complexe qu’une plante à
fleurs, réactiver un virus devait être également possible. Donc il a pris
contact avec les gens qui travaillent là-dessus. Mais avant ça, on avait une
logique. Nous avons organisé notre première mission au Chili, dans le
Pacifique, car c’est un lieu où il y a beaucoup de nutriments, beaucoup de
brassages qui permettent d’avoir un biotope hyper-varié et très riche.
Progressivement, on s’est rendu compte qu’en ramassant un peu de terre sous
l’arbre situé à l’entrée du laboratoire, il était possible de réactiver des
Pandoravirus. Au début, on ne pensait pas qu’ils étaient aussi abondants
dans l’environnement. Maintenant, on le sait, ils sont partout et on veut
tous les attraper, comme les Pokémon !
Quel est votre virus géant préféré, est-ce qu’il y en a un qui sort
un peu du lot ?
CA : Chacun sort du lot à sa manière, mais mes préférés sont les
Pandoravirus car ils sont tellement différents de tous les virus à ADN ! Il
y à 90 % du génome dont on n’a pas la moindre idée de la fonction. Donc je
pense que ce sont ceux-là qui peuvent avoir des voies métaboliques
originales. On a découvert récemment qu’ils étaient capables de créer leurs
propres gènes ab initio. Au niveau évolutif, c’est magistral car cela veut
dire que ces gènes n’ont pas d’histoire. Il y a encore de la « créativité »
qui peut apparaître.
|
|
Maturation des ARN messagers Chez les eucaryotes, l’ADN est transcrit en ARN-pré messager qui sera ensuite
transformé en ARNm. Il existe trois grandes modifications : l’addition de la
coiffe à l’extrémité 5’, permettant la protection du transcrit contre la
dégradation, son exportation vers le cytoplasme, et sa traduction. Elle
consiste en la modification du premier nucléotide transcrit, catalysée par
la guanylyl transférase et la guanine-7-méthyltransférase. Suite à cela,
l’étape d’épissage consiste en l’excision des introns (séquences non
codantes de l’ARN primaire) et en l’assemblage bout à bout des exons
(séquences codantes). Ces deux étapes se font de manière
co-transcriptionnelle. Pour finir, l’addition d’une queue polyadénylée
(200 nucléotides environ) à l’extrémité 3’, est une étape
post-transcriptionnelle réalisée par la polyA polymérase, permettant la
stabilité de l’ARNm et sa traduction. La maturation de l’ARNm est un
processus indispensable à sa traduction [10]. Les virus vont donc aussi devoir
effectuer ces modifications afin de pouvoir réaliser la traduction de leurs
ARN et donc effectuer leur cycle infectieux.
|
|
Synthèse et ouverture sur les mécanismes de transcription chez les virus
géants Depuis leur découverte, les virus géants n’ont cessé de susciter l’intérêt, notamment
à cause de la diversité des environnements où ils sont retrouvés, de leurs tailles,
formes, cycles infectieux, etc. Dans cet article, nous avons synthétisé les études
récentes réalisées sur les mécanismes de transcription virale et catégorisé au moins
trois grands procédés. Les virus les plus autonomes, qui possèdent l’intégralité des
protéines nécessaires à la transcription et à la maturation de leur ARNm, illustrés
ici par les Mimiviridae dont la transcription est cytoplasmique. Un
second système à l’opposé de ce dernier, est utilisé par Mollivirus sibericum et les
Pandoraviridae qui, eux, sont nucléaires et détournent la
machinerie de transcription de l’hôte en phase précoce. Enfin, les
Marseilleviridae utilisent un système intermédiaire, qui
requiert la migration de la machinerie de l’hôte du noyau vers l’usine virale. Les
données rassemblées jusqu’à ce jour ne permettent pas d’avancer un réel lien
évolutif entre tous ces mécanismes, ni d’éliminer l’hypothèse que l’un d’entre eux
puisse être la version la plus aboutie. Toutefois, si un parallèle est réalisé avec
le schéma évolutif le plus fréquent chez les pathogènes intracellulaires, qui
consiste en une réduction génomique, favorisant le détournement des machineries de
leur hôte, il serait possible d’imaginer une évolution d’un système de cycle viral
cytoplasmique et autonome vers un système nucléaire et dépendant de l’hôte.
L’évolution des virus géants est une des questions ouvertes et des plus
intéressantes qui restent à élucider en virologie. Ainsi, il se peut que des études
plus approfondies des cycles infectieux et des nombreux gènes viraux orphelins
permettent de trouver un début de réponse à cette question. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
1.
Scola
BL
. A giant virus in Amoebae .
Science.
2003; ; 299 : :2033.. 2.
Brandes
N
,
Linial
M
. Giant viruses. Big surprises .
Viruses.
2019; ; 11 : :404.. 3.
Raoult
D.
. The 1.2-megabase genome sequence of
mimivirus . Science.
2004; ; 306 :
:1344.–1350. 4.
Byrne
D
,
Grzela
R
,
Lartigue
A
, et al.
The polyadenylation site of mimivirus transcripts obeys a
stringent hairpin rule . Genome Res.
2009; ; 19 :
:1233.–1242. 5.
Priet
S
,
Lartigue
A
,
Debart
F
, et al.
mRNA maturation in giant viruses: variation on a
theme . Nucleic Acids Res.
2015; ; 43 :
:3776.–3788. 6.
Legendre
M
,
Lartigue
A
,
Bertaux
L
, et al.
In-depth study of Mollivirus sibericum, a new 30,000-y-old giant
virus infecting Acanthamoeba . Proc Natl Acad Sci
USA.
2015; ; 112 :
:E5327.–E5335. 7.
Philippe
N
,
Legendre
M
,
Doutre
G
, et al.
Pandoraviruses: Amoeba viruses with genomes up to 2.5 Mb reaching
that of parasitic eukaryotes . Science.
2013; ; 341 :
:281.–286. 8. Pereira Andrade AC
dos
S, Victor de
Miranda Boratto
P,
Rodrigues
RAL, et al.
New isolates of pandoraviruses: contribution to the study of
replication cycle steps . J Virol.
2018;; 93 :
e01942–18/jvi/93/5/JVI.01942-18.atom.. 9.
Fabre
E
,
Jeudy
S
,
Santini
S
, et al.
Noumeavirus replication relies on a transient remote control of
the host nucleus . Nat Commun.
2017; ; 8 : :15087.. 10.
Bentley
DL
. Coupling mRNA processing with transcription in
time and space . Nat Rev Genet.
2014; ; 15 :
:163.–175. 11.
Bekliz
M
,
Levasseur
A
,
La Scola
B
,
Raoult
D
. MIMIVIRE, un système de défense chez mimivirus
qui illustre l’hypothèse de la Reine Rouge . Med Sci
(Paris).
2016; ; 32 :
:818.–819. 12.
Claverie
JM
,
Abergel
C
. Les virus géants. État des connaissances,
énigmes, controverses et perspectives . Med Sci
(Paris).
2016; ; 32 :
:1087.–1096. |