| |
| Med Sci (Paris). 36(4): 326–329. doi: 10.1051/medsci/2020049.Exploiter les dépendances non-oncogéniques afin
d’identifier de nouvelles cibles thérapeutiques dans les cancers
pédiatriques Virginie Menez1 and Marie-Anne Debily1,2* 1Inserm U981, Molecular predictors and new
targetsin oncology, Gustave Roussy, Université
Paris-Saclay, 94805Villejuif,
France 2Univ. Évry, Université Paris-Saclay,
91000Évry,
France MeSH keywords: Âge de début, Tumeurs du cerveau, Enfant, Dépistage génétique, Gliome, Humains, Oncologie médicale, Thérapie moléculaire ciblée, Tumeurs, Protein-Serine-Threonine Kinases, Mutations synthétiques létales, génétique, mortalité, thérapie, méthodes, tendances, épidémiologie |
Les cribles de létalité synthétique, un espoir pour la recherche en
cancérologie Depuis 2007, les techniques de séquençage de l’ADN à haut débit ont révolutionné la
biologie et, en association avec les outils d’« ARN interférence » utilisés pour
éteindre sélectivement l’expression d’un gène, ont permis une avancée majeure en
génomique fonctionnelle en facilitant les criblages systématiques de « perte de
fonction » dudit gène. La comparaison de cellules tumorales et de leur contrepartie
non tumorale, dépourvue d’altération oncogénique, permet d’identifier rapidement les
gènes dont l’inhibition permet de tuer uniquement les cellules cancéreuses. De cette
façon, la présence d’altérations oncogéniques qui procurent un avantage aux cellules
cancéreuses, mais qui sont souvent difficiles à cibler de manière systémique du fait
de leur rôle majeur dans le fonctionnement cellulaire normal, est exploitée afin de
les détruire sélectivement. Soixante-dix-sept molécules candidates permettant
d’induire la mort des cellules de cancer colorectal présentant des mutations de
l’oncogène RAS ont ainsi été identifiées [11]. De même, dans une étude réalisée avec 58
lignées de cancer du poumon, BRM (Brahma), un régulateur
épigénétique essentiel à la prolifération des cellules cancéreuses mutées pour
BRG1 (Brahma-related gene-1) est apparu comme
une cible thérapeutique prometteuse [1]. |
Le gliome diffus de la ligne médiane, un cancer incurable Le gliome diffus de la ligne médiane (diffuse midline glioma, DMG),
la plus sévère des tumeurs cérébrales pédiatriques, affecte majoritairement des
enfants âgés de 5 à 10 ans, qui décèdent dans un délai de deux ans après le
diagnostic [2]. Ces tumeurs
inopérables du fait de leur localisation dans le tronc cérébral et de leur caractère
diffus sont particulièrement résistantes aux traitements, comme nous l’avons montré
in vitro dans un crible chimique incluant 83 médicaments [3], et aucune chimiothérapie ne
s’est avérée efficace dans les nombreux essais cliniques réalisés. La radiothérapie
demeure le traitement de référence depuis plus de 50 ans, mais ses effets bénéfiques
ne sont que transitoires. En 2012, une mutation somatique du gène codant l’histone H3 (H3-K27M), un acteur
majeur du contrôle épigénétique de l’expression des gènes, a été identifiée comme
l’évènement initiateur du développement de la tumeur du fait de sa fréquence très
élevée (90 %) chez les patients [4, 5]. Cette
altération, jamais décrite dans d’autres cancers, entraîne la perte globale de la
triméthylation du résidu lysine en position 27 de l’histone H3 et une dérégulation
majeure de l’expression de nombreux gènes, qui produit un contexte favorable à la
transformation maligne des cellules. La substitution K27M est retrouvée
majoritairement dans les gènes codant les histones H3.1 (15 % des cas) et H3.3 (75 %
des cas) [6]. Elle pourrait
constituer une cible thérapeutique de choix dans cette maladie, mais ne peut être
directement ciblée à l’heure actuelle, d’autant que la protéine mutée est dépourvue
d’activité enzymatique propre. Ces cancers présentent peu d’anomalies génétiques
additionnelles [10]
(→) et aucune de celles trouvées de manière récurrente ne peut être
facilement ciblée dans un but thérapeutique. C’est le cas notamment des mutations de
TP53 qui code la protéine p53, du fait du rôle central que joue
cette protéine dans la réparation de l’ADN et dans le cycle cellulaire. Il est donc
primordial d’améliorer notre connaissance du mécanisme tumoral dans cette maladie et
d’utiliser des approches sans a priori pour découvrir de nouvelles
façons de traiter ce cancer encore incurable.
(→) Voir la Nouvelle de P.Y. Dietrich et V. Dutoit, , page 319 de ce
numéro
|
Identification de vulnérabilités dans les DMG Nous avons entrepris la recherche, à grande échelle, de vulnérabilités des gliomes
diffus de la ligne médiane en nous focalisant d’abord sur les 672 protéine kinases
qui font fréquemment l’objet de thérapies ciblées inhibitrices [7]. Un crible a été réalisé à l’aide
d’une banque de 7 450 lentivirus recombinants, qui s’intègrent dans le génome des
cellules hôtes et produisent différents ARN interférents (short-hairpin
ARN, shARN), permettant ainsi d’éteindre de manière irréversible les
gènes ciblés. Quatre modèles in vitro de DMG, développés dans notre
laboratoire, utilisant des cellules souches de gliomes (glioma stem
cells, GSC) dérivées de biopsies de patients ont été étudiés en
parallèle : deux comportent une mutation de H3.3 et deux autres une mutation de
H3.1, afin de prendre en compte l’hétérogénéité génétique de ces tumeurs. Deux
modèles de cellules souches neurales (neural stem cells, NSC)
normales cultivées dans des conditions identiques à celles des GSC ont été utilisés
comme témoins négatifs. Dans ces modèles, les cellules sont transduites par les
lentivirus codant les shARN de sorte qu’une cellule ne produise qu’un seul ARN
interférent. La moitié des cellules sont collectées quarante heures après la
transduction afin de vérifier, par séquençage à haut débit de l’ADN lentiviral
intégré, la représentation de l’intégralité de la collection de shARN dans la
population cellulaire. L’autre moitié des cellules transduites est maintenue en
culture pendant 22 jours, et l’ensemble des shARN synthétisés par ces cellules est
alors également caractérisé par séquençage de l’ADN lentiviral intégré. Les gènes
essentiels à la survie des DMG sont identifiés grâce aux shARN qui les ciblent, car
ceux-ci présentent une franche diminution de leur fréquence observée à 22 jours par
rapport à 40 heures post-transduction dans les cellules cancéreuses, et dans une
moindre mesure dans les cellules saines. En effet, cette déplétion du shARN à 22
jours témoigne d’un arrêt de la prolifération ou de la mort des cellules qui
l’expriment, consécutivement à l’extinction du gène cible. La robustesse de la
sélection des candidats est assurée pour partie par la redondance de la banque
permettant de se focaliser sur les gènes pour lesquels plusieurs shARN présentent le
même effet. De même, les gènes dont l’expression est vitale dans au moins trois des
quatre modèles cellulaires de DMG ont été sélectionnés afin de s’assurer que les
résultats étaient généralisables à l’ensemble des patients. Grâce à ce crible de
létalité synthétique1, nous avons identifié 41
gènes nécessaires à la survie des cellules de DMG, parmi lesquels trois font
actuellement l’objet d’une étude de l’effet de leur extinction dans des essais
cliniques (Figure 1A).
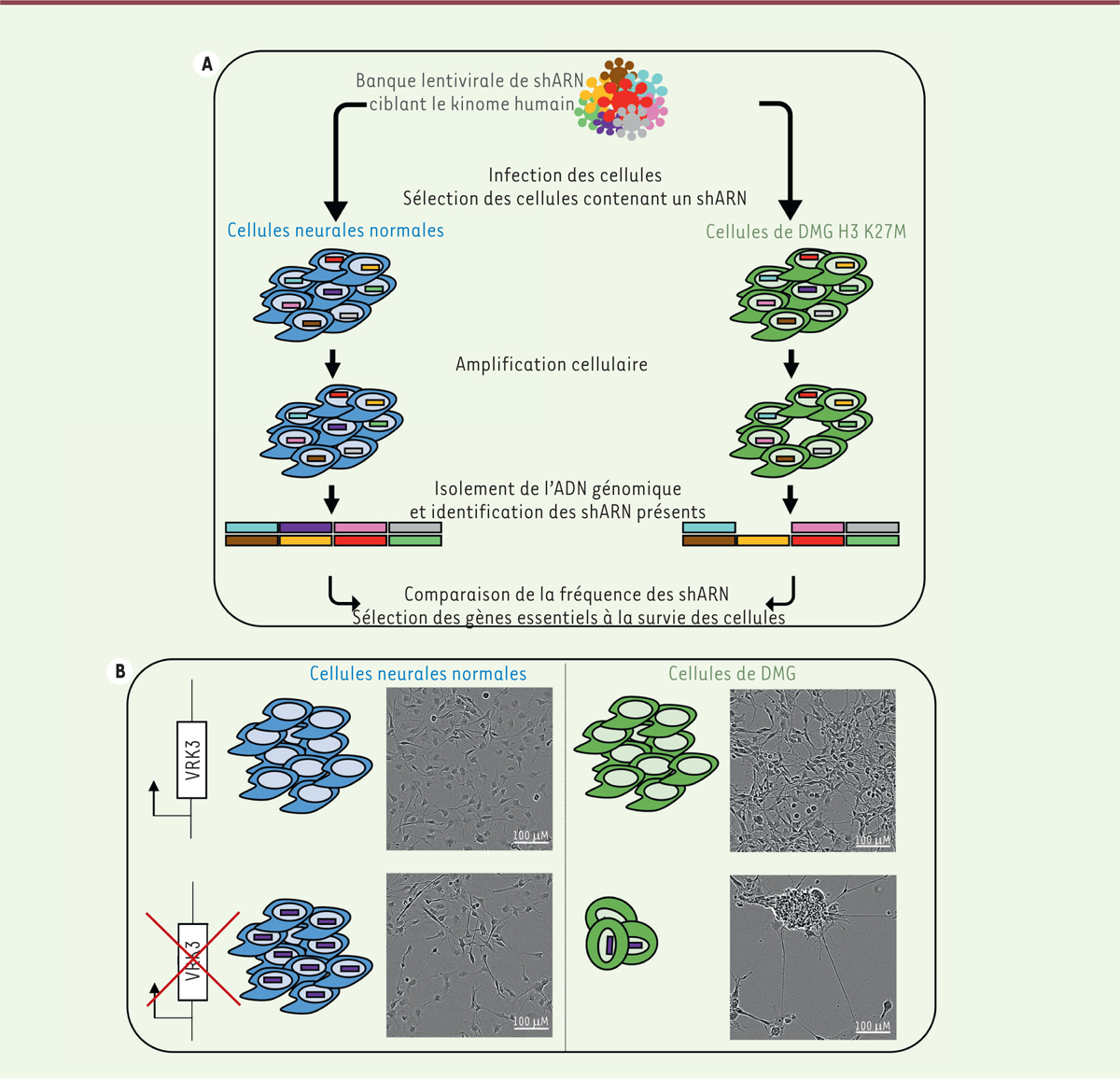
 | Figure 1. A. Stratégie du crible de létalité synthétique dans les DMG
comportant la mutation H3-K27M. Les cellules
neurales normales prises comme témoins et les cellules de DMG portant la
mutation H3-K27M sont transduites avec une banque de vecteurs
lentiviraux recombinants qui codent des shARN ciblant le kinome
(i.e., l’ensemble des kinases) humain. Les cellules
contenant un shARN sont sélectionnées, puis maintenues en culture. À 22
jours post-infection, l’ADN génomique des cellules est extrait, et les
parties codant les shARN sont amplifiées par PCR et identifiées par
séquençage à haut débit. Les shARN présentant une réduction
significative de la fréquence de leur ADN codant parmi les cellules de
DMG H3-K27M récoltées après 22 jours de culture post-infection (en
violet), mais pas parmi les cellules témoins, sont sélectionnés.
B. Effet de la répression de VRK3 sur les cellules
de DMG H3-K27M. Cinq jours après introduction
d’un shARN ciblant VRK3 (violet), on observe une
diminution significative de la prolifération des cellules de DMG,
associée à un changement de morphologie et un décollement des cellules,
phénotype caractéristique de la mort cellulaire. Ce phénotype n’est en
revanche pas observé avec les cellules neurales normales, ni avec les
cellules de DMG ne présentant pas de répression de
VRK3. |
|
VRK3, le talon d’achille des DMG Parmi les gènes candidats, VRK3, codant une sérine/thréonine kinase
de la famille des vaccinia-related kinases (VRK), a retenu notre
attention, notamment du fait des interactions de VRK3 avec plusieurs protéines
impliquées dans l’assemblage de la chromatine, dont l’accessibilité est directement
impactée par la mutation H3-K27M. Bien que ses fonctions demeurent assez mal
connues, cette protéine a été impliquée dans la progression du cycle cellulaire
(notamment dans le cancer du foie), dans la réparation de l’ADN, ou encore dans le
développement neuronal [8,
9]. Il convient de noter
qu’aucune mutation de VRK3 ou altération de son expression n’a
jusqu’ici été identifiée dans les DMG, ce qui témoigne de l’efficacité d’une
approche sans « a priori » pour dévoiler des gènes qu’une approche
« classique » n’aurait jamais identifiés comme cibles thérapeutiques
potentielles. Nous avons validé l’impact majeur de l’inhibition de VRK3 sur la
prolifération dans les quatre modèles de GSC, en utilisant 4 ARN interférents
distincts qui ont entraîné chacun une répression de ce gène (Figure 1B). Nous avons montré, en
utilisant une molécule imperméante ne pénétrant que dans les cellules mortes, que
cet effet résultait d’une augmentation de la mortalité des cellules H3.3-K27M, alors
que les cellules H3.1-K27M présentaient uniquement des altérations morphologiques
associées à un arrêt du cycle cellulaire. Ce résultat suggère que le mécanisme de
l’action anti-tumorale issue de la répression de VRK3 diffère dans
les deux sous-groupes principaux de DMG. |
Ce travail a révélé l’implication de nouvelles protéines, telles que VRK3, dans les
DMG. La mise en place de cribles de létalité synthétique peut ainsi permettre de
découvrir de nouvelles pistes thérapeutiques dans des cancers contre lesquels les
traitements classiques sont très peu efficaces. Il a été montré que la suppression
totale de la protéine VRK3 par knock-out du gène correspondant dans
des modèles murins entraîne des symptômes similaires à ceux présents dans l’autisme,
mais n’altère pas la viabilité des souris [9].
Ce résultat suggère qu’un traitement systémique ciblant VRK3 chez
les patients atteints de DMG pourrait ne pas avoir d’effet indésirable majeur sur
les tissus sains. Des études in vivo devront confirmer l’effet
bénéfique de l’extinction de VRK3 dans les modèles de xénogreffe
tumorale que nous avons développés au laboratoire. De plus, aucun inhibiteur de la
protéine VRK3 n’étant disponible à l’heure actuelle, il est logique de s’intéresser
aux ligands de cette protéine et aux conséquences moléculaires de la répression de
VRK3 dans ces tumeurs afin d’identifier de nouvelles cibles
thérapeutiques potentielles, dont la manipulation mimerait l’effet anti-tumoral de
la répression de VRK3. |
Les auteures déclarent n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
Footnotes |
1.
Hoffman
GR
,
Rahal
R
,
Buxton
F
, et al.
Functional epigenetics approach identifies BRM/SMARCA2 as a
critical synthetic lethal target in BRG1-deficient cancers .
Proc Natl Acad Sci USA.
2014; ; 111 :
:3128.–3133. 2.
Puget
S
,
Beccaria
K
,
Blauwblomme
T
, et al.
Biopsy in a series of 130 pediatric diffuse intrinsic Pontine
gliomas . Childs Nerv Syst.
2015; ; 31 :
:1773.–1780. 3.
Grasso
CS
,
Tang
Y
,
Truffaux
N
, et al.
Functionally defined therapeutic targets in diffuse intrinsic
pontine glioma . Nat Med.
2015; ; 21 :
:555.–559. 4.
Schwartzentruber
J
,
Korshunov
A
,
Liu
XY
, et al.
Driver mutations in histone H3.3 and chromatin remodelling genes
in paediatric glioblastoma . Nature.
2012; ; 482 :
:226.–231. 5.
Wu
G
,
Broniscer
A
,
McEachron
TA
, et al.
Somatic histone H3 alterations in pediatric diffuse intrinsic
pontine gliomas and non-brainstem glioblastomas . Nat
Genet.
2012; ; 44 :
:251.–253. 6.
Castel
D
,
Philippe
C
,
Calmon
R
, et al.
Histone H3F3A and HIST1H3B K27M mutations define two subgroups of
diffuse intrinsic pontine gliomas with different prognosis and
phenotypes . Acta Neuropathol.
2015; ; 130 :
:815.–827. 7.
Silva-Evangelista
C
,
Barret
E
,
Ménez
V
, et al.
A kinome-wide shRNA screen uncovers vaccinia-related kinase 3
(VRK3) as an essential gene for diffuse intrinsic pontine glioma
survival . Oncogene.
2019; ; 38 :
:6479.–6490. 8.
Lee
N
,
Kim
DK
,
Han
SH
, et al.
Comparative Interactomes of VRK1 and VRK3 with their distinct
roles in the cell cycle of liver cancer . Mol
Cells.
2017; ; 40 :
:621.–631. 9.
Kang
M-S
,
Choi
TY
,
Ryu
HG
, et al.
Autism-like behavior caused by deletion of vaccinia-related
kinase 3 is improved by TrkB stimulation . J Exp
Med.
2017; ; 214 :
:2947.–2966. 10. Dietrich
PY,
Dutoit
V. Stratégies de
vaccination thérapeutique dans le glioblastome . Med
Sci (Paris).
2020;; 36 :
:319.–22. 11.
Luo
J
,
Emanuele
MJ
,
Li
D
, et al.
A genome-wide RNAi screen identifies multiple synthetic lethal
interactions with the Ras oncogene . Cell.
2009; ; 137 :
:835.–848. |