| |
| Med Sci (Paris). 36(1): 87–89. doi: 10.1051/medsci/2019258.Méprise sur les cibles Chroniques génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université /EFS/CNRS ; CoReBio PACA,
case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09,
France. MeSH keywords: Animaux, Antinéoplasiques, Survie cellulaire, Développement de médicament, Découverte de médicament, Humains, Thérapie moléculaire ciblée, Naphtyridines, Protein-Serine-Threonine Kinases, Erreur expérimentale, Terminologie comme sujet, Études de validation comme sujet, synthèse chimique, isolation et purification, pharmacologie, usage thérapeutique, effets des médicaments et substances chimiques, normes, tendances, méthodes, antagonistes et inhibiteurs, physiologie |

Vignette (Photo © Juan Ioavanna). La recherche d’un médicament anticancéreux passe souvent par l’identification d’une
protéine indispensable à la survie ou à la prolifération des cellules tumorales, puis
par la mise au point d’un agent thérapeutique (souvent une petite molécule) capable de
bloquer la fonction de cette protéine et de déclencher ainsi l’apoptose des cellules
cancéreuses et la régression de la tumeur. Ce type de thérapie ciblée, mis en œuvre
depuis une vingtaine d’années [1], a permis par exemple la mise au point d’un inhibiteur de kinase appelé
lapatinib qui est efficace dans le cancer du sein positif pour HER2 (human
epidermal growth factor receptor 2). L’identification de la protéine cible
est essentielle dans ce processus ; or un article récent [2] montre que celle-ci est sujette à caution, et
rapporte une dizaine d’exemples de médicaments en cours de développement et d’essais
cliniques pour lesquels cette identification est fausse : l’observation de base sur
laquelle est fondé tout un processus de mise au point long et coûteux est erronée. Cette
situation pourrait expliquer en partie le taux d’échec très élevé dans ce domaine :
seuls 3,4 % des médicaments anticancéreux entrant en phase I d’essais cliniques
aboutissent à un produit approuvé et mis sur le marché [3], alors que le chiffre est supérieur à 30 % pour
les vaccins. |
Premier avertissement : la kinase MELK Dans un premier article, paru en 2017 dans la revue en libre accès
eLife [4], les mêmes auteurs s’intéressaient à la kinase MELK (maternal
embryonic leucine zipper kinase) et à un inhibiteur spécifique appelé
OTS167 alors en cours d’essais cliniques de phase II. Ils avaient constaté que,
étonnament, l’inactivation du gène correspondant dans les cellules de diverses
lignées cancéreuses n’affectait nullement leur viabilité, alors que la kinase MELK,
sur la base d’expériences d’inactivation par ARN interférent
(RNAi), était considérée comme indispensable à la croissance
cellulaire. Quant à l’inhibiteur OTS167, il tuait bien les cellules cancéreuses,
mais avec une efficacité identique, que ces cellules comportent ou non un gène
MELK fonctionnel. L’interprétation la plus probable était donc
que MELK n’était pas essentiel, et que OTS167 inactivait les cellules en
interagissant avec des protéines autres que cette kinase. En d’autres termes, tout
le travail de R&D (jusqu’à la phase II en cours !) investi sur cet inhibiteur
était fondé sur des bases fausses, sur un mécanisme d’action erroné. Bien sûr, dans
ces conditions, les chances d’aboutir à la validation de ce composé étaient très
minces. On pouvait dès lors se demander s’il s’agissait d’un exemple isolé ou, au
contraire, d’un cas fréquent, représentant une faille importante dans le dispositif
en vigueur de drug development dans la Big Pharma.
Comme nous allons le voir, le cas de MELK ne semble pas exceptionnel… |
Dix agents qui n’agissent pas sur leur cible présumée - laquelle n’est pas
indispensable à la prolifération cellulaire L’article qui fait l’objet de cette chronique, paru en septembre 2019 dans
Science Translational Medicine [2] montre que le problème décrit ci-dessus n’est pas rarissime, et même
peut-être assez fréquent. Cette publication s’est visiblement heurtée à de sérieuses
réticences (soumise début 2019, refusée puis re-soumise six mois plus tard), et
comporte de nombreux contrôles et expériences complémentaires qui sont sans doute la
marque de reviewers exigeants. En réalité, c’est un très joli
travail avec des données impeccables, dont je ne pourrai pas détailler les 18 pages
et les 257 références (sans compter les suppléments) dans une simple chronique. En
voici néanmoins les points principaux. Le choix des cibles : les auteurs ont choisi des cibles dont l’inhibition était
censée bloquer la prolifération de cellules cancéreuses et pour lesquelles un ou
deux inhibiteurs étaient en cours d’essais cliniques (dix inhibiteurs, voir
Tableau I).
Puisqu’ils étaient à la recherche de cas de fausse identification, ils ont écarté
les exemples dans lesquels l’interaction drogue/cible avait été prouvée de manière
concluante par l’existence de mutations dans la cible provoquant une résistance à
l’inhibiteur. Dans la plupart des cas, les cibles avaient été identifiées par des
expériences d’interférence ARN (RNAi).
Tableau I.
| Cible |
Agent |
Nombre d’essais cliniques |
| CASP3 |
1541B |
Préclinique |
| PAC-1 |
3 |
| HDAC6 |
Citarinostat |
5 |
| Ricolinostat |
10 |
| MAPK14 (p38α) |
Ralimetinib |
5 |
| SCIO-469 |
3 |
| PAK4 |
PF-03758309 |
1 |
| PBK (TORK) |
OTS514 |
Préclinique |
| OTS964 |
Préclinique |
| PIM1 |
SGI-1776 |
2 |
Cibles et agents anticancéreux. |
Les gènes-cible sont-ils nécessaires ? Une inactivation de chacun d’entre eux a été
effectuée par le système CRISPR-Cas9 dans des conditions particulières. D’une part,
la transfection des cellules par le vecteur portant Cas9 et l’ARN guide est
effectuée de telle manière qu’une partie seulement des cellules soit modifiée ;
d’autre part, un gène codant la protéine GFP (green fluorescent
protein) est inclus dans le vecteur. À l’arrivée, on a donc un mélange
de cellules transfectées et fluorescentes dans lesquelles le gène cible est
inactivé, et de cellules non fluorescentes dans lesquelles ce gène est intact.
L’expérience consiste alors à cultiver ce mélange de cellules et à relever
l’évolution de la fluorescence. Si la mutation introduite n’affecte pas la
croissance des cellules, la proportion de cellules fluorescentes restera constante.
Si au contraire leur viabilité est réduite, ces cellules fluorescentes vont
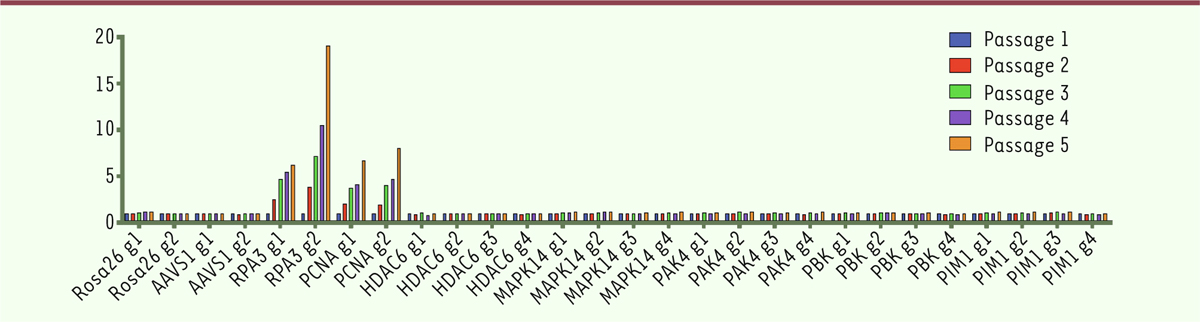
disparaitre au fur et à mesure des passages en culture. La Figure 1 montre les résultats pour
une lignée de cancer colorectal, après inactivation partielle et cinq passages en
culture. L’échelle verticale mesure le taux de réduction des cellules fluorescentes
(10 = réduction d’un facteur 10). Les gènes inactivés par CRISPR-Cas9 sont indiqués
en bas, avec chaque fois deux ou trois constructions différentes (ARN guide ciblant
différentes zones) pour le même gène. On voit qu’il n’y a aucune réduction pour les
deux témoins négatifs, Rosa26 et AAVS1 (adeno-associated
virus integration site 1), dont on sait qu’ils ne sont pas nécessaires
à la croissance. Par contre pour RPA3 (replication protein
A) et PCNA (proliferating cell nuclear antigen),
témoins positifs correspondant à des gènes essentiels, le taux de réduction des
cellules fluorescentes (mutées) augmente au fur et à mesure des passages successifs
en culture, confirmant que ces gènes sont bien nécessaires. Enfin, pour les cinq
gènes testés (HDAC6, MAPK14, PAK4, PBK, PIM1)1, aucun changement n’est visible, indiquant donc que
l’inactivation de ces gènes n’affecte pas la viabilité des cellules. Or il s’agit là
de gènes qui étaient censés être essentiels ! Ces résultats sont confirmés à de
multiples reprises avec 32 lignées différentes ; la conclusion inévitable est que
l’identification de ces cibles est erronée.
 | Figure 1.Effet de l’inactivation d’un gène cible (en abscisse) sur la viabilité
des cellules. En ordonnée, le taux de réduction des cellules KO en
coculture avec des cellules normales (voir le texte) (extrait partiel et
remanié de la figure 1 de [ 2]). |
|
Confirmations et compléments Une bonne partie de l’article est alors consacrée à la confirmation de cette
conclusion grâce à des expériences complémentaires. Des clones purs de cellules dont
les différents gènes ont été invalidés (KO) ont été obtenus dans plusieurs lignées
de cellules cancéreuses ; l’élimination de la protéine-cible est vérifiée par
western blot avec deux anticorps différents, et la croissance
en culture et sur agar est comparée avec celle des cellules-témoins non modifiées,
avec des résultats équivalents. La possibilité que l’inactivation du gène par
CRISPR-Cas9 induise la surexpression d’un homologue de ce gène a été testée et
éliminée. Une ré-analyse de criblages génome entier par CRISPR dans 485 lignées
cellulaires de cancer a confirmé les résultats pour les six gènes étudiés. Enfin,
les auteurs ont vérifié que des agents de chimiothérapie ne font pas apparaître une
sensibilité à l’inactivation de la protéine-cible (supposée). |
Deux expériences critiques, et un bel exercice de style Les gènes étudiés ici avaient été identifiés par des expériences d’interférence ARN
(RNAi) dans lesquelles un ARN complémentaire à une région de
l’ARN messager (ARNm) correspondant est introduit dans les cellules et induit une
baisse d’expression et une perte de viabilité (si le gène est essentiel). Les
auteurs ont pu se procurer quatre constructions utilisées pour ces expériences, et
ont montré que celles-ci inhibaient aussi bien la prolifération de cellules KO pour
le gène-cible que celle de cellules normales ! Il faut donc admettre que l’effet sur
la croissance des cellules passe par une interaction avec d’autres cibles que celle
que l’on a cru identifier – une insuffisance de spécificité pour la technique
RNAi qui est maintenant reconnue [5]. L’autre expérience critique a consisté à tester les dix inhibiteurs (Tableau I) sur les lignées
cellulaires KO pour les gènes correspondants. Normalement, ils ne devraient pas
avoir d’effet sur ces cellules puisque leur cible est absente – or ces lignées KO
montrent la même sensibilité que les lignées normales : il est donc patent que ces
drogues agissent par d’autres mécanismes que ceux qui sont annoncés. Un dernier point, fort intéressant mais que je ne vais pas détailler ici : les
auteurs ont aussi cherché (et réussi) à identifier la véritable cible du composé
OTS964 (censé, à tort, agir sur la molécule PBK) (Tableau I). Ils ont utilisé pour cela une approche
génétique, en isolant des mutants résistant à OTS964 et en identifiant par
séquençage le gène muté, qui s’avère être CDK11B
(cyclin-dependent kinase 11B). Cette identification a été
ensuite confirmée par toute une série d’expériences : un bel exercice de style
montrant que cette équipe est non seulement capable de démontrer que les cibles
annoncées sont erronées, mais aussi de trouver les véritables cibles par une
approche élégante et rigoureuse. |
Moralité : la rigueur expérimentale n’est pas un luxe Ces résultats sans appel sont très importants pour le domaine de l’oncologie. Ils ont
sans nul doute semé la consternation dans les états-majors d’entreprises qui avaient
misé des millions sur le développement pré-clinique (et même souvent clinique) de
molécules qui n’agissent pas sur leur cible supposée, laquelle – contrairement au
présupposé – n’est pas indispensable au développement tumoral. Ils montrent qu’une
identification sérieuse de ces cibles, notamment grâce à des données génétiques, est
un préalable incontournable à de lourds investissements, et que la caractérisation
du mécanisme d’action des candidats-médicaments est essentielle si l’on veut
diminuer le risque de coûteux échecs. Cet article dans lequel tous les artefacts
imaginables ont été envisagés et soigneusement éliminés par des expériences
impeccables est un bel exemple de la voie à suivre – je dirais même qu’il comporte
un peu d’overkill, une surabondance de contrôles qui représentent
un travail considérable et sont sans doute la trace des avis critiques dont il a dû
faire l’objet de la part d’experts dont il dérangeait les certitudes. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
Footnotes |
1.
Sharma
SV
Settleman
J
Oncogene addiction: setting the stage for molecularly targeted
cancer therapy . Genes Dev.
2007; ; 21 :
:3214.–3231. 2.
Lin
A,
Giuliano
CJ,
Palladino
A, et al.
Off-target toxicity is a common mechanism of action of cancer
drugs undergoing clinical trials . Sci Transl
Med.
2019;; 11. Pii :
eaaw8412.. 3.
Wong
CH
Siah
KW
Lo
AW
Estimation of clinical trial success rates and related
parameters . Biostatistics.
2019; ; 20 :
:273.–286. 4.
Lin
A,
Giuliano
CJ,
Sayles
NM,
Sheltzer
JM. CRISPR/Cas9
mutagenesis invalidates a putative cancer dependency targeted in on-going
clinical trials . eLife.
2017;; 6. Pii :
e24179.. 5.
W.
Putzbach, Q.
Q.
Gao,
M.
Patel, S.
et al.
Many si/shRNAs can kill cancer cells by targeting multiple
survival genes through an off-target mechanism .
eLife.
2017;; 6. Pii :
e29702.. |