Chez les eucaryotes, les gènes peuvent être « morcelés » en différents exons contenant l’information génétique qui sera présente dans les ARN messagers (ARNm) matures. Les exons sont séparés par des séquences, appelées introns, également transcrites mais éliminées lors de l’épissage qui va produire l’ARNm mature, lui-même, traduit en protéine. La transcription des gènes est réalisée dans le noyau de la cellule où l’ARN synthétisé va subir un certain nombre de modifications pour donner naissance à un ARNm fonctionnel, dit mature, qui, exporté vers le cytoplasme, sera traduit en protéine. Il est maintenant clairement établi que l’export nucléaire des ARNm et leur traduction dépendent de l’étape d’épissage. La maturation des ARNm est en effet un processus complexe couplé à la transcription des gènes, le devenir de ces ARN étant lui-même dépendant de leur maturation (Figure 1). Lors de ce processus de maturation, les ARN reconnus comme « anormaux », notamment les ARNm non épissés ou improprement épissés, sont efficacement détectés puis dégradés dans le noyau de la cellule par des processus encore mal compris à ce jour. Le complexe multi-protéique, appelé « exosome à ARN », joue un rôle essentiel dans ce processus de dégradation des ARN « anormaux » [1] (→).
(→) Voir la Synthèse de N. Leporé et D. Lafontaine, m/s n° 3, mars 2010, page 259
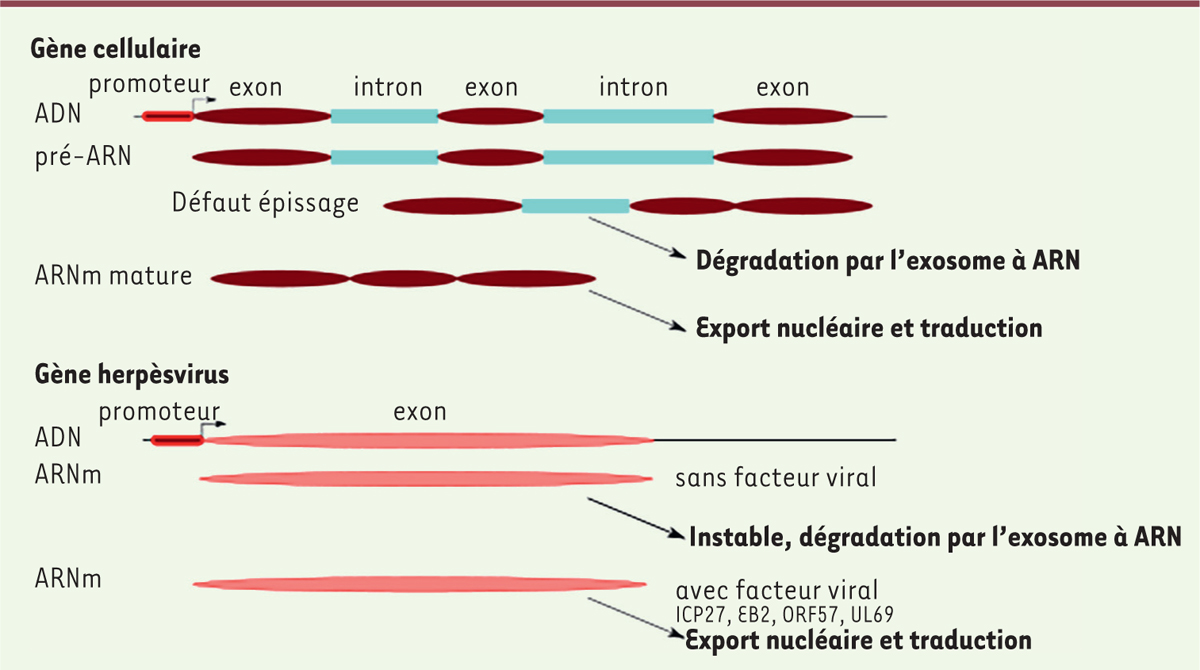
| Figure 1. Différence entre les gènes cellulaires et les gènes des virus
herpès. Une très large majorité des gènes
cellulaires est morcelée (gènes composés d’exons séparés par des introns).
Au cours de la transcription de ces gènes, les ARN sont maturés (mise en
place de la coiffe en 5’, épissage et polyadénylation). Si cette maturation
est incomplète ou incorrecte, l’ARN est dégradé dans le noyau de la cellule
par le complexe exosomal à ARN. Contrairement aux gènes cellulaires, les
gènes des herpèsvirus sont dépourvus d’intron et donc les ARN issus de ces
gènes sont normalement reconnus comme immatures et rapidement dégradés dans
le noyau. Mais les herpèsvirus codent des protéines – ICP27
[infected cell polypeptide 27] pour l’herpès simplex
virus 1 [HSV1], UL69 [homologue ICP27] pour le cytomégalovirus [CMV] ou EB2
pour l’Epstein-Barr virus [EBV] – dont le rôle est de favoriser
l’accumulation cytoplasmique des ARNm viraux. |
Si dans leur grande majorité, les gènes humains contiennent des introns, environ 3 % de ces gènes ne contiennent qu’un exon et donc aucun intron : c’est le cas, par exemple, des gènes des histones. Ces gènes cellulaires dépourvus d’intron sont pourtant correctement exprimés dans les cellules grâce à l’intervention de mécanismes spécifiques : il a en effet été montré que des composants de la machinerie d’épissage peuvent être recrutés sur ces ARN et faciliter leur export nucléaire [2].
Les virus sont connus pour leur capacité à utiliser et détourner les machineries cellulaires à leur profit. Ainsi, les virus à ADN, qui se multiplient au moins en partie dans le noyau de leur cellule hôte, ont en général une structure génomique semblable à celle des gènes cellulaires et utilisent l’ARN polymérase II cellulaire pour transcrire leurs gènes. Les herpèsvirus sont des virus à ADN très largement répandus dans le règne animal. Neuf d’entre eux infectent l’homme et sont responsables de pathologies variées. Certains peuvent également être associés au développement de cancers. Ainsi, le virus d’Epstein Barr (EBV), un herpèsvirus ubiquitaire, est l’agent causal de la mononucléose infectieuse et il est associé au développement de différents cancers affectant soit des cellules lymphoïdes (par exemple, le lymphome de Burkitt et les lymphomes post-transplantation), soit des cellules épithéliales (par exemple, le cancer du rhinopharynx et un sous-type de cancer gastrique). L’une des caractéristiques des gènes des herpèsvirus est que, contrairement aux gènes cellulaires, ils sont pour la plupart dépourvus d’intron (Figure 1). Cette absence d’intron pourrait être un handicap pour l’expression des gènes viraux dans la cellule hôte. Or, les ARN viraux sont exprimés et s’accumulent dans le cytoplasme cellulaire afin de conduire à une production virale efficace. Les travaux entrepris depuis plusieurs années sur le virus d’Epstein-Barr par notre équipe, ou sur d’autres herpèsvirus par d’autres équipes, ont permis de montrer que ces virus codent un facteur dont le rôle est de favoriser l’accumulation cytoplasmique des ARNm viraux produits à partir de gènes dépourvus d’intron. Dans le cas des virus Herpès Simplex (HSV-1 et -2), il s’agit de la protéine ICP27 (infected cell polypeptide 27) ; pour le cytomégalovirus (CMV), il s’agit de la protéine UL69 (homologue d’ICP27) ; pour le virus associé au sarcome de Kaposi (KSHV), il s’agit de la protéine ORF57 (open reading frame 57) ; et pour le virus d’Epstein-Barr (EBV), de la protéine EB2 (ou SM). Pour l’EBV, mais ceci est également vrai dans le cas des autres herpèsvirus, un virus recombinant n’exprimant pas EB2 est incapable de se multiplier. En effet, en l’absence de cette protéine, certains ARNm viraux ne s’accumulent pas dans le cytoplasme de la cellule hôte [3] et sont inefficacement traduits [4]. Les travaux initiaux entrepris pour comprendre le mode d’action de la protéine EB2 avaient révélé son rôle important dans l’export nucléaire des ARNm viraux cibles. EB2 fait en effet la navette entre le noyau et le cytoplasme des cellules et interagit avec des protéines cellulaires, comme TAP/NXF1 (nuclear RNA export factor 1) ou ALYREF (nuclear export adaptor protein), qui sont directement impliquées dans l’export nucléaire des ARNm [5,6]. Nos travaux récents montrent cependant qu’EB2 peut également être impliquée dans la stabilisation nucléaire de ses ARNm cibles [7]. En effet, en son absence, les ARN viraux sont rapidement dégradés dans le noyau de la cellule par le complexe exosomal à ARN. La présence de la protéine EB2 permet d’augmenter significativement la demi-vie de ces ARNm. La déplétion du complexe exosomal à ARN de la cellule entraîne également l’accumulation nucléaire de ces ARNm. N’étant plus dégradés, ceux-ci peuvent être exportés dans le cytoplasme de la cellule hôte. Ces résultats suggèrent que la protéine virale EB2 pourrait agir à l’encontre de l’exosome à ARN sur ces ARNm.
Afin de déterminer comment EB2 protège les ARNm viraux de la dégradation nucléaire, une recherche des partenaires cellulaires d’EB2 a été réalisée. Cette analyse a permis d’identifier la protéine SRSF3 (serine and arginine-rich splicing factor 3, également nommée SRp20), une protéine participant au spliceosome [8, 9]. Comme la déplétion du complexe exosomal à ARN, l’inhibition de l’expression de SRSF3 dans les cellules induit le même effet que la présence d’EB2, c’est-à-dire une stabilisation des ARNm viraux produits à partir de gènes dépourvus d’intron. Ces résultats ont conduit à émettre l’hypothèse d’un recrutement du complexe exosomal à ARN par la protéine SRSF3. Le complexe exosomal à ARN est recruté sur ses ARN cibles par l’un des trois complexes adaptateurs : TRAMP (Trf4p/Air2p/Mtr4p polyadenylation), PAXT [poly(A) tail exosome targeting] ou NEXT (nuclear exosome targeting) [10]. La liaison entre SRSF3 et ces composants a donc été examinée par des expériences de co-immunoprécipitation et une interaction entre SRSF3 et RBM7 (RNA-binding motif protein 7), un composant du complexe NEXT, a pu être mise en évidence, suggérant le recrutement par SRSF3, lié à l’ARNm cible, du complexe exosomal à ARN par l’intermédiaire de la protéine RBM7 du complexe NEXT. Un lien a donc été établi pour la première fois entre la protéine SRSF3 du spliceosome et le complexe exosomal à ARN. L’interaction entre EB2 et la protéine SRSF3, déjà démontrée [8], permet probablement d’inhiber le recrutement du complexe exosomal à ARN par SRSF3, favorisant la stabilisation nucléaire des ARNm viraux ainsi que leur export vers le cytoplasme où ils seront efficacement traduits en protéine (Figure 2). EB2 permet de plus d’accroître très efficacement la traduction de ses ARNm cibles en favorisant le recrutement du complexe d’initiation de la traduction [11]. Si SRSF3 recrute le complexe exosomal à ARN en l’absence d’EB2, en présence de la protéine virale, SRSF3 apparaît nécessaire à l’export nucléaire des ARNm viraux transcrits à partir de gènes dépourvus d’intron [8].
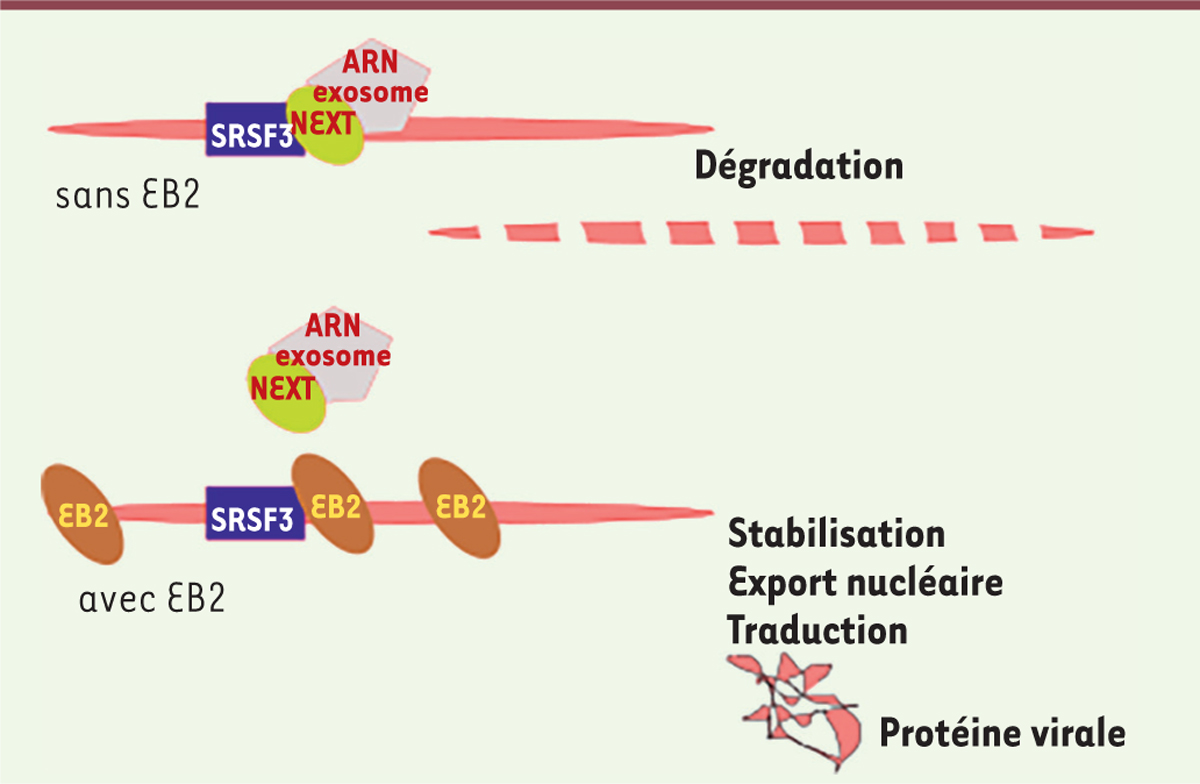
| Figure 2. Rôle de la protéine EB2 d’EBV dans la stabilité, l’export
nucléaire et la traduction des protéines virales. En
l’absence de la protéine virale EB2 (Epstein Barr protein
2), la protéine cellulaire SRSF3 (serine and arginine-rich
splicing factor 3) fixée sur l’ARN viral recrute le complexe
exosomal à ARN en interagissant avec la sous-unité RBM7 (RNA-binding
motif protein 7) du complexe adaptateur NEXT (nuclear
exosome targeting). L’ARN viral est ainsi rapidement dégradé
dans le noyau de la cellule. Par contre, en présence de la protéine virale
EB2 qui, elle-même, interagit avec SRSF3, l’ARN viral est stabilisé dans le
noyau, protégé contre la dégradation par le complexe exosomal à ARN.
L’interaction entre EB2 et SRSF3 favorise, de plus, l’export nucléaire de
l’ARN viral et sa traduction dans le cytoplasme. |
Outre la démonstration du rôle multiple et complexe de la protéine virale EB2, ces travaux ont permis de mettre en évidence une nouvelle fonction pour la protéine cellulaire SRSF3, connue pour être impliquée dans l’épissage et l’export de certains ARNm. Ainsi, SRSF3 apparaît également capable de recruter le complexe exosomal à ARN qui dégrade les ARN. Se pose alors la question de savoir comment SRSF3 est recrutée sur les ARN viraux issus de gènes dépourvus d’intron, et comment EB2 peut bloquer ce recrutement et utiliser SRSF3 pour favoriser l’export des ARNm viraux. Il a été montré précédemment que les protéines SR (une famille conservée de protéines contenant un domaine protéique riche en résidus sérine et arginine, impliquées dans l’épissage d’ARN), dont fait partie SRSF3, permettent l’export des ARNm cellulaires transcrits à partir de gènes dépourvus d’intron [12]. Si ces ARN recrutent SRSF3 comme cela a été montré, la question de leur stabilité dans la cellule où il ne semble pas exister de protéine homologue à la protéine virale EB2 reste donc à élucider.