| |
| Med Sci (Paris). 35(1): 39–45. doi: 10.1051/medsci/2018311.Génétique et origine d’Homo sapiens Jean-Pierre Henry1* 1Université Paris Diderot, Laboratoire Matière et Systèmes Complexes, CNRS UMR7057, bâtiment Condorcet, 10, rue Alice Domon et Léonie Duquet, 75013Paris, France |
Vignette (Photo © Inserm - Delphine Benarroch-Popivker).
Homo sapiens est une branche des hominidés, une famille à laquelle appartiennent les grands singes (chimpanzés, bonobos, gorilles et orangs-outans). Le processus d’hominisation a été lent et complexe, de nombreuses formes primitives ont été décrites, formant l’ensemble des hominines. La paléontologie s’efforce depuis de nombreuses années d’établir la phylogénie (l’arbre généalogique) de notre espèce. Fondée sur la découverte des fossiles et des outils associés, son approche est éminemment aléatoire. Les découvertes récentes, comme celle du crâne de Sahelanthropus [1] en 2002, considéré comme le plus ancien hominine, ou celle des restes d’H. sapiens au Maroc [2], reculant à 300 000 ou 400 000 ans l’apparition de notre espèce, montrent que nous sommes loin d’avoir une image complète de nos ancêtres. Le côté fragmentaire des restes complique évidemment les propositions de filiation. Par ailleurs, la datation est difficile. Initialement appuyée sur la stratigraphie des terrains contenant les fossiles, elle bénéficie de progrès technologiques importants : l’utilisation de différents isotopes (carbone C14, mais aussi potassium, argon, ou uranium-thorium) qui, selon leur vitesse de décroissance, permettent de sonder différentes périodes, ou d’autres méthodes, comme la thermoluminescence ou la résonance de spin électronique (ESR)1, plus spécifiques, qui sont maintenant d’usage courant. Depuis le début des années 2000, le génome humain a été séquencé. Le projet de séquençage de 1 000 génomes d’êtres humains vivant actuellement et d’origines géographiques définies, terminé en 2012, a permis d’aborder la diversité de la population humaine [3]. Chaque individu est caractérisé par son génotype, sa séquence personnelle, qui, en moyenne, diffère de 0,6 % de celle d’un autre être humain (20 millions de nucléotides différents sur les 3 milliards que compte le génome entier). Les génomes du chimpanzé [4], du bonobo [5] et du gorille [6] ont également été décryptés. Plus récemment, celui d’hominines disparus, comme Homo neanderthalis, a été séquencé à partir d’ossements datés du Pléistocène2 tardif (50 000 ans) [7]. Les progrès tout à fait spectaculaires réalisés dans le traitement des ADN anciens a entrainé la multiplication des analyses par de nombreux laboratoires et, depuis le début de 2018, plusieurs centaines de génomes anciens ont été séquencés. À l’automne 2017, le laboratoire de génétique de l’évolution de Svante Pääbo, à Leipzig, annonçait les résultats d’un séquençage à partir de sédiments, sans ossements fossiles visibles, présents dans les grottes [8] ! Quels apports cette nouvelle science, la paléogénomique, apporte-t-elle à la connaissance de l’origine d’H. sapiens ? Rechercher l’origine des traits qui différencient l’homme du chimpanzé est un premier objectif et, a priori, la comparaison des génomes est une première direction de recherche. Un second objectif est dynamique : suivre l’évolution des espèces à travers l’étude de la diversité des génomes. Cette seconde approche, s’appuyant sur les progrès de la génomique, permet d’accéder à la généalogie d’H. sapiens. Dans cette revue, nous aborderons ces deux voies de recherche et nous esquisserons une description des méthodes utilisées. |
Comparaison des séquences d’hominidés Les différences morphologiques et physiologiques entre H. sapiens et le chimpanzé sont bien décrites. La différence est frappante au niveau du cerveau, trois fois plus volumineux chez l’homme. Peut-on associer des différences de séquences d’ADN à cette observation ? Entre l’homme et le chimpanzé, les séquences diffèrent de 1,3 à 4 % selon les méthodes et données utilisées pour effectuer la mesure. En termes d’acides aminés, la divergence est inférieure à 1 % (les séquences codant des protéines représentent moins de 2 % de l’ADN humain) [6]. La mise en évidence d’une différence fonctionnelle associée à des différences génomiques est un exercice difficile et aléatoire, pratiqué en ciblant a priori des gènes candidats. Le gène FOXP2, qui code la protéine FoxP2 (Forkhead-box P2), est un exemple de cette approche. Lorsqu’il est muté, il produit chez l’homme des troubles sévères de la parole, suggérant son implication dans le développement du langage [9]. Quelles sont les différences dans la séquence de ce gène entre l’homme et le chimpanzé ? Bien que ce gène soit très conservé chez les mammifères, deux mutations non conservatives (entraînant une modification fonctionnelle de la protéine FoxP2) distinguent homme et chimpanzé. L’effet de ces mutations a été étudié en introduisant la forme humaine du gène chez la souris (qui possède la forme retrouvée chez le chimpanzé) [10]. Cette « humanisation de FoxP2 » n’a pas permis aux souris de parler… mais elle a montré que ce gène jouait un rôle important dans le développement du système nerveux. FoxP2 est en fait un facteur de transcription contrôlant l’expression de différents gènes pouvant former des réseaux fonctionnels. L’analyse du gène FOXP2 d’H. neanderthalis a montré la présence des deux mutations caractéristiques d’H. sapiens. L’homme de Néanderthal parlait-il ? La question reste ouverte, d’autant que la séquence de FOXP2 de ce dernier possède une mutation absente chez H. sapiens, au niveau d’une séquence intronique codant un autre facteur de transcription [11]. Cet exemple montre la difficulté de l’approche, l’évolution étant souvent liée à une modification de structure fine d’un gène régulateur. Cette remarque est importante pour comprendre l’évolution : la chenille et le papillon ont strictement le même génome et ce sont les gènes régulateurs qui sont responsables de la différence de leur forme, leur expression à des temps ou des localisations différents produisant ces variations. Le gène ASPM (abnormal spindle-like microcephaly associated), impliqué dans le développement du cerveau, offre un autre exemple [12]. Chez l’homme, sa mutation entraîne une microcéphalie primaire. Ce gène code une protéine qui s’exprime dans le cerveau au moment de la prolifération des neuroblastes, les précurseurs des neurones. Il est présent chez tous les mammifères, et sa phylogénie a été établie chez six espèces de singes et comparée à celle d’H. sapiens. Les différences que l’on observe au niveau de ce gène indiquent-elles une évolution favorisant la taille du cerveau ? D’une manière générale, un changement de nucléotide dans une séquence codant une protéine peut avoir un effet sur sa fonctionnalité (mutation non synonyme), en l’altérant ou en l’améliorant. Ce changement sera alors soumis à la sélection : un effet délétère sera éliminé par une sélection négative, tandis qu’une solution positive favorisera l’extension de ce nouveau génotype à l’ensemble de la population. Alternativement, le changement d’un nucléotide peut ne pas changer la séquence protéique : un acide aminé est, en effet, codé par trois nucléotides (triplets) et plusieurs triplets codent le même acide aminé. On parle alors de mutation synonyme et, en l’absence d’effet sur la séquence protéique, la sélection n’aura pas de prise sur la mutation. Ainsi, sur ce principe, en suivant les changements du rapport des mutations non synonymes aux mutations synonymes à travers les différentes branches de la phylogénie des singes, le gène ASPM est apparu comme étant constamment sous une pression de sélection positive dans la branche des primates conduisant à H. sapiens. Ce résultat suggère que l’évolution s’est effectuée par de multiples petits sauts, difficiles à mettre en évidence. Peut-on envisager une recherche systématique des gènes impliqués dans l’hominisation à partir de la comparaison des génomes ? Lorsqu’on recherche les positions de l’ADN où le même nucléotide est présent dans tous les génotypes de sapiens répertoriés dans le projet des 1 000 génomes mais absent chez le chimpanzé et les hommes archaïques, on constate que cette condition est remplie dans un nombre relativement limité de positions : 31 389 (auxquelles il faut ajouter des courtes délétions ou insertions). Ce chiffre correspond à la modification de 87 protéines et peut-être à l’expression de 3 000 autres. Un premier examen indique que certains des gènes correspondants sont impliqués dans la prolifération des neurones du cerveau, ouvrant un champ de recherche intéressant [13]. |
L’apport de la génomique à la généalogie des hominidés La mise en évidence des gènes sous pression de sélection positive est un moyen d’accéder aux mécanismes qui ont façonné l’homme moderne. Toutefois, la nécessité d’examiner les génomes dans leur totalité pour avoir une vue plus intégrée, requiert des approches différentes, plus mathématiques. L’évolution s’intéresse à la génétique des populations : comment des évènements aléatoires isolés (des mutations), formant de nouveaux génotypes, envahissent-ils une population ? Les deux mécanismes retenus sont la dérive génique, fruit du hasard, importante sur de petites populations, et la sélection. Confrontée à un ensemble de génotypes dérivant d’un même ancêtre, la génomique s’efforcera d’établir la phylogénie, de rechercher les ancêtres, d’établir l’âge de l’ancêtre commun et de proposer la taille de la communauté de cet ancêtre. La recherche se fait en considérant le maximum de gènes, parfois l’ADN mitochondrial, mais souvent le génome le plus complet possible. Les données de séquence du Consortium des 1 000 génomes constituent une base de travail intéressante : les 1 000 séquences déterminées sont réparties en 14 groupes définis géographiquement. Comment s’exprime l’hétérogénéité de ces séquences ? La plus simple variation de séquence est le remplacement d’un nucléotide par un autre, créant ainsi un polymorphisme nucléotidique (ou SNP pour single nucleotide polymorphism). Pour les 1 000 génomes analysés, 38 millions de SNP ont été détectés, avec des fréquences variables [3]. Ce chiffre peut paraître énorme, mais cela signifie qu’environ une position sur cent est susceptible de variations. Ces polymorphismes varient entre les 14 populations du projet des 1 000 génomes et l’application de méthodes mathématiques a permis de tracer un arbre généalogique reliant ces populations [14]. Le résultat est net : la population ancestrale à l’origine du peuplement du monde est indubitablement africaine ; elle était composée d’un nombre limité d’individus (20 000) vivant il y a 200 000 ans. La comparaison du polymorphisme des populations africaines et hors d’Afrique produit un autre résultat original : la diversité génétique des populations diminue linéairement en fonction de la distance géographique avec Addis-Abeba, la capitale de l’Éthiopie [14]. Pour expliquer ce résultat surprenant, un modèle propose qu’une fraction des Africains a quitté ce continent en emportant qu’une fraction des génotypes ancestraux (Figure 1). La taille réduite de ces groupes a favorisé la dérive génique due au hasard, un effet connu sous le nom d’effet fondateur. Se fondant sur différentes hypothèses, les chercheurs ont pu mettre des chiffres sur ce modèle [15] : la population sortant d’Afrique aurait été composée de 2 000 individus et cette migration aurait eu lieu il y a 50 000 ans. Des petits groupes (quelques dizaines de personnes) se seraient alors déplacés jusqu’à couvrir l’ensemble des continents (Figure 2) ; les migrations vers l’Europe se seraient ensuite séparées des voies asiatiques il y a 23 000 ans. Il a été proposé que cette expansion se soit réalisée par de multiples colonisations, peut-être 180, chacune durant 280 ans [16], le groupe sortant perdant de sa diversité génique par effet fondateur, expliquant ainsi la relation directe entre le génome et le trajet effectué.
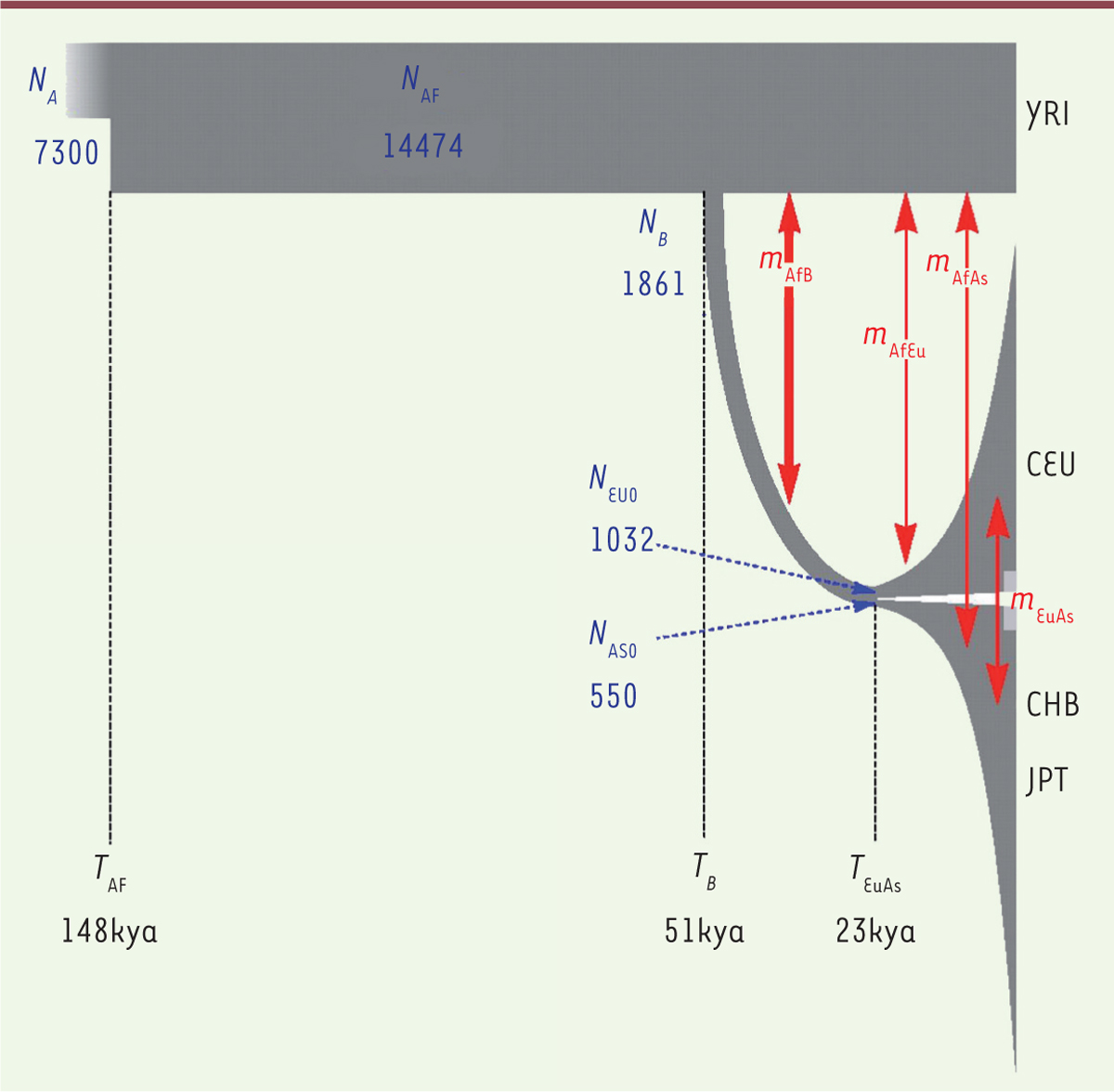
 | Figure 1.Modèle de la grande expansion dérivé de la génétique. Ce modèle a été déterminé à partir des données sur la diversité génique révélée par le projet du séquençage des 1 000 génomes. Le temps s’écoule vers la droite (kya : milliers d’années) ; la largeur des bandes grises indique la taille des populations ( N : nombre d’individus ; YRI : Africains ; CEU : Européens ; CHB : Chinois ; JPT : Japonais). La sortie d’Afrique est datée à 51 000 avant le temps présent, et la séparation entre Européens et Asiatiques à 23 000 ans. T B, T EuAs indiquent les dates de la sortie d’Afrique et de la séparation vers l’Europe et vers l’Asie ; l’épaisseur des flèches rouges indique les intensités relatives des migrations. m AfB : migration hors d’Afrique ; m AfEu : migration vers l’Europe ; m AfAs : vers l’Asie ; m EuAs : entre l’Europe et l’Asie. T AF : date de la population africaine ancestrale (d’après [ 15]). |
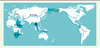 | Figure 2.
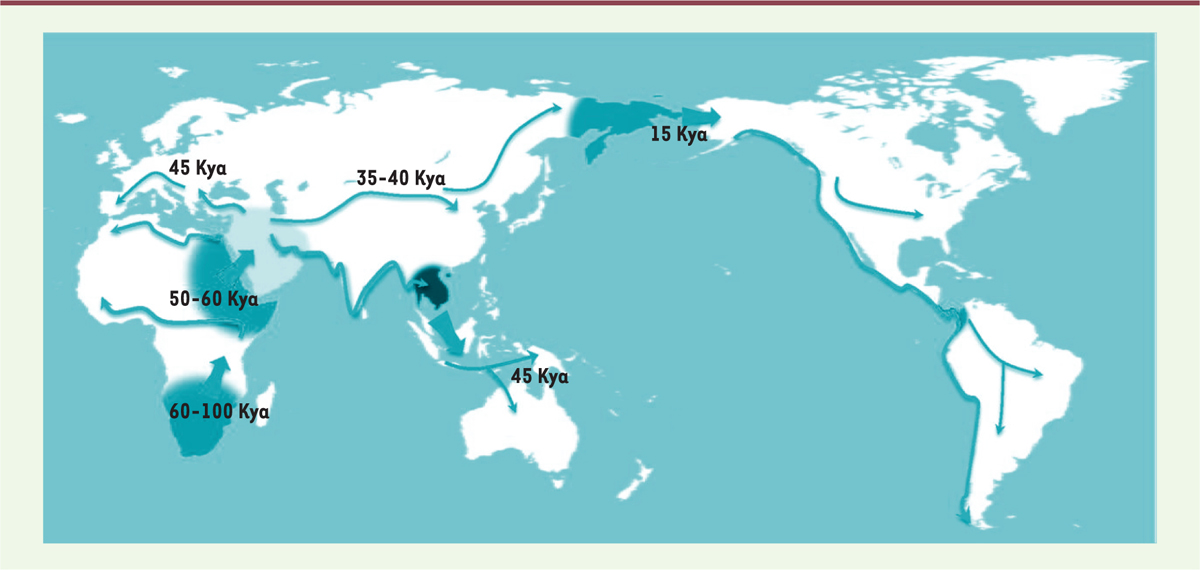
Schéma possible de la grande expansion. La carte montre la sortie d’Afrique (50-60 000 ans), la séparation des voies vers l’Europe et vers l’Asie, le peuplement de l’Australie et de l’Amérique, en supposant des conditions géographiques permettant le franchissement du détroit de Behring. Les flèches larges indiquent des « effets fondateurs », la diminution de la diversité génique par scission d’une sous-population. Kya : milliers d’années (d’après [ 14]). |
Quelle confiance peut-on avoir dans ce modèle ? Il utilise un certain nombre de paramètres qu’il est nécessaire de discuter. La généalogie se calcule en nombre de générations et non en années ; la datation suppose aussi un taux de mutations de l’ADN constant avec le temps et constant sur l’ensemble du génome. C’est donc avec une certaine réserve qu’il faut considérer les résultats numériques. Par ailleurs, ces données doivent s’intégrer avec l’ensemble des résultats paléontologiques, et certains désaccords, sans permettre de rejeter l’hypothèse, doivent être discutés [17–20]. Ainsi, des fossiles trouvés au Maroc repousseraient l’âge d’apparition d’H. sapiens à 300 000-400 000 ans [2]. Des ossements et des traces archéologiques datant d’avant la grande expansion d’Afrique ont été mis à jour hors de ce continent. En Israël, la présence d’H. sapiens sur les sites de Skhul et Qafzeh a été datée entre 90 000 et 120 000 ans [19] et une présence ancienne a été suggérée dans des sites asiatiques. Y-a-t-il eu des sorties d’Afrique d’H. sapiens avant 50 000 ans ? Ces populations sont-elles vraiment constituées d’hommes modernes ? Quels ont été leurs rapports avec les sapiens de la grande expansion venue d’Afrique ? De même que l’emploi des 1 000 génomes a permis des avancées dans la compréhension de l’histoire d’H. sapiens, les génomes des grands singes ont été analysés afin de définir la généalogie des Hominidés. Selon les paramètres utilisés, la séparation de l’homme et du chimpanzé se situerait entre 5 et 10 millions d’années [6]. Plus productive a été l’utilisation des génomes des formes humaines archaïques comme l’homme de Néanderthal. Ce génome a été séquencé une première fois en 2010 par Svante Pääbo, à partir d’ossements trouvés en Croatie et datés de 38 000 ans [21]. La séquence a ensuite été complétée, en 2014 [22], à partir d’une phalange d’un orteil découverte dans la grotte de Denisova dans les Monts Altaï (en Sibérie), puis, de nouveau, sur des ossements croates de 50 000 ans [7]. La phalange de doigt a fourni une séquence d’ADN différente de celles d’H. sapiens et d’H. neanderthalis, permettant de conclure à l’existence d’une autre forme archaïque, H. denisova, dont on ne connaît actuellement aucun autre ossement [23, 24] ! La qualité des séquences obtenues à partir de la phalange a permis une exploitation des résultats comparable à celle dérivée du projet des 1 000 génomes [22]. Une première donnée issue de cette étude est la datation du dernier ancêtre commun aux hommes archaïques et à H. sapiens, évaluée à environ 800 000 ans. Cette date n’est pas celle de la séparation des espèces : c’est en effet entre 550 000 et 765 000 ans que les hommes modernes se seraient séparés des Néanderthaliens et des Dénisoviens. Quant à la séparation de ces deux formes archaïques, elle aurait eu lieu il y a environ 400 000 ans. Ces populations étaient constituées de peu d’individus et une analyse a conclu à un cas d’endogamie3. Leurs génomes indiquaient une très faible hétérozygotie (peu de différence entre les génomes maternel et paternel). Pour rechercher la proximité des populations actuelles avec les néanderthaliens, des génotypes bien caractérisés géographiquement ont été comparés au génome archaïque. Un résultat de ces analyses est l’absence d’éléments archaïques dans les génomes africains, alors que chez les non-africains modernes, on retrouve entre 1 et 3 % d’éléments néanderthaliens. Le même type d’analyse conduit à proposer que des éléments du génome de Denisova sont présents chez les populations actuelles d’Océanie, atteignant 6 % chez les Papous. Comment ces séquences archaïques sont-elles parvenues dans les génomes d’H. sapiens ? Les analyses génomiques concluent à l’existence de croisements entre les différentes espèces [22]. Il est possible de repérer les segments provenant de l’hybridation : ils se répartissent sur l’ensemble du génome moderne et, mis bout à bout, représentent 20 % du génome de Néanderthal [25, 36] (→). (→) Voir la Chronique génomique de B. Jordan, m/s n° 12, décembre 2012, page 1129 À quelle époque ces croisements se sont-ils produits ? Néanderthal semble descendre de préhominiens, peut-être H. erectus, très tôt sortis d’Afrique. On ne trouve, en effet, pas de traces archéologiques de Néanderthaliens en Afrique. Les principaux sites où leur présence est révélée sont en Eurasie, de l’Espagne à la Sibérie. D’après les recherches archéologiques, Néanderthal disparaît il y a environ 40 000 ans. La coexistence des deux espèces n’aurait donc duré que quelques dizaines de milliers d’années [26]. Le séquençage de l’ADN d’un H. sapiens trouvé en Roumanie et âgé de 37 000 à 42 000 ans, indique une proportion élevée d’ADN néanderthalien (6 à 9 %), provenant d’un ancêtre qui se situerait à 4 ou 6 générations en amont [27]. Cette observation est compatible avec l’hypothèse d’une sortie d’Afrique d’H. sapiens qui daterait de 50 000 à 60 000 ans. D’autres travaux, fondés sur la longueur des segments d’ADN archaïques, proposent néanmoins que les croisements se seraient déroulés il y a entre 37 000 et 86 000 ans [28], ce qui suppose des mécanismes plus complexes impliquant vraisemblablement la participation de formes précoces de sapiens sorties d’Afrique avant la grande expansion, ou des croisements avec des hommes archaïques disparus. |
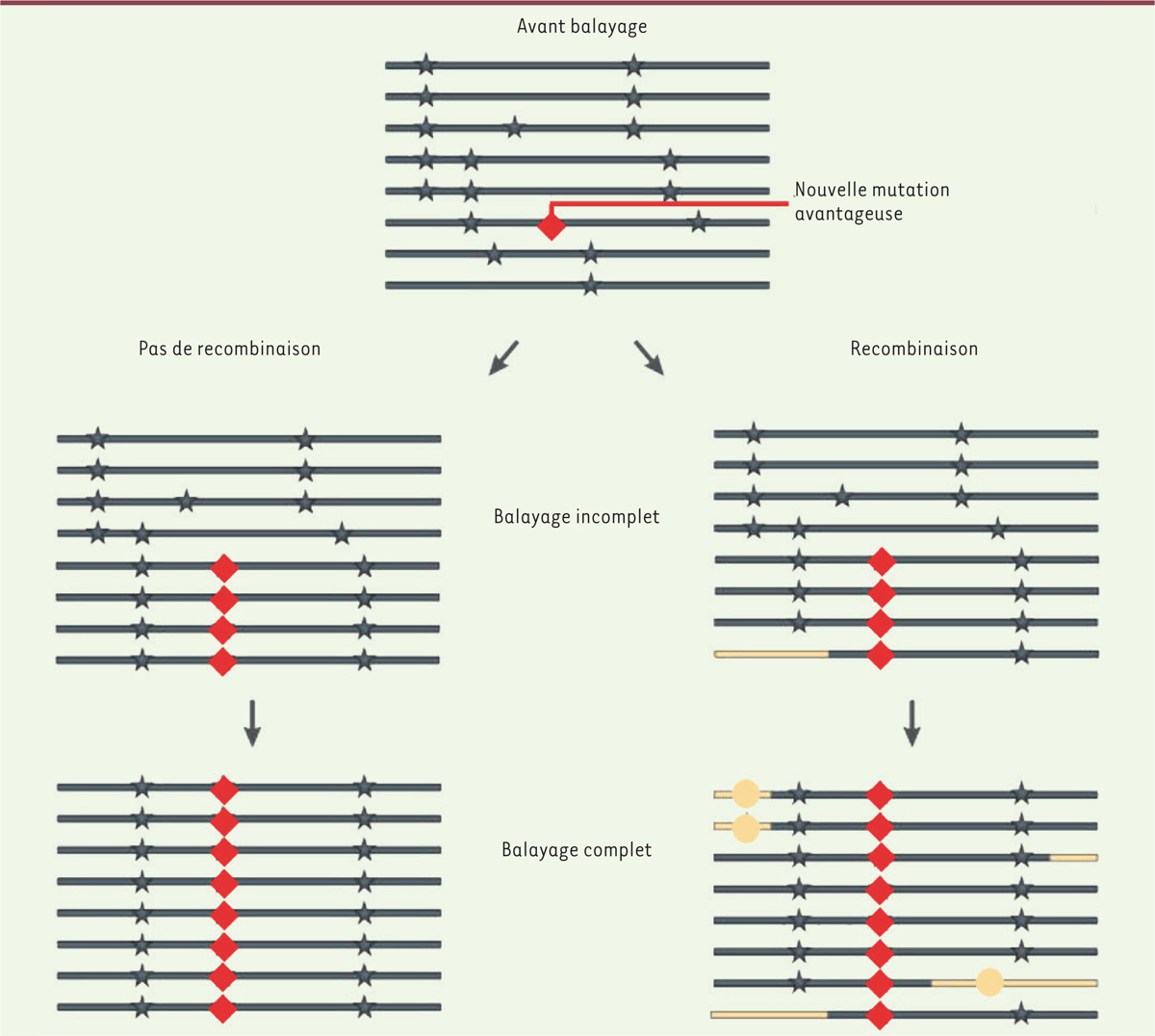
Comment l’évolution façonne le génome humain Nous avons vu comment la comparaison des génomes de la famille des hominidés permet de révéler des différences pouvant avoir une importance potentielle dans l’hominisation. Une approche différente propose de rechercher des séquences qui seraient soumises à une pression de sélection positive, marquant une évolution vers le génome d’H. sapiens moderne. La méthode la plus utilisée pour cette approche s’appuie sur le balayage sélectif (Figure 3), qui caractérise le polymorphisme nucléotidique (SNP) au voisinage d’une mutation à effet positif [29]. L’évolution sélectionnera cette mutation qui envahira alors la population des génotypes. Initialement, les positions voisines de cette mutation seront sélectionnées passivement, le segment d’ADN correspondant sera suffisamment long pour être détecté par un balayage recherchant les SNP conservés. Au cours du temps, ce segment contenant les SNP conservés se raccourcira sous l’influence des recombinaisons qui modèlent l’ADN lors de la méiose, avec pour conséquence que cette méthode détectera surtout les mutations récentes soumises à pression positive.
 | Figure 3.
Mise en évidence d’une sélection positive par l’étude du balayage sélectif. La figure montre une population de génotypes dans laquelle une mutation avantageuse s’est introduite, repérée par un losange rouge (les étoiles représentent des polymorphismes nucléotidiques, SNP). À gauche, en l’absence de toute recombinaison, la mutation va envahir la population et, avec elle, les segments voisins : on observe une perte de variabilité autour du gène sélectionné. De manière plus réaliste (à droite), la recombinaison et de nouvelles mutations vont introduire de nouvelles hétérogénéités et la détection deviendra plus difficile. Les segments jaunes résultent de phénomènes de recombinaison (d’après [ 29]). |
Cette approche a été appliquée aux génomes issus du projet des 1 000 génomes et a révélé un nombre raisonnable de gènes impliqués. Après sa sortie d’Afrique, la sélection a permis à H. sapiens de s’adapter aux nouveaux environnements qu’il a rencontrés (avec la mise en place de défenses immunitaires contre les maladies infectieuses, les modifications de couleurs de la peau et le développement des phanères) et aux changements du mode de vie (adaptation du métabolisme aux nutriments nouveaux). Deux exemples illustrent cette recherche. Le premier décrit la distribution géographique des individus capables de digérer le lait [30]. La lactase est nécessaire pour hydrolyser le lactose, présent dans le lait, en galactose et glucose, assimilables. Or cette protéine, présente chez l’enfant, disparaît chez de nombreux adultes. Plusieurs mutations permettent néanmoins sa persistance chez l’adulte, comme la mutation 13910C>T (cytosine 13910 remplacée par une thymine) : absente chez les Africains et les Asiatiques, elle est portée par 77 % des Européens (Suédois 82 %, Français 43 %, Italiens 6 %). Un examen des génomes possédant cet haplotype montre une conservation des SNP sur plus d’un million de bases autour du gène codant la lactase, indiquant une sélection récente. La date est estimée à 5 000 à 10 000 ans, une date qui correspond au développement de l’élevage d’animaux produisant le lait. D’autres mutations dans la même région de l’ADN ont conduit au même résultat, indiquant une sélection forte par différentes voies : on parle d’évolution convergente. Un autre exemple concerne l’adaptation des Tibétains à l’altitude. Habitant sur des plateaux situés à 4 000 mètres d’altitude, ces populations vivent dans une atmosphère appauvrie en oxygène. La comparaison de leurs génomes à ceux des Han chinois, l’ethnie majoritaire en Chine qui constitue 92 % de la population, indique une sélection forte d’un gène codant un facteur de transcription, EPAS1 (endothelial PAS domain protein 1), dont l’expression module le nombre d’hématies, ce qui permet une réponse à l’hypoxie engendrée par l’altitude [31]. L’adaptation observée chez les Tibétains est plus fine qu’une simple surexpression du gène EPAS1 à l’origine d’une augmentation du nombre des hématies et, par là même, une plus grande viscosité du sang, ce qui peut être dommageable, particulièrement lors de la grossesse (éclampsie) : le génotype tibétain évite ces accidents. Il serait, de manière surprenante mais bien documentée, un héritage denisovien [32] ! Les vestiges d’ADN archaïques (1 à 3 % pour Néanderthal) provenant de croisements anciens pourraient donc avoir subsisté dans le génome de sapiens car ils offrent à des individus sortant d’Afrique et plongés dans des environnements nouveaux des solutions expérimentées depuis longtemps. L’intégration de ces nouveaux gènes aurait été accélérée par les croisements entre espèces. D’autres exemples d’héritage avantageux de gènes ont été décrits, en particulier pour des gènes impliqués dans les réponses du système immunitaire inné, peu spécifique, qui est la première ligne de défense [33, 37] (→) et du système immunitaire acquis, qui est dirigé très finement vers les agresseurs [34]. (→) Voir la Synthèse de M. Deschamps et L. Quintana-Murci, m/s n° 12, décembre 2016, page 1079 Il faut néanmoins noter que l’environnement moderne est très différent de celui des hommes archaïques et que certains gènes sélectionnés à partir des croisements, afin d’améliorer les réponses de nos ancêtres à leur environnement, sont désormais associés à des pathologies, notamment chroniques [35]. En conclusion, peut-on penser que la paléogénomique apporte les nombreuses réponses que l’on attend sur l’origine et l’évolution d’H. sapiens ? L’avenir le dira, mais elle représente désormais une branche active de la recherche qui doit s’intégrer dans un ensemble de travaux composant la paléoanthropologie. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Je tiens à remercier la direction et l’ensemble du personnel du laboratoire pour la qualité de leur accueil.
|
Footnotes |
1.
Brunet M, Guy F, Pilbeam D, et al. A new hominid from the upper Miocene of Chad . Central Africa. Nature. 2002; ; 418 ::145.–151. 2.
Hublin JJ, Ben-Ncer A, Bailey SE, et al. New fossils from Jebel Irhoud, Morocco and the pan-African origin of Homo sapiens . Nature. 2017; ; 546 ::289.. 3. The Genomes Project C . An integrated map of genetic variation from 1,092 human genomes . Nature. 2012; ; 491 ::56.. 4.
The Chimpanzee S, Analysis C . Initial sequence of the chimpanzee genome and comparison with the human genome . Nature. 2005;; 437 ::69..
5.
Prüfer K, Munch K, Hellmann I, et al. The bonobo genome compared with the chimpanzee and human genomes . Nature. 2012; ; 486 ::527.. 6.
Scally A, Dutheil JY, Hillier LW, et al. Insights into hominid evolution from the gorilla genome sequence . Nature. 2012; ; 483 ::169.. 7.
Prüfer K, de Filippo C, Grote S, et al. A high-coverage Neandertal genome from Vindija cave in Croatia . Science. 2017; ; 358 ::655.–658. 8.
Slon V, Hopfe C, Weiß CL, et al. Neandertal and Denisovan DNA from Pleistocene sediments . Science. 2017; ; 356 ::605.–608. 9.
Enard W, Przeworski M, Fisher SE, et al. Molecular evolution of FOXP2, a gene involved in speech and language . Nature. 2002; ; 418 ::869.–872. 10.
Enard W, Gehre S, Hammerschmidt K, et al. A humanized version of Foxp2 affects cortico-basal ganglia circuits in mice . Cell. 2009; ; 137 ::961.–971. 11.
Maricic T, Günther V, Georgiev O, et al. A recent evolutionary change affects a regulatory element in the human FOXP2 gene . Mol Biol Evol. 2013; ; 30 ::844.–852. 12.
Evans PD, Anderson JR, Vallender EJ, et al. Adaptive evolution of ASPM, a major determinant of cerebral cortical size in humans . Hum Mol Genet. 2004; ; 13 ::489.–494. 13.
Pääbo S.. The human condition: a molecular approach . Cell. 2014; ; 157 ::216.–226. 14.
Li JZ, Absher DM, Tang H, et al. Worldwide human relationships inferred from genome-wide patterns of variation . Science. 2008; ; 319 ::1100.–1104. 15.
Gravel S, Henn BM, Gutenkunst RN, et al. Demographic history and rare allele sharing among human populations . Proc Natl Acad Sci USA. 2011; ; 108 ::11983.–11988. 16.
Henn BM, Cavalli-Sforza LL, Feldman MW. The great human expansion . Proc Natl Acad Sci USA. 2012; ; 109 ::17758.–17764. 17.
Stringer C.. The origin and evolution of Homo sapiens . Philos Trans R Soc Lond B Biol Sci. 2016; ; 371 : 18.
Stringer C, Galway-Witham J. On the origin of our species . Nature. 2017; ; 546 ::212.. 19.
Hershkovitz I, Weber GW, Quam R, et al. The earliest modern humans outside Africa . Science. 2018; ; 359 ::456.–459. 20.
Bae CJ, Douka K, Petraglia MD. On the origin of modern humans: Asian perspectives . Science. 2017 ; :358.. 21.
Green RE, Krause J, Briggs AW, et al. A draft sequence of the neandertal genome . Science. 2010; ; 328 ::710.–722. 22.
Prufer K, Racimo F, Patterson N, et al. The complete genome sequence of a Neanderthal from the Altai mountains . Nature. 2014; ; 505 ::43.–49. 23.
Meyer M, Kircher M, Gansauge MT, et al. A high-coverage genome sequence from an archaic denisovan individual . Science. 2012; ; 338 ::222.–226. 24.
Reich D, Green RE, Kircher M, et al. Genetic history of an archaic hominin group from Denisova cave in Siberia . Nature. 2010; ; 468 ::1053.–1060. 25.
Vernot B, Akey JM. Resurrecting surviving neandertal lineages from modern human genomes . Science. 2014; ; 343 ::1017.–1021. 26.
Slatkin M, Racimo F. Ancient DNA and human history . Proc Natl Acad Sci USA. 2016; ; 113 ::6380.–6387. 27.
Fu Q, Hajdinjak M, Moldovan OT, et al. An early modern human from Romania with a recent Neanderthal ancestor . Nature. 2015; ; 524 ::216.. 28.
Sankararaman S, Patterson N, Li H, et al. The date of interbreeding between neandertals and modern humans . PLoS Genet. 2012; ; 8 ::e1002947.. 29.
Nielsen R, Hellmann I, Hubisz M, et al. Recent and ongoing selection in the human genome . Nat Rev Genet. 2007; ; 8 ::857.. 30.
Bersaglieri T, Sabeti PC, Patterson N, et al. Genetic signatures of strong recent positive selection at the lactase gene . Am J Hum Genet. 2004; ; 74 ::1111.–1120. 31.
Yi X, Liang Y, Huerta-Sanchez E, et al. Sequencing of 50 human exomes reveals adaptation to high altitude . Science. 2010; ; 329 ::75.–78. 32.
Huerta-Sánchez E, Asan Jin X, et al. Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA . Nature. 2014; ; 512 ::194.. 33.
Dannemann M, Andrés Aida M, Kelso J. Introgression of Neandertal- and Denisovan-like haplotypes contributes to adaptive variation in human toll-like receptors . Am J Hum Genet. 2016; ; 98 ::22.–33. 34.
Abi-Rached L, Jobin MJ, Kulkarni S, et al. The shaping of modern human immune systems by multiregional admixture with archaic humans . Science. 2011; ; 334 ::89.–94. 35.
Simonti CN, Vernot B, Bastarache L, et al. The phenotypic legacy of admixture between modern humans and Neandertals . Science. 2016; ; 351 ::737.–741. 36.
Jordan B.. Néandertal et Homo sapiens: to meet, or not to meet? . Med Sci (Paris). 2012; ; 28 ::1129.–1132. 37.
Deschamps M, Quintana-Murci L. Immunité innée et maladies chez l’homme . Med Sci (Paris). 2016; ; 32 ::1079.–1086. |