| |
| Med Sci (Paris). 2009 August; 25(8-9): 761–762. Published online 2009 August 15. doi: 10.1051/medsci/2009258-9761.Les génomes des schistosomes : une étape clé dans la lutte contre la bilharziose Raymond J. Pierce,1,2,3* Guillaume Mitta,4 and Emmanuel Roger1,2,3 1Inserm U 547, Lille, France 2Université Lille Nord de France, F-59000 Lille, France 3Institut Pasteur de Lille, IFR 142, 1, rue du Professeur A. Calmette, 59019 Lille, France 4UMR 5244, CNRS EPHE, Université de Perpignan, équipe parasitologie fonctionnelle et évolutive, 52, avenue Paul Alduy, 66860 Perpignan Cedex, France MeSH keywords: Animaux, Chromosomes artificiels de bactérie, Exons, Génome, Interactions hôte-parasite, Humains, Schistosoma, Schistosoma mansoni, Schistosomiase, Schistosomiase à Schistosoma mansoni, Transcription génétique, Régulation positive |
La publication de deux articles [
1,
2] sur le séquençage des génomes de deux des trois principales espèces de schistosomes parasites de l’homme, Schistosoma mansoni et Schistosoma japonicum, représente l’aboutissement de travaux débutés il y a quatorze ans. Initialement focalisé sur le transcriptome, via le séquençage d’Expressed sequence tags [
3,
4], le projet s’est ensuite orienté vers la cartographie et le séquençage génomique, grâce à la construction de banques de grands fragments dans des Bacterial artificial chromosomes (BAC) [
5]. Toutefois, le séquençage complet de certains de ces BAC, ainsi que le séquençage étendu de leurs extrémités, ont démontré l’impossibilité d’effectuer une « marche sur le génome » à cause de la présence massive de séquences répétées. Par conséquent, le séquençage du génome a été réalisé par la stratégie aléatoire (shotgun) sur les génomes entiers, conjointement par The institute for genome research/The J. Craig Venter institute et le Wellcome Trust Sanger institute pour S. mansoni et par le Chinese national human genome center à Shanghai pour S. japonicum. Néanmoins, les séquences répétées (rétrotransposons, rétroposons, etc.) ont rendu les assemblages des génomes difficiles, et ils restent incomplets à l’heure actuelle. |
Des génomes riches en séquences répétées Le séquençage des génomes des schistosomes présente un double intérêt. Tout d’abord, les schistosomes sont des pathogènes majeurs des pays tropicaux qui infectent plus de 200 millions d’individus dans 75 pays. Mais ces parasites sont également des métazoaires invertébrés représentant le clade des Lophotrochozoaires dont les génomes étaient jusqu’à présent méconnus contrairement aux Ecdysozoaires (tels Drosophila, Caenorhabditis, Brugia) pour lesquels les données génomiques sont nombreuses. Le génome du schistosome s’avère être plus grand (363 Mb pour S. mansoni) que celui des vers nématodes filaires (90 Mb pour Brugia malayi). Environ 40 % du génome est constitué de séquences répétées, dont 72 familles de rétrotransposons de type LTR (long terminal repeat) et non LTR. La comparaison entre les génomes de S. mansoni et S. japonicum permet de montrer, chez le premier, une flambée d’événements de transposition pour deux familles de rétrotransposons non LTR, après la divergence entre ces deux espèces. Malgré la proportion élevée de séquences répétées, 50 % du génome est assemblé dans de grands échafaudages (> 824 kb) et 43 % a été localisé sur les chromosomes (7 autosomes et les chromosomes Z et W déterminant le sexe) par hybridation in situ fluorescente. L’analyse du génome a permis l’identification de 11 809 gènes codant 13 197 transcrits. Une originalité des gènes caractérisés concerne les introns. En effet, la distribution de la taille de ces introns est très différente de celle des gènes des autres eucaryotes, les introns en 5’ des gènes étant globalement plus petits que ceux en 3’, suggérant ainsi un mode de contrôle transcriptionnel atypique. |
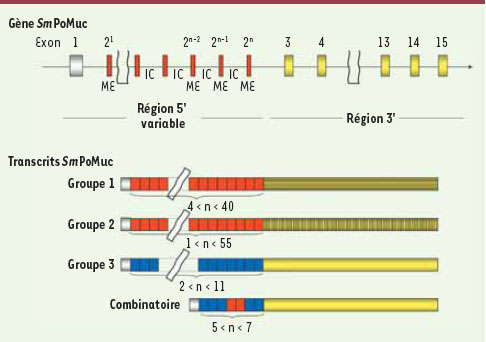
Des gènes avec une structure originale : les « MEG » Certains gènes se distinguent par une structure remarquable avec de nombreux « micro-exons » et sont nommés « MEG » pour micro exon genes. Bien que de tels exons (composés de 6 à 36 bases) soient présents de façon sporadique dans des gènes d’autres espèces, chez S. mansoni au moins 45 gènes possèdent des micro-exons composant 75 % de la séquence codante. Il n’y a que peu de points communs entre ces 45 gènes. Toutefois, presque tous codent un peptide signal en 5’ et trois des protéines codées par ces gènes seraient ancrées à la membrane. Par ailleurs, une famille de MEG, non annotée dans le génome de S. mansoni à cause de sa structure en micro-exons et de la présence d’introns répétés, a été décrite récemment [
6]. Cette famille de gènes, appelée SmPoMuc, code des protéines mucine-like, exprimées spécifiquement dans les stades parasitaires interagissant avec le mollusque d’eau douce Biomphalaria glabrata qui est l’hôte intermédiaire. Elles sont caractérisées par un polymorphisme exceptionnel (Figure 1) généré par une cascade de mécanismes complexes (un « chaos contrôlé ») maintenu par une « course aux armements » entre l’hôte et le parasite. En effet, les glycoprotéines correspondantes auraient pour fonction leur évasion de la réponse immunitaire de l’hôte. Il serait tentant de suggérer un rôle similaire pour les autres MEG dans les interactions avec l’hôte intermédiaire ou définitif. En effet, une grande variabilité des transcrits a été démontrée pour les trois autres familles de MEG étudiées jusqu’à présent.  | Figure 1.
Représentation schématique d’un gène SmPoMuc composé de 15 exons. La région 5’ est composée de micro-exons (ME) et d’introns conservés (IC) répétés n fois. Illustration du polymorphisme observé au niveau des transcrits correspondant aux SmPoMuc. Deux types d’unités (carrés rouge et bleu), répétées n fois et codées par les ME, composent la région 5’variable des transcrits. Il existe 3 groupes de transcrits discriminés par leur région 3’. |
|
Vers de nouvelles stratégies de lutte : une pléthore de cibles chimiothérapeutiques L’une des retombées principales du séquençage de ces génomes est l’identification de cibles moléculaires intéressantes pour le développement de nouvelles stratégies thérapeutiques. Une pléthore de telles cibles a été identifiée par deux stratégies bioinformatiques ; la reconstruction de voies métaboliques pour identifier des goulots d’étranglement et la recherche de gènes codant des protéines homologues à des cibles connues de drogues utilisées dans d’autres maladies. Par la première approche, 120 enzymes ont été identifiées et correspondent à des points clés du métabolisme. Certaines sont des cibles chimiothérapeutiques chez d’autres organismes et d’autres sont déjà caractérisées et ciblées chez S. mansoni. Le criblage « chimiogénomique » à la recherche des cibles de drogues déjà existantes qui pourraient être utilisées contre les schistosomes, a également fourni des résultats intéressants. Par exemple, le disulfiram, une drogue utilisée pour contrôler l’addiction, est potentiellement active contre les schistosomes, et ses propriétés antiparasitaires ont déjà été testées [
7]. On peut également citer des drogues ciblant les histones déacétylases, qui font l’objet de recherches intensives dans le domaine du cancer, dont la capacité d’induire l’apoptose et la mort chez les larves de S. mansoni a été démontrée [
8]. Toutefois, une stratégie de modification de ces drogues pour les rendre plus spécifiques du parasite sera indispensable pour leur application future à grande échelle. Le séquençage de ces deux génomes de parasites pathogènes majeurs de l’homme représente une étape clé permettant à la fois une meilleure compréhension de l’évolution de leur adaptation à leurs hôtes et l’espoir d’un développement rapide de nouvelles stratégies chimiothérapeutiques. Toutefois, l’analyse encore très incomplète de ces génomes montre la nécessité de continuer les efforts dans ce domaine. Étant donné la nature encore fragmentaire des séquences obtenues, des efforts supplémentaires de séquençage pour combler les manques, corriger les erreurs d’assemblage et améliorer l’annotation, semblent indispensables. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1. Berriman M, Haas B, Loverde PT, et al. The genome of the blood fluke Schistosoma mansoni. Nature 2009; 460 : 352–8. 2. The Schistosoma japonicum Genome Sequencing and Functional Analysis Consortium. The Schistosoma japonicum genome reveals features of host-parasite interplay. Nature 2009; 460 : 345–51. 3. Verjovski-Almeida S, De Marco R, Martins EA, et al. Transcriptome analysis of the acoelomate human parasite Schistosoma mansoni. Nat Genet 2003; 35 : 148–57. 4. Hu W, Tan Q, Shen DK, et al. Evolutionary and biomedical implications of a Schistosoma japonicum complementary DNA resource. Nat Genet 2003; 35 : 139–47. 5. Le Paslier MC, Pierce RJ, Merlin F, et al. Construction and characterization of a Schistosoma mansoni bacterial artificial chromosome library. Genomics 2000; 65 : 87–94. 6. Roger E, Grunau C, Pierce RJ, et al. Controlled chaos of antigenic variants in a metazoan parasite (Schistosoma mansoni) interacting with its invertebrate host (Biomphalaria glabrata). PLOS Neg Dis 2008; 2 : e330. 7. Seed JL, Pratt MC, Bennett JL. The effects of chronic disulfiram treatment on mice infected with Schistosoma mansoni. Am J Trop Med Hyg 1979; 28 : 508–14. 8. Dubois F, Caby S, Oger F, et al. Histone deacetylase inhibitors induce apoptosis, histone hyperacetylation and up-regulation of gene transcription in Schistosoma mansoni. Mol Biochem Parasitol 2009 (sous presse). |