| |
| Med Sci (Paris). 2009 October; 25(10): 871–877. Published online 2009 October 15. doi: 10.1051/medsci/20092510871.Apports de la chémo-informatique dans la recherche et l’optimisation des molécules d’intérêt thérapeutique Philippe Vayer,1* Alban Arrault,2* Brigitte Lesur,5* Marc Bertrand,3* and Bernard Walther4* 1Chef de section modélisation moléculaire 2Responsable de service modélisation moléculaire 3Directeur coordination biopharmacie 4Directeur du centre de pharmacocinétique et de métabolisme, Technologie Servier, 25-27, rue Eugène Vignat, BP 11749, 45007 Orléans Cedex 01, France 5Directeur de division chimie-partenariat modélisation
moléculaire, Institut de recherches Servier, 125, Chemin de ronde, 78290 Croissy-sur-Seine, France |
La recherche de candidats médicaments s’oriente de nos jours très tôt vers une analyse du potentiel de diversité chimique disponible, pour une large part dans des chémothèques (librairies ou banques de molécules) publiques ou privées. L’objectif est de cribler virtuellement (réduction du nombre candidats) puis expérimentalement des collections ciblées pour la recherche de molécules (hits) ayant des caractéristiques prometteuses sur de nouvelles cibles, mais également d’orienter le chimiste thérapeute, dès ce stade de tri, vers des molécules aux propriétés biopharmaceutiques (absence de toxicité et devenir dans l’organisme optimisé) compatibles avec le développement d’un futur candidat médicament. Ce criblage virtuel, utilisant un modèle de la cible, du type de ligand visé et/ou de propriétés biologiques ou physico-chimiques particulières, permet de tester très précocement et rapidement ces librairies de molécules. Les molécules identifiées sont testées expérimentalement, ce qui permet l’identification d’une ou de plusieurs têtes de séries (lead). La puissance sur la cible, la sélectivité vis-à-vis d’autres cibles proches et certaines propriétés biopharmaceutiques seront améliorées jusqu’à l’obtention d’un candidat médicament. Il sera testé sur des modèles animaux, outils thérapeutiques ayant des caractéristiques très proches de la maladie chez l’homme avant d’entrer dans les phases de développement. Cette approche moderne est basée ou accompagnée par des techniques dites de chémo-informatique, discipline permettant de décrire la relation entre la structure et les propriétés pharmacologiques et biopharmaceutiques (Chémo-informatique : outils de recherche, de description et d’exploitation des liens entre structures et propriétés).
La chémographie, sous-discipline de la chémo-informatique, permet de disposer d’outils très puissants dans la construction et l’exploitation de l’espace chimique ainsi que dans le suivi et l’optimisation des propriétés chimiques et biologiques des molécules d’un projet de recherche. |
Utilisation du criblage virtuel dans la sélection de molécules pour des tests à haut débit Le criblage virtuel a été historiquement essentiellement basé sur la connaissance de la structure tridimensionnelle de la cible (structure based virtual screening, SBVS). Dans ce cas, les molécules à évaluer sont d’abord arrimées (docking) artificiellement dans le site actif de la cible. L’interaction est ensuite quantifiée (scoring) pour conduire à la sélection de nouvelles entités chimiques potentiellement actives [
1,
2]. L’autre alternative, que nous allons développer plus avant dans cet article, utilise certaines propriétés de la molécule et des activités biologiques mesurées comme point de départ (ligand based virtual screening, LBVS) [
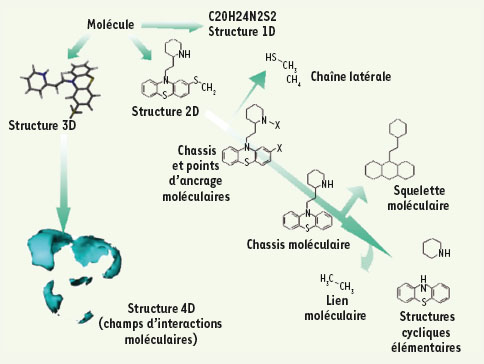
3]. Elle repose sur l’utilisation des descripteurs moléculaires (de topologie, de géométrie, d’interactions électroniques et/ou lipophiles, de propriétés quantiques ou graphes moléculaires), véritable carte d’identité d’une molécule. La Figure 1 résume les différents niveaux de description d’une molécule en commençant par les plus simples, le nom et la représentation chimique, jusqu’aux descripteurs 3D faisant appel à des volumes et des surfaces moléculaires (4D). 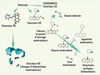 | Figure 1.
Niveaux de description d’une molécule utilisés dans le criblage virtuel. Les descripteurs 1D sont déduits de la formule brute (nombres d’atomes, nombre de C, O, N …)., les descripteurs 2D de la structure plane de la molécule (donneurs/accepteurs d’hydrogène, nombre de cycles…). Les descripteurs 3D sont déduits de la structure 3D de la molécule (volume, surface accessible au solvant…). La notion de 4D peut être employée quand on explore l’espace autour de la molécule (surfaces moléculaires). |
Il existe plus de 3 000 descripteurs différents recensés, mais nombre de ces descripteurs contiennent le même type d’information. Simple filtrage via des descripteurs chimiques Ces descripteurs sont utilisés pour filtrer des bases de structures chimiques permettant d’éliminer des composés dont les caractéristiques sont trop éloignées de celles d’un médicament. Les règles dites de Lipinsky utilisent, par exemple, une combinaison de propriétés physicochimiques essentielles dans l’absorption orale [
4]. Mais on peut également vouloir s’affranchir de certains fragments chimiques comme :
-
les fonctions réactives susceptibles de former des liaisons covalentes [
5] ;
-
les fonctions connues comme générant des faux positifs pour des raisons physico-chimiques du type agents chélatants, inhibiteurs suicides, composés polyanioniques, etc. [
6] (warheads) ;
-
les fonctions chimiques susceptibles de réaliser des agrégats (promiscuous aggregating inhibitors) et faussant le résultat biologique [
7].
Ces approches sont souvent utilisées dans des processus de focalisation de bases. Elles permettent, dans le travail de Monge et al. [
8], de réduire une base chimique de 173 803 composés à 10 000 molécules ayant des propriétés compatibles avec celles d’un futur médicament. Mais elles ont également été utilisées avec succès pour enrichir des chimiothèques avec des apports externes tant sur la base du potentiel médicamenteux que d’un point de vue diversité chimique. Sélection à l’aide de modèle QSAR Ensuite s’engage la construction du modèle, par des approches QSAR ( quantitative structure activity relationship) le plus souvent [
9]. Ces techniques permettent de relier par une relation mathématique les descripteurs chimiques, soit à l’activité biologique dans une approche de relation structure-activité quantitative, soit à une propriété (physicochimique ou pharmacocinétique) dans le cas des relations structure-propriété quantitative (QSPR pour quantitative structure property relationship). De tels modèles QSAR ou QSPR, basés sur des descripteurs 2D ou 3D, sont le point de départ de nombreux processus de sélection de molécules. Ces modèles sont tous construits sur le même mode mettant en œuvre un jeu de référence appelé jeu d’apprentissage permettant de sélectionner le(s) descripteur(s) le(s) plus adapté(s) dans la construction d’un modèle. Les modes de construction de ces modèles peuvent être simples (régression linéaire ou multilinéaire) ou plus sophistiqués permettant de rechercher la meilleure combinaison de descripteurs (algorithmes génétiques, réseaux de neurones, forêts aléatoires, séparateurs à vaste marge). Elle sera suivie d’une étape de validation appelée validation externe et réalisée avec un nouveau jeu de données, souvent généré postérieurement, et qui va permettre de bien mesurer la pertinence du modèle et de définir le domaine d’application. Le pouvoir prédictif de ces modèles et leur domaine d’application pour de futures séries chimiques dépendent de la composition du jeu d’apprentissage utilisé pour construire le modèle. Il faut se rappeler qu’un modèle QSAR parfait ne pourra donc pas permettre une prédiction pour toute sorte de substances (molécules trop différentes de celles du jeu d’apprentissage) et avoir un niveau de précision supérieur à celui de la méthode expérimentale qui a permis de générer les données du jeu d’apprentissage. |
Espace chimique et domaine d’application Il est essentiel dans les approches de criblage virtuel de connaître le potentiel de diversité chimique que le chercheur a à sa disposition. Cela requiert : une représentation unique de l’espace chimique, une définition de la méthode et des critères à utiliser pour différencier les molécules (similarité et dissimilarité) et surtout une bonne compréhension des limites d’utilisation de ces méthodes (le type de descripteur utilisé), point clé de la réussite et de la crédibilité de ces approches de diversité chimique. L’espace chimique L’espace chimique total comprend l’ensemble des molécules organiques possibles (entre 10 14 à 10 200 molécules). Si tout cet espace n’est pas nécessaire pour un projet donné qui va se focaliser sur une zone bien définie de cet espace, il est tout de même essentiel de pouvoir se repérer et d’y naviguer pour comparer différents projets. Ceci implique, pour un type de description, de pouvoir fixer cet espace multidimensionnel (axes, distances, etc.) et ce sont des molécules choisies comme références qui vont pouvoir nous aider dans cette tâche. Selon la nature des descripteurs utilisés, des méthodes de représentation seront mieux adaptées à certains sujets que d’autres. Des sous-ensembles de quelques centaines à quelques milliers de molécules, représentatifs de toutes les autres sont identifiés. Les tris de molécules peuvent être réalisés par des analyses de groupement (clustering) permettant de classer les molécules en un certain nombre de groupes homogènes ayant des caractéristiques chimiques et/ou biologiques communes ou proches. Lors des phases initiales de recherche de molécules prometteuses, on peut travailler sur un sous-ensemble de molécules assez diverses qui permet de couvrir la diversité moléculaire de la chimiothèque initiale. En revanche, une fois une tête de série identifiée, il est important de pouvoir explorer l’espace plus proche de la molécule choisie afin de ne pas trop s’éloigner des propriétés intéressantes identifiées. La codification de cet espace, global d’une part dans la recherche d’une diversité la plus grande possible, et local d’autre part lors de phases d’optimisation, est essentielle dans toutes les approches virtuelles. Le domaine d’application des modèles Les modèles QSAR ne couvrent généralement qu’une partie de l’espace chimique, ils sont focalisés sur les molécules qui ont servi à les créer et les prédictions réalisées avec ces modèles ne sont valables que dans cette même zone de l’espace chimique. Le domaine d’applicabilité correspond à la zone de l’espace chimique dans laquelle les propriétés sont interpolées. En dehors de ce domaine, on est amené à réaliser une extrapolation qui reste le point critique de ces approches. Il est dès lors essentiel de pouvoir, pour toute molécule nouvelle, y associer une fiabilité de la prédiction réalisée et, si nécessaire, de ne pas faire de prédiction dans le cas où la molécule est en dehors du domaine d’application. Le domaine d’application correspond à la région de l’espace chimique incluant le jeu d’apprentissage et les molécules qui lui sont similaires. Il faut pouvoir déterminer si une molécule appartient ou non au périmètre initial du modèle, et si elle s’en écarte, la tolérance que l’on peut s’autoriser sans mettre en péril les prédictions. Le jeu de validation externe du modèle est essentiel dans la détermination de cette tolérance d’utilisation d’un modèle. La détermination du domaine d’application repose sur des analyses de descripteurs ou de similitudes moléculaires. La plus stricte (la frontière de boîte) n’autorise aucune extrapolation. Si, en revanche, pour chaque nouvelle molécule, on peut déterminer ses plus proches voisins dans le jeu d’apprentissage, la moyenne des distances obtenues et le choix d’un seuil de distance peuvent être utilisés pour discriminer une molécule dans ou en dehors du domaine d’application. |
La représentation d’objets chimiques (structures chimiques ou de schémas réactionnels) est un élément important dans la perception et l’analyse de l’information chimique, tant au niveau de l’exploitation de la diversité chimique que de la corrélation avec des propriétés expérimentales. La représentation de structures chimiques sous forme de points dans un espace chimique peut être assimilée à celle d’un objet dans l’espace qui nous entoure. Par analogie avec une représentation cartographique, il est nécessaire de fixer une unité de distance indépendante des objets observés (appelée métrique), incluant un repère de référence ainsi qu’une méthode de projection de l’espace multidimensionnel sur un plan. Ce type d’approche a donné naissance à une description graphique de l’espace chimique (chémographie) où, à l’instar de la navigation routière (2D) ou aérienne (3D), il est possible de créer une représentation de l’espace basée sur des descripteurs moléculaires ou sur des combinaisons simples de ces descripteurs, mais également de se déplacer d’une zone de l’espace vers une autre en intégrant des propriétés chimiques cibles [
10]. Il est d’ailleurs fréquemment appelé système de GPS (global positioning system) chimique. Dans de telles représentations, le référentiel ainsi que la métrique dépendent du jeu de molécules de référence appelées également « satellites », placées intentionnellement à l’extérieur de l’espace chimique étudié. Ils sont un moyen de comparaison d’espaces chimiques ou de collections de molécules. Les coordonnées de chaque nouvelle molécule sont déterminées dans ce repère de satellites (molécules de références) et elles peuvent, sur la base des mêmes descripteurs moléculaires, être projetées dans l’espace chimique. Oprea et al. ont appliqué cette approche à la prédiction de l’absorption orale de molécules en utilisant les paramètres et concepts utilisés dans la classification biopharmaceutique des médicaments. Cette dernière, basée sur les paramètres de solubilité et de perméabilité membranaire des molécules, permet de regrouper les molécules en 4 classes. Par exemple, les molécules de la classe IV qui correspondent aux molécules de faible solubilité et de faible perméabilité, sont connues pour avoir des caractéristiques d’absorption non optimales menant à des variabilités d’exposition chez l’homme très difficiles à gérer dans le développement d’un médicament. Pour ce faire, les auteurs ont choisi un ensemble de satellites situés en périphérie de l’espace chimique, sélectionnés de façon à obtenir une diversité chimique maximale. Le système de GPS chimique (ChemGPS) est basé sur l’utilisation de descripteurs moléculaires décrivant les interactions des composés avec leur environnement (champs d’interaction moléculaire basés sur le logiciel GRID) [
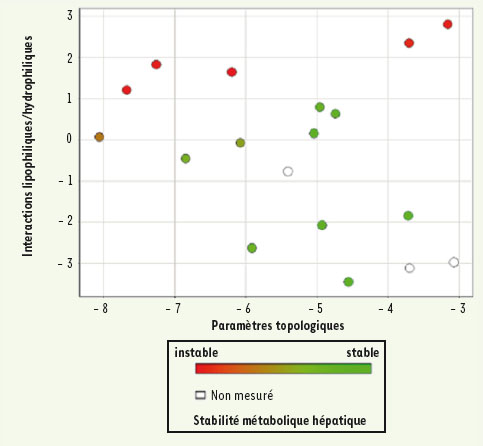
11]. Cette approche donne une visualisation de la classification biopharmaceutique, permettant non seulement de classer les composés, mais également de représenter la direction à prendre (propriétés à améliorer) pour atteindre la zone d’absorption maximale. L’utilisation d’un jeu de satellites et l’approche de type ChemGPS décrite précédemment, utilisant des descripteurs moléculaires issus de Volsurf, ont permis de décrire, d’utiliser et de valoriser le patrimoine chimique des Laboratoires Servier, tant du point de vue de la diversité chimique que du point de vue de leurs propriétés pharmacologiques et biopharmaceutiques. En revanche, des satellites internes à l’espace chimique ont été ajoutés réalisant un maillage plus précis de l’espace chimique pour l’ensemble de nos composés. Cent satellites (molécules) permettent de décrire plus de 70 % de l’information chimique. Si un code couleur est attribué à chaque molécule en fonction d’une propriété choisie, ici la stabilité métabolique (Figure 2), cette approche devient, en comparant simultanément la diversité chimique et expérimentale, un outil d’exploitation simple des données dans l’analyse avancée de résultats (data mining).  | Figure 2.
Représentation de la diversité chimique de type GPS (global positioning system) basée sur la notion d’interactions avec les membranes (interactions hydrophiles/lipophiles) et de formes (paramètres topologiques). Le code couleur renseigne sur une propriété biologique, ici la stabilité métabolique des produits de la série. |
Cette représentation chϩmographique peut, également grâce à la partition fine de l’espace chimique (cell based approach), intégrer la notion de densité chimique dans une zone donnée de l’espace. Pour comprendre cette approche originale réalisée par Arrault et al. [
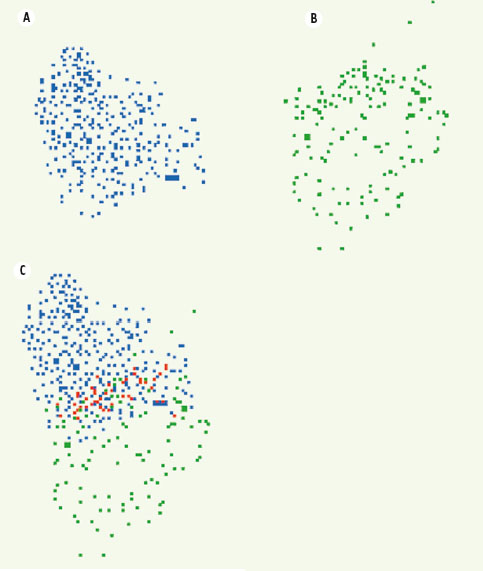
12], on peut faire une analogie avec la photographie, pour laquelle l’espace photographié est divisé en pixels comme ici l’espace chimique a pu être divisé en cellules représentant un damier. Chaque cellule peut comprendre 0, une ou plusieurs molécules et en fonction de la résolution utilisée (nombre de cellules totales pour un même espace), il est possible de changer la finesse de l’exploration de l’espace chimique. L’outil Grider, issu de ce travail d’exploitation de la densité chimique, permet de comparer la complémentarité de deux jeux de molécules (Figure 3). Il est ainsi possible de mieux approcher les descriptions de jeux de données et de leurs diversités relatives et de permettre, en particulier, d’exploiter des zones restreintes de l’espace chimique, dans des phases d’optimisation de molécules.  | Figure 3.
Comparaison de deux bibliothèques de molécules dans la représentation GPS (global positioning system), permettant d’augmenter la diversité chimique en éliminant les redondances de molécules. A. Collection 1. B. Collection 2. C. Comparaison de collections avec identification en rouge des molécules communes ou proches des deux collections. |
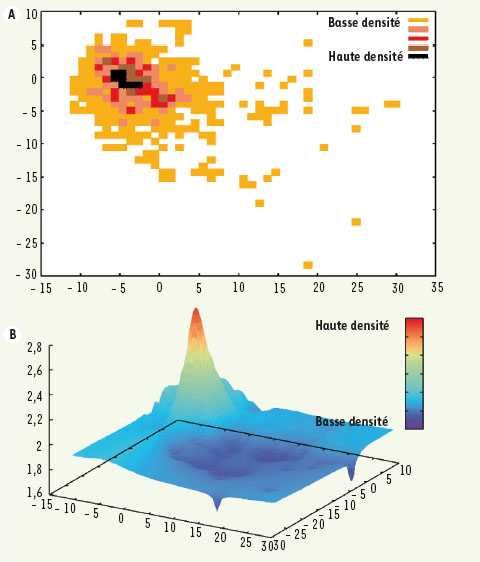
On peut également réaliser des cartes de densité d’une chimiothèque. La Figure 4 illustre la répartition des médicaments sur le marché. Ces molécules sont localisées dans une zone relativement restreinte de l’espace chimique global et permettent de montrer tout le potentiel théorique de diversité chimique disponible pour trouver de nouveaux médicaments, en dehors et à l’intérieur de cet espace. Afin de mieux exploiter cette diversité, Rosen et al. [
13] ont introduit le ChemGPS-NP où de nouvelles idées de têtes de séries sont basées sur des composés naturels proches de composés actifs connus dans le traitement de certaines maladies.  | Figure 4.
Carte de densité d’une chimiothèque (médicaments sur le marché) réalisée avec l’outil Grider [29]. A. Carte de densité 2D. B. Carte de densité 3D (faible densité (low density) et forte densité (high density) de molécules). |
Pour aller encore plus loin dans ce qui peut être codé dans un graphique, certains ont ajouté une dimension supplémentaire, réactionnelle, à la représentation chimique des molécules. Il est en effet possible de coder dans une seule formule chimique un point d’attaque chimique ou enzymatique, ainsi que la réaction concernée. Les travaux récents de l’équipe d’Aires de Sousa [
14] permettent de manipuler des cycles enzymatiques impliqués dans le fonctionnement cellulaire, et ceux de Faulon et al. [
15] exploitent les réactions décrites dans la base KEGG. Les enzymes du métabolisme du médicament peuvent ainsi être représentées par leur activité [
16] et des comparaisons de similitude fonctionnelle et des classifications d’enzymes sont alors possibles. |
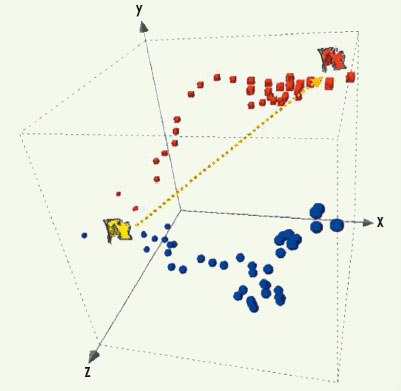
L’ensemble de ces outils chémographiques permet aujourd’hui d’intégrer dans un projet de recherche donné l’ensemble des informations chimiques vis-à-vis d’une propriété biologique, qu’elles soient bibliographiques, mesurées expérimentalement ou calculées in silico, afin de faciliter l’exploitation des « profils de développabilité » de chaque molécule. Par extension de la chémographie décrite précédemment, la pharmacographie vise à utiliser une approche condensée similaire dans l’esprit au ChemGPS, mais basée sur l’ensemble des propriétés biologiques disponibles. Chaque molécule reste représentée par un point dans ce nouvel espace de résultats (que l’on a baptisé GPR pour global positioning of results ou BioGPS pour biological global positioning system). L’impact de chaque type de résultats biologiques pourra être modulé par des pondérations différentes en fonction du type de projet ou de cible et de l’avancée du projet. Ainsi deux molécules proches dans ce type d’espace ont des profils biologiques (activité, toxicité, comportement biopharmaceutique, etc.) similaires. La Figure 5 illustre cette approche avec l’évolution de l’optimisation de deux anciennes séries chimiques représentées dans le GPR. L’ensemble des données physico-chimiques, pharmacologiques et biopharmaceutiques se retrouve intégré dans une même visualisation (condensée derrière chaque axe). On peut différencier avec des codes couleurs les deux séries choisies dans cet exemple, mais également suivre par la taille des points l’évolution chronologique du projet et le chemin parcouru par rapport au chemin théorique.  | Figure 5.
Représentation de deux anciennes séries chimiques dans un espace de résultats (axe des x : propriétés biopharmaceutiques ; y : propriétés pharmacologiques ; z : propriétés physicochimiques). La taille des symboles permet de suivre l’évolution chronologique des deux projets (matérialisés par les carrés rouges et les sphères bleues) entre le début du projet (drapeau jaune) et le cahier des charges du projet de recherche (drapeau rouge). La flèche jaune permet de visualiser le chemin idéal d’optimisation. |
Cette approche que l’on pourrait qualifier d’hypercondensée permet de réaliser un suivi nettement facilité des deux projets de recherche et une sélection de molécules conforme au cahier des charges. Les deux projets représentés ont été améliorés, le bleu sur des axes biopharmaceutiques et chimiques uniquement tandis que le projet rouge intègre également la composante pharmacologique essentielle pour un candidat médicament Cette notion de molécule idéale, véritable cahier des charges virtuel d’un projet, peut être introduite dans ce GPR et la distance cartographique entre les molécules d’un projet et cette cible à atteindre correspond, en quelque sorte, au chemin restant à parcourir idéalement lors des phases d’optimisation dans les dimensions pharmacologiques et biopharmaceutiques. Cette nouvelle dimension, que l’on pourrait qualifier de supra-biologique, combiné à un Chem GPS, permet désormais de manipuler les données biologiques dans leur ensemble en combinaison avec la diversité chimique des molécules. Une application élégante de Cheider et al. [
17] permet de naviguer dans l’espace chimique construit à partir des effets secondaires de médicaments (adverse reactions) donnant la possibilité de prédire mais également de « corriger » la structure chimique des composés. |
La chémo-informatique doit aujourd’hui être vue comme un outil extrêmement puissant et adapté à la transformation de simples données générées dans les programmes de recherche en vraies connaissances essentielles pour l’innovation thérapeutique. Mais au-delà de l’aide efficace au tri intelligent de données (data mining), ces nouveaux outils vont permettre de focaliser les efforts des chercheurs et des développeurs dans les directions les plus prometteuses. Ce type d’outil permet un gain de temps dans l’exploitation et la compréhension des programmes de recherche, tout en conservant une vue globale (limites et orientations) du projet, ce qui nous paraît essentiel aujourd’hui dans l’univers compétitif de la pharmacie. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
1. Walters WP, Stahl MT, Murcko MA. A virtual screening: an overview. Drug Discov Today 1998; 3 : 160–78. 2. Shoichet BK. Virtual screening of chemical librairies. Nature 2004; 432 : 862–5. 3. Hristozov DP, Oprea TI, Gasteiger J. Virtual screening applications: a study of ligand based methods and different structure representations in four different scenarios. J Comput Aided Mol Des 2007; 21 : 617–40. 4. Lipinsky CA. Lead to drug like compounds : the rule of five revolution drug like properties. Drug Discov Today 2004; 1 : 337–41. 5. Opera T. Property distribution of drug-related chemical databases. J Comput Aid Mol Des 2000; 14 : 251–64. 6. Richton G. Nonleadlikness and leadlikness in biochemical screening Drug Discov Today 2003; 8 : 86–96. 7. Serdler J, Mc Govern S, Danan T, Shaichet B. Identification and prediction of promiscuous aggregating inhibitors among known drugs. J Med Chem 2003; 46 : 4447–86. 8. Monge O. Création et utilisation de chimiothèques optimisées pour la recherche in silico de nouveaux composés bioactifs. Novembre 2006, réalisé à Orléans. 9. Sperandio O, Miteva MA, Villoutreix BO. Combining ligand-and struture-based methods in drug design projects. Curr Comput Aid Drug Design 2008; 4 : 250–8. 10. Oprea TI, Gottfries J. Chemography: the art of navigating in chemical space, J Comb Chem 2001; 3 :157–66. 11. Oprea TI, Zamora I. Ungel AL. Pharmacokinetically based mapping device for chemical space navigation. J Comb Chem 2002; 4 : 258–66. 12. Arrault A, Hamon V, Vayer P, Bertrand M. “Grider”, a visual data mining tool for inspection of chemical spaces. Extension to diversity exploration. Poster Ecole d’été d’Obernai juin 2008. « A visual data mining tool for inspection of chemical spaces. Extension to diversity exploration soumis à J Chem Inf Model
13. Rosen J, Gottfries J, Muresan S, et al. Novel chemical space exploration via natural products. J Med Chem 2009; 52 : 1953–62. 14. Latino DA., Aires-de-Sousa J. Genomic-scale classification of metabolic reactions: a chemoinformatics approach Angew. Chem Int 2006; 45, 13 : 2066–9. 15. Faulon JL, Misra M, Martin S, et al. Genome-scale enzyme-metabolite and drug-target interaction predictions using the signature molecular descriptors. Bioinformatics 2008; 24 : 225–233. 16. Ridder L, Wagener M. SyGma: combining expert knowledge and empirical scoring in the prediction of metabolites. Chem Med Chem 2008; 3 : 821–32. 17. Scheiber J, Jenkins JL, Sukuru SC, et al. Mapping adverse reactions in chemical space. J Med Chem 2009; 52 : 3103–7. |