| |
| Med Sci (Paris). 2008 August; 24(8-9): 753–757. Published online 2008 August 15. doi: 10.1051/medsci/20082489753.L’empreinte parentale, un phénomène épigénétique essentiel pour la reproduction des plantes Pauline E. Jullien and Frédéric Berger* Temasek Life Sciences Laboratory, 1 Research Link, National University of Singapore, 117604 Singapore, République de Singapour |
La reproduction sexuelle chez les plantes et les mammifères est marquée par un mode de contrôle particulier de l’expression des gènes, l’empreinte génétique parentale. Certains gènes, dits « imprimés », sont exprimés principalement par l’un des deux allèles parentaux et ce d’une façon stable au cours du développement embryonnaire et dans certains cas chez l’adulte. Cette expression monoparentale résulte de modifications de l’environnement de la séquence codante du gène imprimé. L’embryon des plantes et des mammifères se développe dans les structures reproductrices maternelles. Le lien entre l’embryon et la mère est assuré par une structure extra-embryonnaire, le placenta chez les mammifères et l’albumen chez les plantes. L’empreinte génomique parentale affecte les gènes exprimés dans les tissus extra-embryonnaires responsables de l’acheminement des nutriments d’origine maternelle vers l’embryon. Les mécanismes moléculaires de l’empreinte génétique parentale chez les mammifères ont été étudiés depuis les années 1990 [
28,
30] (→).
(→) Voir l’ article de A. Henckel et R. Feil, page 747 de ce numéro
Chez les plantes, une partie des mécanismes impliqués dans la mise en place et le maintien de l’empreinte n’a été dévoilée que récemment. Ces études montrent une remarquable convergence entre l’évolution de l’empreinte dans ces deux groupes d’organismes qui ont pourtant divergé l’un de l’autre il y a plus d’un milliard d’années. Cette convergence évolutive indique une probable similitude des causes et forces évolutives en oeuvre. Cette revue présente une vue d’ensemble des mécanismes impliqués et des fonctions des gènes soumis à l’empreinte chez les plantes. |
Mécanismes moléculaires de l’empreinte Les plantes et les mammifères sont des organismes diploïdes et les cellules héritent, pour chaque gène, d’un allèle de la part de chaque parent. Un gène soumis à l’empreinte est caractérisé par un déséquilibre du niveau d’expression des allèles parentaux. Dans les cas extrêmes, un allèle est exprimé tandis que l’autre allèle est conservé dans un état silencieux. Cet état silencieux résulte de la méthylation de l’ADN. Cette méthylation affecte les résidus cytosines associés à un résidu guanine (CG). Le maintien de la méthylation est essentiel pour l’état imprimé du gène. Ainsi l’allèle silencieux d’un gène particulier est marqué par la méthylation des CG tandis que cette méthylation est absente sur l’autre allèle. Chez les plantes, les gènes soumis à empreinte sont isolés les uns des autres dans le génome contrairement aux locus comprenant plusieurs de ces gènes décrits chez les mammifères. Des études récentes ont identifié trois gènes dont l’empreinte est contrôlée directement par la méthylation de l’ADN : deux gènes d’Arabidopsis FERTILIZATION INDEPENDENT SEED 2 (FIS2) [
1], FLOWERING LOCUS A (FWA) [
2] et un gène de maïs FERTILIZATION INDEPENDENT ENDOSPERM (FIE) [
3,
4]. Dans chaque cas, la région méthylée est située en amont de la séquence codante des gènes (Figure 1). Dans le cas de FWA la région méthylée est formée de deux répétitions [
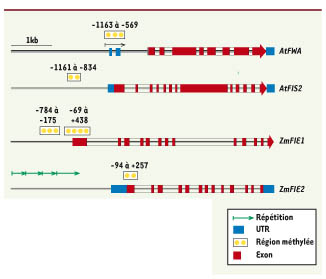
5] tandis que pour FIS2 et FIE, la région méthylée ne présente pas de particularité à l’exception d’une densité plus élevée en CG que la moyenne.  | Figure 1.
Représentation schématique des locus soumis à empreinte contrôlés par la méthylation de l’ADN.
AtFWA et AtFIS2 sont deux gènes soumis à empreinte et exprimés par l’allèle maternel dans l’albumen d’Arabidopsis thaliana. ZmFIE1 et ZmFIE2 sont deux gènes soumis à empreinte et exprimés par l’allèle maternel dans l’albumen du maïs. Les régions méthylées en 5’ des gènes sont indiquées sur le schéma, les positions de ces régions sont indiquées en fonction du site d’initiation de la traduction ou ATG. Les répétitions sont indiquées par des flèches. |
|
Le rôle clé des méthyltransférases dans le maintien de l’empreinte Chez Arabidopsis, la méthylation des CG est maintenue par la méthyltransférase MET1, homologue de la méthyltransférase Dnmt1 des mammifères [
6]. Cette enzyme est très probablement produite pendant la phase S du cycle cellulaire et agit à la fourche de réplication de l’ADN causant une conservation du patron de méthylation sur les nouveaux brins d’ADN à partir du patron préexistant [
7] (Figure 2). Le maintien de l’état d’empreinte requiert l’activité de la méthyltransférase tant chez les plantes que chez les animaux [
8]. Les gènes FWA et FIS2 perdent leur état imprimé dans les tissus privés d’activité méthyltransférase [1]. L’empreinte est donc basée sur le maintien d’une différence de méthylation entre les deux allèles parentaux. Chez les mammifères, l’empreinte est maintenue pendant le développement embryonnaire et pour certains gènes se poursuit au-delà et même à l’état adulte. Chez les plantes l’empreinte génomique n’a été démontrée que dans l’albumen et ne peut être transmise à la descendance puisque l’albumen ne participe pas directement au développement embryonnaire [
9].
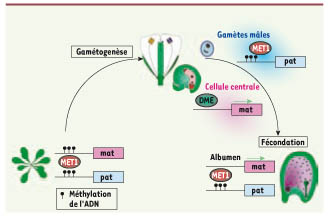
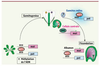 | Figure 2.
Cycle de l’empreinte lié à la méthylation de l’ADN chez Arabidopsis. Durant la croissance végétative d’Arabidopsis, la méthyltransférase de maintenance, MET1, maintient l’allèle paternel et l’allèle maternel inactifs en copiant le patron de méthylation de l’ADN au cours des réplications de l’ADN. Pendant la gamétogenèse femelle, la glycosylase DEMETER (DME) active l’expression de l’allèle maternel en enlevant la méthylation de l’ADN. Dans le pollen, le patron de méthylation est maintenu par MET1 sur l’allèle paternel inactif. Après la fécondation, le gène est seulement exprimé par l’allèle maternel qui est actif alors que l’allèle paternel est maintenu inactif par MET1. Dans le lignage embryonnaire, les deux allèles sont maintenus inactifs par l’action de MET1. Ce cycle est caractéristique de l’empreinte de FWA et FIS2. |
La méthyltransférase maintient un état imprimé mais ne peut le créer puisque l’activité enzymatique nécessite un patron de méthylation préexistant. Il existe donc un mécanisme spécifique qui met en place l’empreinte. Ce mécanisme a lieu lors de la gamétogenèse avec la création de marques distinctes selon le type de gamète, mâle ou femelle [9, 30] (→). (→) Voir l’ article de A. Henckel et R. Feil, page 747 de ce numéro
Chez les plantes un mécanisme particulier a été identifié pour l’activation de gènes soumis à empreinte exprimés par l’allèle maternel [
10]. Au cours du cycle de vie de la plante, FWA et FIS2 sont exprimés d’abord dans le gamète femelle qui produit l’albumen - la cellule centrale - et restent silencieux dans les gamètes mâles [1] (Figure 2). Il se produit donc au cours de la gamétogenèse mâle, un maintien de l’état silencieux tandis que l’état silencieux est perdu à la fin de la gamétogenèse femelle. Cette perte apparaît résulter de l’élimination de la méthylation de l’ADN. Deux mécanismes peuvent, en théorie, concourir à cette élimination. Un mécanisme passif par dilution des marques en l’absence de l’activité de MET1, ou l’élimination active des marques. Chez Arabidopsis, il a été démontré que la glycosylase DEMETER (DME), exprimée dans la cellule centrale à maturité, cause l’excision des résidus cytosine méthylés [
11]. La glycosylase DME fait partie d’une petite famille de gènes dont les autres membres sont exprimés dans les tissus végétatifs et dont le rôle n’est pas encore compris. DME élimine la méthylation des cytosines des éléments cis causant ainsi l’activation transcriptionnelle de FIS2 et FWA dans la cellule centrale [1]. Contrairement à ce qui avait été décrit initialement [1, 11] l’expression de DME a été détectée dans les tissus végétatifs [
12] et le pollen [
13]. Cette observation met en doute le rôle exclusif de DME dans l’activation des gènes soumis à l’empreinte, et d’autres facteurs doivent agir dans la cellule centrale pour garantir l’activation spécifique des gènes soumis à l’empreinte. Tout récemment, il a été démontré que la transcription de MET1 est inhibée par la voie du rétinoblastome, causant une déméthylation de l’ADN [
31]. |
Méthylation des histones et maintien de l’empreinte parentale Un mécanisme additionnel lié à l’empreinte génétique parentale mais indépendant de la méthylation de l’ADN, a été récemment démontré. Ce mécanisme repose sur d’autres types de marques transmises au cours des divisions cellulaires. Les histones 2A, 2B, 3 et 4 qui constituent les nucléosomes autours desquels est enroulé l’ADN, possèdent toutes des extensions amino-terminales qui peuvent être modifiées par une grande variété de complexes multiprotéiques. Ces modifications forment un code, interprété par d’autres complexes protéiques qui régulent l’accessibilité des promoteurs aux facteurs de transcription [
29]. Le code histone a de nombreuses fonctions qui ne sont pas discutées ici. Parmi les divers types de modifications, la méthylation des résidus lysine de l’histone H3 est l’un des codes les mieux caractérisés. Particulièrement, la méthylation sur le résidu lysine 27 de l’histone H3 (H3K27me) est assurée par un complexe Polycomb de type 2 [
14]. Les complexes Polycomb mettent en place une répression transcriptionnelle et sont conservés chez tous les eucaryotes pluricellulaires. Chez les plantes, la marque H3K27me différencie les allèles paternel et maternel du gène MEDEA (MEA) [11,
15] (Figure 3). MEA est exprimé maternellement dans l’albumen et l’allèle paternel de MEA est réprimé par le maintien des marques H3K27me par le complexe Polycomb exprimé dans l’albumen. De façon similaire, l’allèle maternel du gène PHERES1 est réprimé par H3K27me et l’allèle paternel est principalement exprimé dans l’albumen [
16]. Des résultats récents suggèrent une interaction entre la méthylation de l’ADN et celle des histones. La répression de l’allèle maternel du gène PHERES1 dépend aussi de l’état méthylé d’une région située à plus de 2 kb en aval du gène [
17]. Cette action à distance rappelle le mode de régulation des gènes soumis à empreinte des mammifères [30] (→).
(→) Voir l’ article de A. Henckel et R. Feil, page 747 de ce numéro
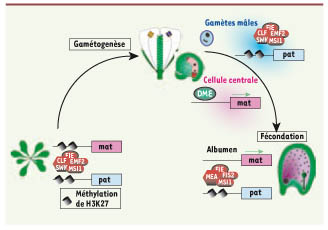
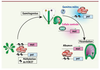 | Figure 3.
Cycle de l’empreinte lié à la méthylation de l’histone 3 sur la Lysine 27 (H3K27) chez Arabidopsis. Durant la croissance végétative d’Arabidopsis, le complexe Polycomb de type 2, constitué de quatre sous-unités, maintient l’allèle paternel et l’allèle maternel inactifs en méthylant H3K27. Pendant la gamétogenèse femelle, la glycosylase DEMETER (DME) active l’expression de l’allèle maternel par un mécanisme inconnu. Dans le pollen, la méthylation de H3K27 est maintenue par le complexe Polycomb de type 2 sur l’allèle paternel inactif. Aprés la fécondation, le gène est seulement exprimé de l’allèle maternel qui est actif alors que l’allèle paternel est maintenu inactif par le complexe Polycomb de type 2. Ce cycle est caractéristique de l’empreinte de MEA. |
De façon remarquable chez la souris, les marques H3K27me différencient également l’état d’expression des allèles maternel et paternel de plusieurs gènes exprimés dans le placenta [
18,
19]. Ainsi les mêmes mécanismes liés à des marques répressives ont été recrutés au cours de l’évolution de l’empreinte chez les mammifères et les plantes [9]. On ne connaît pas encore les mécanismes responsables de l’élimination des marques H3K27me dans l’un des deux types de gamètes et qui sont nécessaires à la mise en place de l’empreinte. |
Le rôle essentiel de l’empreinte a été démontré par les expériences qui ont conduit à sa découverte. Chez les plantes, des croisements entre plantes diploïdes et tétraploïdes ont montré que l’augmentation du dosage des allèles maternels n’a pas un effet équivalent à l’augmentation du dosage des allèles paternels [
20]. Une augmentation de la dose de génome maternel cause une réduction de la taille de l’albumen ; au contraire, celle-ci augmente quand la dose de génome paternel augmente. Ces résultats indiquent que l’activité des gènes soumis à empreinte cible la régulation de la taille de l’annexe embryonnaire, l’albumen. Des expériences similaires réalisées chez la souris ont abouti à des conclusions analogues [
21]. Dans le placenta des mammifères, la proportion de gènes exprimés soumise à l’empreinte est supérieure à la moyenne. La dérégulation de l’empreinte de l’ensemble de ces gènes, ou de certains d’entre eux, affecte également la croissance du placenta embryonnaire (trophoblaste). Chez Arabidopsis les gènes FIS2, MEA et FIE sont des membres du complexe Polycomb de type 2 qui est actif uniquement dans l’albumen [
22]. Ce complexe contrôle la taille de l’albumen probablement par la régulation d’une structure impliquée dans le transfert des nutriments d’origine maternelle vers l’albumen. Cette fonction générale de l’empreinte est aussi mise en évidence par l’impact sur le développement de l’albumen de l’absence d’un des génomes parentaux [
23]. Ainsi la fonction globale de l’empreinte génomique parentale apparaît liée au développement des annexes embryonnaires. |
La sélection et la conservation de mécanismes aboutissant à l’empreinte génomique indiquent une convergence évolutive entre plantes et mammifères [9]. Cette idée est également soutenue par la similitude des fonctions liées aux gènes soumis à empreinte, et en particulier par le rôle prépondérant que joue l’empreinte génomique sur les annexes embryonnaires. Plusieurs théories ont été avancées pour expliquer l’origine évolutive de l’empreinte et nous n’en citerons ici que deux.
-
Les développements de l’albumen et du placenta sont profondément influencés par l’action des complexes Polycomb. Les complexes Polycomb sont impliqués dans la détermination cellulaire et pourraient donc avoir été employés pour mettre en place un lignage de type « annexe embryonnaire » au cours de l’évolution. La restriction de l’empreinte au lignage de l’albumen chez les plantes, et le rôle prépondérant de l’empreinte dans le développement placentaire suggèrent un lien entre les mécanismes de l’empreinte et ceux spécifiant le destin cellulaire vers la voie « annexe embryonnaire ». Il est ainsi possible qu’une régulation par les complexes polycomb initialement destinée au contrôle du développement, ait été détournée pour la mise en place de l’empreinte. [9].
-
Chez les plantes et les mammifères, la dépendance complète de l’embryon vis-à-vis de sa mère provoque un déséquilibre entre les contributions des deux parents à la reproduction. De plus la présence dans une même portée d’embryons de pères différents entraîne une compétition pour les ressources maternelles entre embryons. Le déséquilibre de la participation des deux parents créerait donc un conflit parental entre une mère recherchant le développement de tous les embryons tandis que le père ne favorise que ceux qui portent son génome. Ce conflit parental ainsi créé se serait traduit par la sélection de mécanismes moléculaires orchestrant une expression non symétrique de certains gènes entre les deux parents, typique de celle des gènes soumis à empreinte [
24,
25]. Quelle que soit l’origine évolutive précise, il y a un lien probable entre la sélection de l’empreinte et la compétition dans une même portée ou un même fruit d’embryons provenant de plusieurs mâles. Quand cette compétition n’est plus présente, comme chez l’homme ou Arabidopsis, le nombre de gènes soumis à empreinte et la pression de sélection diminuent [
26,
27]. Chez Arabidopsis l’impact de l’empreinte est réduit au point qu’il est possible d’obtenir des graines viables en l’absence d’empreinte dans l’albumen [23].
|
Impacts biotechnologiques La similitude des mécanismes de l’empreinte chez les plantes et les mammifères permet d’envisager un échange d’informations entre les deux champs de recherches. L’impact des avancées obtenues chez les plantes sur la compréhension des mécanismes à l’œuvre chez les animaux peuvent donc avoir un impact biomédical et vétérinaire. Chez les plantes la fonction de l’empreinte dans l’albumen a un impact biotechnologique direct. L’albumen est le lieu de stockage des réserves de la graine dans les céréales. Un grain de riz, de maïs ou de blé est constitué d’albumen. De plus dans toutes les espèces, la taille de la graine est définie par la taille de l’albumen qui est contrôlée par les gènes soumis à empreinte. La manipulation génétique et la découverte de traits naturels associés à l’empreinte sont donc essentielles pour l’amélioration des rendements des productions céréalières. |
PEJ et FB sont financés par Temasek LifeSciences Laboratory. Les auteurs déclarent une absence de conflit d’intérêt.
|
1. Jullien PE, Kinoshita T, Ohad N, Berger F. Maintenance of DNA methylation during the Arabidopsis life cycle is essential for parental imprinting. Plant Cell 2006; 18 : 1360–72. 2. Kinoshita T, Miura A, Choi Y, et al. One-way control of FWA imprinting in Arabidopsis endosperm by DNA methylation. Science 2004; 303 : 521–3. 3. Hermon P, Srilunchang KO, Zou J, et al. Activation of the imprinted Polycomb Group Fie1 gene in maize endosperm requires demethylation of the maternal allele. Plant Mol Biol 2007; 64 : 387–95. 4. Gutierrez-Marcos JF, Costa LM, Dal Pra M, et al. Epigenetic asymmetry of imprinted genes in plant gametes. Nat Genet 2006; 38 : 876–8. 5. Kinoshita Y, Saze H, Kinoshita T, et al. Control of FWA gene silencing in Arabidopsis thaliana by SINE-related direct repeats. Plant J 2006; 49 : 38–45. 6. Finnegan EJ, Peacock WJ, Dennis ES. Reduced DNA methylation in Arabidopsis thaliana results in abnormal plant development. Proc Natl Acad Sci USA 1996; 93 : 8449–54. 7. Saze H, Scheid OM, Paszkowski J. Maintenance of CpG methylation is essential for epigenetic inheritance during plant gametogenesis. Nat Genet 2003; 34 : 65–9. 8. Morgan HD, Santos F, Green K, et al. Epigenetic reprogramming in mammals. Hum Mol Genet 2001; 14 (suppl 1) : 47–58. 9. Feil R, Berger F. Convergent evolution of genomic imprinting in plants and mammals. Trends Genet 2007; 23 : 192–9. 10. Gehring M, Choi Y, Fischer RL. Imprinting and seed development. Plant Cell 2006; 16 (suppl) : 203–13. 11. Gehring M, Huh JH, Hsieh TF, et al. DEMETER DNA glycosylase establishes MEDEA Polycomb gene self-imprinting by allele-specific demethylation. Cell 2006; 124 : 495–506. 12. Mathieu O, Reinders J, Caikovski M, et al. Transgenerational stability of the Arabidopsis epigenome Is coordinated by CG methylation. Cell 2007; 130 : 851–62. 13. Honys D, Twell D. Transcriptome analysis of haploid male gametophyte development in Arabidopsis. Genome Biol 2004; 5 : R85. 14. Guitton AE, Berger F. Control of reproduction by Polycomb group complexes in animals and plants. Int J Dev Biol 2005; 49 : 707–16. 15. Jullien PE, Katz A, Oliva M, et al. Polycomb Group complexes self-regulate imprinting of the polycomb group gene MEDEA in Arabidopsis. Curr Biol 2006; 16 : 486–92. 16. Makarevich G, Leroy O, Akinck U, et al. Different Polycomb group complexes regulate common target genes in Arabidopsis. EMBO J 2006; 7 : 947–52. 17. Makarevitch G, Villar CB, Erilova A, Kohler C. Mechanism of PHERES1 imprinting in Arabidopsis. J Cell Sci 2008; 121 : 906–12. 18. Umlauf D, Goto Y, Cao R, et al. Imprinting along the Kcnq1 domain on mouse chromosome 7 involves repressive histone methylation and recruitment of Polycomb group complexes. Nat Genet 2006; 36 : 1296–300. 19. Lewis A, Mitsuya K, Umlauf D, et al. Imprinting on distal chromosome 7 in the placenta involves repressive histone methylation independent of DNA methylation. Nat Genet 2004; 36 : 1291–5. 20. Scott RJ, Spielman M, Bailey J, Dickinson HG. Parent-of-origin effects on seed development in Arabidopsis thaliana. Development 1998; 125 : 3329–41. 21. Wagschal A, Feil R. Genomic imprinting in the placenta. Cytogenet Genome Res 2006; 13 : 90–8. 22. Guitton AE, Page DR, Chambrier P, et al. Identification of new members of fertilisation independent seed Polycomb group pathway involved in the control of seed development in Arabidopsis thaliana. Development 2004; 131 : 2971–81. 23. Nowack MK, Shirzadi R, Dissmeyer N, et al. Bypassing genomic imprinting allows seed development. Nature 2007; 447 : 312–5. 24. Spielman M, Vinkenoog R, Dickinson HG, Scott RJ. The epigenetic basis of gender in flowering plants and mammals. Trends Genet 2001; 17 : 705–11. 25. Haig D. Genomic imprinting and kinship: how good is the evidence ? Annu Rev Genet 2001; 38 : 553–85. 26. Spillane C, Schmid KJ, Laoueille-Duprat S, et al. Positive darwinian selection at the imprinted MEDEA locus in plants. Nature 2007; 448 : 349–52. 27. Monk D, Arnaud P, Apostolidou S, et al. Limited evolutionary conservation of imprinting in the human placenta. Proc Natl Acad Sci USA 2006; 103 : 6623–8. 28. Gabory A, Dandolo L. Épigénétique et développement : l’empreinte prentale. Med Sci (Paris) 2005; 21 : 390–5. 29. Prouteau M, Colot V. Contrôles épigénétiques, développement et variation génétique naturelle chez les plantes. Med Sci (Paris) 2005; 21 : 422–7. 30. Henckel A, Feil R. Asymétrie des génomes parentaux : implications en pathologie. Med Sci (Paris) 2008; 24 : 747–52. 31. Julien PE, Mosquna A, Ingouff M. et al. Retinoblastoma and its binding partner MSI1 control imprinting in Arabidopsis. Plos Biol 2008 (sous presse). |