| |
| Med Sci (Paris). 2008 August; 24(8-9): 742–746. Published online 2008 August 15. doi: 10.1051/medsci/20082489742.Les histones désacétylases Nouvelles cibles pour les thérapies anti-cancéreuses Denis Mottet and Vincent Castronovo* Laboratoire de Recherche sur les Métastases, GIGA-Cancer (CRCE), Université de Liège, Tour de Pathologie-1, Building B23, Sart Tilman, 4000 Liège, Belgique |
Dans les pays industrialisés, le cancer reste un problème de santé publique majeur. L’étude des causes biologiques du cancer s’est longtemps concentrée sur l’identification des altérations génétiques qui peuvent en être la source. Au cours de ces dernières années, il est devenu évident que l’initiation et la progression du cancer peuvent également dépendre de modifications de l’expression des gènes qui ne s’accompagnent pas de changements des séquences nucléotidiques et qui sont qualifiées d’épigénétiques. Les modifications épigénétiques de l’expression des gènes peuvent être associées à des modifications de l’ADN lui-même, comme la méthylation de résidus cytosine le plus souvent au niveau des séquences promotrices des gènes [
16] (→), ou par des modifications post-traductionnelles de molécules associées à l’ADN, comme les histones.
(→) Voir l’article de M. Weber, page 731 de ce numéro
|
Compaction de l’ADN dans le nucléosome La régulation de la transcription génique fait intervenir des modifications de l’état de compaction de l’ADN [
17] (→). Dans le noyau de la cellule, la molécule d’ADN n’existe pas, ou très peu, sous la forme d’une molécule libre. De manière répétée, elle s’associe et s’enroule de 146 paires de bases autour d’un octamère de quatre protéines appelées les histones (H4, H3, H2A et H2B) pour former le nucléosome. Les histones sont des petites protéines (12-16 kDa), extrêmement bien conservées au cours de l’évolution, composées de plus de 20 % de résidus dont la charge globale est positive (arginine ou lysine), faisant partie des protéines les plus basiques existantes. La présence de ces charges positives, notamment au niveau de leur extrémité amino-terminale, leur permet d’interagir avec le squelette phosphodiester de l’ADN chargé négativement. Ces interactions sont à la base du premier niveau de compaction de l’ADN : l’ADN nucléosomal [17]. À ce premier niveau de compaction, l’information génétique n’est déjà plus accessible. Pour contrecarrer cette répression physique avant d’amorcer la transcription, la cellule a développé des mécanismes lui permettant de modifier la structure de la chromatine. Complexes et encore largement incompris, les mécanismes de décondensation de la chromatine impliquent une série de modifications post-traductionnelles des histones qui fonctionneraient comme un code, le « code histones », autorisant ou non l’accès à l’information génétique. Ces modifications post-traductionnelles, covalentes et réversibles, touchent la queue amino-terminale des histones et sont principalement de quatre types : l’acétylation, la méthylation, la phosphorylation et l’ubiquitinylation. Parmi les modifications citées ci-dessus, l’acétylation des histone est probablement le mécanisme le plus étudié à ce jour. (→) Voir l’article de A. Bertin et S. Mangenot, page 715 de ce numéro
|
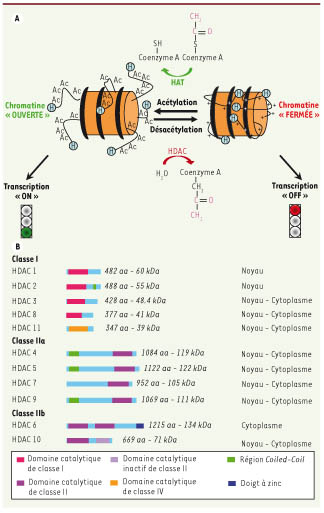
Degré d’acétylation des histones et accessibilité génique L’état d’acétylation des histones dépend de deux groupes d’enzymes aux activités opposées mais en équilibre permanent : les acétyltransférases d’histones (HAT) et les désacétylases d’histones (HDAC) qui ajoutent et enlèvent, respectivement, un groupement acétyle sur les lysines conservées des queues amino-terminales des histones. La charge portée par la lysine est annulée lorsqu’elle est acétylée et restaurée lorsqu’elle est désacétylée. Cette perte de charge augmente l’accessibilité à l’ADN en réduisant sa compaction et donc finalement favorise la transcription (Figure 1A). À ce jour, 18 HDAC humaines ont été identifiées. Elles sont réparties en 3 classes sur la base de leur homologie de séquence avec différentes HDAC de la levure, de leur poids moléculaire et de leur localisation intracellulaire (Figure 1B) [
1,
2].  | Figure 1.
Représentation schématique de l’accessibilité à l’ADN en fonction de l’acétylation des queues amino-terminales des histones (A) et résumé des principales caractéristiques des trois classes d’HDAC (B).
|
Le degré d’acétylation des histones assuré par les HAT et les HDAC joue ainsi un rôle clé dans le contrôle de la transcription génique. Des modifications de l’expression et/ou de l’activité des HDAC ont été identifiées dans des cellules tumorales et la rupture de l’équilibre entre les niveaux d’acétylation et de désacétylation de la chromatine contribue aux altérations de l’expression des gènes retrouvées dans de nombreuses tumeurs [
3–
5]. Le réajustement de cet équilibre HDAC/HAT représente une stratégie anti-tumorale séduisante et a conduit rapidement au développement d’une famille de molécules capables d’inhiber les HDAC. |
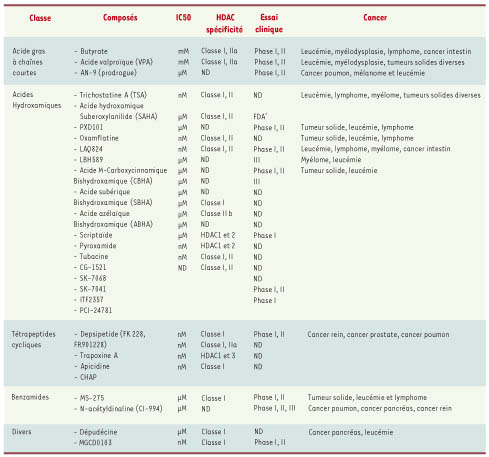
Les inhibiteurs des désacétylases d’histones en thérapeutique anticancéreuse Les inhibiteurs d’HDAC (HDACi) sont classés en quatre grandes familles chimiques en fonction de leur structure : les acides gras à chaîne courte (dérivés de l’acide butyrique), les dérivés d’acide hydroxamique (SAHA, LAQ824, LBH589…), les benzamides (CI994, MS275) et les tétrapeptides cycliques (FK228, FR901228). Tous ces composés sont capables d’inhiber plus ou moins sélectivement les HDAC de classe I et II [3,
4,
6]. Ils présentent une large gamme d’effets sur des cellules en culture et dans des modèles animaux. Ces effets incluent l’activation ou la répression spécifique de gènes impliqués dans de nombreux processus biologiques tels que la prolifération, la différenciation, la sénescence et l’apoptose [3, 5,
7]. Leur effet drastique in vitro a rapidement conduit à tester leur effet clinique dans différents types de tumeurs solides et hématologiques. À ce jour, au moins 14 inhibiteurs, administrés seuls ou en combinaison avec d’autres molécules chimiothérapeutiques, sont en phase avancée de développement clinique (y compris Phase III) et montrent une activité anti-tumorale intéressante à des doses relativement bien tolérées par les patients [
8–
10] (Tableau I). Même si certaines de ces molécules méritent assurément un développement supplémentaire concernant notamment les doses et modes d’administration, tous les inhibiteurs d’HDAC testés présentent une activité clinique au moins partielle témoignant bien de leur pouvoir anti-cancéreux. Cependant, beaucoup de questions devront être résolues avant de connaître l’intérêt thérapeutique réel de ces inhibiteurs.  | Tableau I.
Différents groupes d’inhibiteurs d’HDAC utilisés actuellement en phase clinique.
|
Un des problèmes potentiels de l’utilisation des HDACi actuellement étudiés est leur non sélectivité car ils ciblent aussi bien les HDAC de classe I que celles de classe II. D’ailleurs, leurs propriétés anti-tumorales se manifestent à travers plusieurs activités sans distinction spécifique de(s) HDAC responsable(s) de l’effet observé. Dans un proche avenir, il faudra définir plus finement les relations entre l’inhibition spécifique d’une ou de plusieurs HDAC et les effets anti-tumoraux observés afin de développer une nouvelle génération d’inhibiteurs d’histone-désacétylases potentiellement plus sélectifs et dotés de moins d’effets secondaires. |
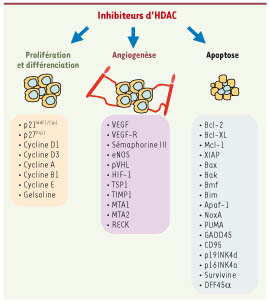
L’action des HDAC ne se restreint pas aux modifications épigénétiques des histones Il est de plus en plus évident que les modifications épigénétiques des histones ne sont pas les seuls mécanismes par lesquels les HDACi agissent sur les cellules cancéreuses. Un nombre croissant d’études indiquent en effet que le spectre d’activité des HDAC s’étend bien au-delà de la simple désacéylation des histones (Figure 2). La découverte de nombreux substrats non histoniques des HDAC permet d’envisager que les effets anti-tumoraux que les HDACi soient également liés à des mécanismes dépendants du niveau d’acétylation de protéines tels que des facteurs de transcription [
11].  | Figure 2.
Schéma récapitulatif des fonctions et gènes modulés par les inhibiteurs d’HDAC dans des cellules en culture et dans des modèles animaux. La liste de gènes n’est pas exhaustive. |
Récemment, il a été proposé que l’action des HDACi pourrait s’exercer de façon indépendante de la transcription génique. Ainsi, l’un des modes d’action des inhibiteurs des HDAC serait associé, au moins en partie, à l’accumulation d’ADN endommagé qui, non réparé, entraîne la mort des cellules cancéreuses. La vulnérabilité des cellules tumorales face aux HDACi serait due à leur incapacité à activer deux mécanismes de surveillance présents dans les cellules normales. Le premier est le point de contrôle en phase G2 du cycle cellulaire (G2-phase checkpoint), qui intervient dans la transition G2-M et se déclenche après la réplication de l’ADN pour empêcher la cellule d’entrer dans une mitose aberrante. Le second mécanisme est le point de contrôle du fuseau mitotique (mitotic-spindle checkpoint), qui détecte l’alignement incorrect des chromosomes sur le fuseau mitotique et arrête le cycle cellulaire en métaphase jusqu’à ce que ceux-ci s’attachent de façon adéquate. La perturbation de ces deux mécanismes aboutit à une mitose avortée et à l’apoptose de la cellule [10,
12,
13]. Un autre facteur de complexité dans la compréhension de la biologie des HDAC réside dans l’élucidation du mécanisme exact par lequel les HDACi induisent sélectivement l’arrêt de la croissance cellulaire, la différenciation et l’apoptose des cellules cancéreuses. Toutes les cellules, normales ou malignes, utilisent les HDAC au cours des différentes étapes du cycle cellulaire. Pourquoi les cellules cancéreuses sont-elles plus sensibles aux HDACi que les cellules normales ? Un élément de réponse pourrait être en relation avec la libération d’espèces réactives de l’oxygène (ROS, reactive oxygen species). En effet, contrairement aux cellules cancéreuses, après traitement par les inhibiteurs d’HDAC, les cellules normales accumulent la thiorédoxine (TRX), un capteur de radicaux libres. Ainsi, les ROS produites à la suite de ce traitement sont neutralisées dans les cellules normales par la TRX, alors que les cellules cancéreuses meurent en raison de l’inactivation de leur TRX par sa fixation à la protéine TBP-2 surexprimée en présence d’HDACi [10,
14]. Quoiqu’il en soit, l’implication des HDACi dans l’induction de la mort des cellules cancéreuses et non celle des cellules saines est un point important à souligner puisqu’il individualise certainement cette classe de médicaments et la rend très prometteuse comparée aux drogues conventionnelles. Malgré les nombreux progrès qu’il reste à accomplir dans la caractérisation des HDAC et de leur inhibition sélective ou non, il ne fait aucun doute aujourd’hui que la modulation de l’activité de ces enzymes ouvre une ère innovante pour le développement de nouvelles stratégies anti-cancéreuses. Dans le cadre de cet article, nous n’avons pas discuté des sirtuines, le groupe III des HDAC, dont l’activité dépend de l’adénine dinucléotide nicotinamide (NAD+), n’est pas affectée par les inhibiteurs des HDAC de classe I et II mais présentent néanmoins un intérêt [
15]. |
1. De Ruijter AJ, van Gennip AH, Caron HN, et al. Histone deacetylases (HDACs): characterization of the classical HDAC family. Biochem J 2003; 370 : 737–49. 2. Marks PA, Miller T, Richon VM. Histone deacetylases. Curr Opin Pharmacol 2003; 3 : 344–51. 3. Bolden JE, Peart MJ, Johnstone RW. Anticancer activities of histone deacetylase inhibitors. Nat Rev Drug Discov 2006; 5 : 769–84. 4. Dokmanovic M, Clarke C, Marks PA. Histone deacetylase inhibitors: overview and perspectives. Mol Cancer Res 2007; 5 : 981–9. 5. Marks PA, Richon VM, Breslow R, Rifkind RA. Histone deacetylase inhibitors as new cancer drugs. Curr Opin Oncol 2001; 13 : 477–83. 6. Dokmanovic M, Marks PA. Prospects: histone deacetylase inhibitors. J Cell Biochem 2005; 96 : 293–304. 7. Johnstone RW. Histone-deacetylase inhibitors: novel drugs for the treatment of cancer. Nat Rev Drug Discov 2002; 1 : 287–99. 8. Piekarz RL, Sackett DL, Bates SE. Histone deacetylase inhibitors and demethylating agents: clinical development of histone deacetylase inhibitors for cancer therapy. Cancer J 2007; 13 : 30–9. 9. Rasheed WK, Johnstone RW, Prince HM. Histone deacetylase inhibitors in cancer therapy. Expert Opin Investig Drugs 2007; 16 : 659–78. 10. Xu WS, Parmigiani RB, Marks PA. Histone deacetylase inhibitors: molecular mechanisms of action. Oncogene 2007; 26 : 5541–52. 11. Glozak MA, Sengupta N, Zhang X, Seto E. Acetylation and deacetylation of non-histone proteins. Gene 2005; 363 : 15–23. 12. Magnaghi-Jaulin L, Eot-Houllier G, Fulcrand G, Jaulin C. Histone deacetylase inhibitors induce premature sister chromatid separation and override the mitotic spindle assembly checkpoint. Cancer Res 2007; 67 : 6360–7. 13. Qiu JW. Role of thioredoxin in the response of normal and transformed cells to histone deacetylase inhibitors. Proc Natl Acad Sci USA 2005; 102 : 673–8. 14. Ungerstedt JS, Sowa Y, Xu WS, et al. Role of thioredoxin in the response of normal and transformed cells to histone deacetylase inhibitors. Proc Natl Acad Sci USA 2005; 102 : 673–8. 15. Saunders LR, Verdin E. Sirtuins: critical regulators at the crossroads between cancer and aging. Oncogene 2007; 26 : 5489–504. 16. Weber M. Profils de méthylation de l’ADN dans les cellules normales et cancéreuses. Med Sci (Paris) 2008; 24 : 731–4. 17. Bertin A, Mangenot S. Structure et dynamique de la particule cœur de nucléosome. Med Sci (Paris) 2008; 24 : 715–9. |