| |
| Med Sci (Paris). 2008 June; 24(6-7): 599–606. Published online 2008 June 15. doi: 10.1051/medsci/20082467599.Classification pronostique du cancer du sein et profils d’expression génique sur puces à ADN François Bertucci,1,2,3,4* Pascal Finetti,2,3,4,5 Nathalie Cervera,2,3,4,5 and Daniel Birnbaum2,3,4,5 1Centre de Recherche en Cancérologie de Marseille, Département d’Oncologie Médicale 2UMR599 Inserm 3Institut Paoli-Calmettes 4Université de la Méditerranée, 232, boulevard Sainte-Marguerite, 13273 Marseille Cedex 09, France 5Centre de Recherche en Cancérologie de Marseille, Département d’Oncologie Moléculaire |
Le cancer du sein est le cancer le plus fréquent et le plus meurtrier chez la femme en France. Les traitements systémiques (chimiothérapie, hormonothérapie, thérapies moléculaires ciblées) censés éradiquer la maladie métastatique clinique ou infra-clinique, sont délivrés en fonction de facteurs pronostiques et prédictifs de la réponse thérapeutique. Malheureusement, ceux-ci rendent insuffisamment compte de l’hétérogénéité évolutive de la maladie, conduisant trop souvent à des traitements inadaptés. Cette inadaptation coïncidant avec la disponibilité croissante de nouvelles drogues anti-cancéreuses, il est crucial d’améliorer la classification pronostique pour affiner les indications thérapeutiques et améliorer la survie. Le développement de thérapies moléculaires ciblées comme le trastuzumab (Herceptine®), anticorps monoclonal ciblant ERBB2 surexprimé dans ~20 % des cancers du sein, est un autre enjeu majeur [
48]. Face aux limites des approches classiques, les espoirs se sont tournés vers une caractérisation moléculaire globale, détaillée et objective de la maladie à l’aide des analyses à haut débit, notamment l’analyse du transcriptome sur puces à ADN [
1]. Établis en parallèle pour plusieurs tumeurs, les portraits moléculaires transcriptionnels sont comparés à l’aide d’outils bio-informatiques puissants. On peut distinguer deux approches d’analyse des données qui se sont succédées dans le temps. L’approche « différentielle », la plus ancienne mais encore d’actualité, consiste à établir une liste de gènes différentiellement exprimés entre deux conditions. L’approche « profils d’expression » cherche à identifier une signature moléculaire multigénique dont l’expression combinée est caractéristique d’une classe pertinente d’échantillons (survie, réponse thérapeutique par exemple). Cette approche peut se faire de façon non supervisée ou supervisée. Les applications potentielles en cancérologie sont multiples [
2]. Plusieurs publications suggèrent un impact pronostique dans le cancer du sein. |
Taxonomie moléculaire pronostique : approches non supervisées Les analyses non supervisées, clustering ou classification hiérarchique par exemple, définissent des classes de tumeurs sur la base de la similitude des profils d’expression, indépendamment de toute donnée histologique ou clinique a priori. Une fois les classes définies, des corrélations sont recherchées entre classes de tumeurs et paramètres histo-cliniques (survie par exemple). C’est par cette approche, appliquée à un jeu de 496 gènes sélectionnés parmi 8 000 testés et une série de cancers du sein localement avancés, qu’une équipe de Stanford a défini 5 sous-types de tumeurs différents d’un point de vue transcriptionnel et pronostique [
3,
4]. Ces sous-types étaient reliés au type cellulaire d’origine et à l’immunohistochimie [
49]. Les tumeurs négatives pour l’expression du récepteur des œstrogènes (RE) étaient divisées en trois sous-types : l’un exprimait un profil d’expression proche de celui des cellules épithéliales basales, un deuxième surexprimait un groupe de gènes dont ERBB2 et le troisième avait un profil proche de celui du tissu mammaire normal. Les tumeurs RE+ présentaient un profil d’expression proche de celui décrit pour les cellules épithéliales luminales et étaient divisées en deux sous-types (luminal A et B), que distinguait une survie différente des patientes. Ces sous-types et leurs corrélations histo-cliniques ont été validés par la même équipe sur un nombre plus important de tumeurs [
5,
6], puis ont été identifiés dans des cancers in situ [
7], et dans le cancer du sein inflammatoire [
8], renforçant leur robustesse et leur universalité. Ces sous-types se distinguent aussi par leur taux de réponse complète sur le plan histologique à la chimiothérapie, une sensibilité plus importante étant observée pour les tumeurs basales et ERBB2+ que pour les tumeurs luminales [8,
9]. |
Signatures moléculaires identifiées par analyses supervisées sans hypothèse biologique de départ : la pêche à la ligne Les analyses non supervisées séparent rarement d’emblée des classes pronostiques d’échantillons. On utilise alors des analyses supervisées qui cherchent à identifier les gènes discriminant deux classes de tumeurs d’évolution différente. L’objectif est de définir un modèle multigénique qui prédira de façon correcte l’appartenance d’un échantillon à l’une des classes. Cela se fait en deux phases successives : apprentissage (identification du modèle) et validation (estimation de sa robustesse), réalisées idéalement sur des tumeurs indépendantes. Cette approche a été appliquée pour prédire la survie et la réponse thérapeutique. |
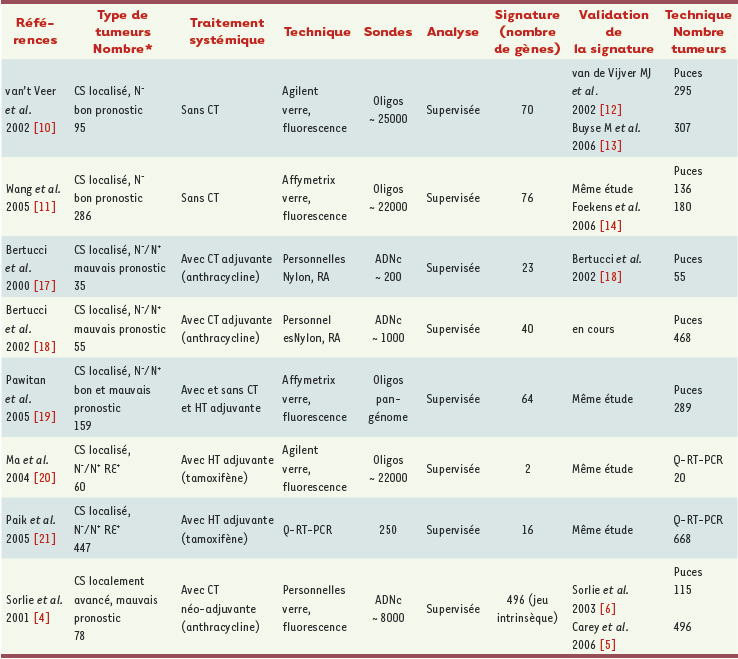
Signatures moléculaires et survie La recherche de signatures associées à la survie, que le patient ait ou non reçu un traitement systémique adjuvant, s’est faite sur la tumeur réséquée lors de la chirurgie initiale (contexte adjuvant) ou sur une biopsie tumorale (contexte néo-adjuvant) avant tout traitement systémique (Tableau I).  | Tableau I.
Profils d’expression génique du cancer du sein et survie. Nombre* : nombre de tumeurs du jeu d’apprentissage ; CS : cancer du sein ; CT : chimiothérapie ; HT : hormonothérapie ; RA : radioactivité ; N−/N+ : sans ou avec envahissement ganglionnaire ; RE : récepteur des œstrogènes. |
Survie sans chimiothérapie adjuvante Deux équipes hollandaises ont abordé la question du sur-traitement par chimiothérapie adjuvante des tumeurs localisées sans envahissement ganglionnaire (N -) [
10,
11]. Dans ces tumeurs, l’impact favorable de la chimiothérapie sur la survie est démontré. Mais, en raison de l’insuffisance des facteurs pronostiques et du faible bénéfice de la chimiothérapie, la majorité des patientes reçoit une chimiothérapie adjuvante, alors qu’elle ne serait en réalité nécessaire que chez 20 % d’entre elles. Quatre-vingt pour cent des patientes sont donc sur-traitées. L’équipe d’Amsterdam [ 10] a identifié, en étudiant une série de 97 patients n’ayant pas été traités par chimiothérapie, une signature pronostique de 70 gènes. Cette signature a été validée sur une série unicentrique de 295 tumeurs [
12], puis sur une série multicentrique de 307 tumeurs [
13]. L’équipe de Rotterdam a identifié sur 286 tumeurs N - [ 11] une signature de 76 gènes (60 pour les tumeurs RE +, 16 pour les RE -), validée ensuite sur 136 tumeurs, puis sur une série multicentrique de 180 cas [
14]. Comme l’étude précédente, elle a été récemment validée sur la série multicentrique européenne de 307 tumeurs [
15]. Dans les deux études, le nombre de patientes candidates à une chimiothérapie adjuvante selon les critères consensuels de St-Gallen et du NIH (National Institute of Health) est largement supérieur à celui que prédit la signature moléculaire (ou prédicteur multigénique) citée ci-dessus, notamment chez les patientes n’ayant pas rechuté. La signature d’Amsterdam est aujourd’hui en phase ultime de validation prospective dans l’essai MINDACT (microarray in node-negative disease may avoid chemotherapy trial) de l’EORTC (European oganisation for research and treatment of cancer), ouvert en 2007. Son objectif est de démontrer que la signature permettra de traiter par chimiothérapie moins de patientes qu’un critère histo-clinique classique avec une survie pourtant similaire. La société hollandaise Agendia commercialise déjà un test (Mammaprint). Il s’agit d’une puce à oligos fabriquée par Agilent contenant les 70 gènes de la signature (trois repliquats) associés à des gènes de normalisation et des contrôles. L’échantillon tumoral congelé est adressé à Agendia qui réalise le test. Les résultats (faible ou haut risque de rechute) sont donnés en moins de 10 jours [
50]. Survie avec chimiothérapie adjuvante L’objectif est différent et complémentaire : identifier chez les patientes traitées par chimiothérapie adjuvante à base d’anthracycline (protocole de référence des 20 dernières années) une signature prédisant la rechute métastatique. L’efficacité inconstante et la toxicité des protocoles, leur nombre de plus en plus grand en raison de l’arrivée de nouvelles drogues - le plus récent étant l’association anthracycline-taxane chez les patientes avec envahissement ganglionnaire (N +) - et l’absence de facteurs prédictifs d’efficacité rendent nécessaire l’identification des patients pouvant bénéficier ou non d’un régime donné de chimiothérapie [
16]. Cela permettrait un choix rationnel entre ces différents régimes, évitant l’escalade thérapeutique parfois inutile, mais toujours coûteuse. Des profils d’expression ont été associés à la survie après chimiothérapie adjuvante avec anthracycline [
17,
18] et sans anthracycline [
19]. Nous avons analysé une série rétrospective multicentrique de 468 cancers traités par chimiothérapie adjuvante à base d’anthracyclines (Bertucci et al., en préparation). Nous avons identifié un modèle multigénique associé à la rechute métastatique sur 323 tumeurs, ensuite validé sur 145 tumeurs indépendantes. En analyse multivariée, ce modèle est un facteur pronostique fort et indépendant. Si les résultats se confirment, il permettra de choisir le protocole optimal de chimiothérapie adjuvante chez des patientes pour qui l’indication est posée. Survie avec hormonothérapie adjuvante Le tamoxifène, un anti-œstrogène, est un traitement majeur du cancer du sein lorsque les cellules expriment les récepteurs hormonaux (RH : RE et RP récepteur de la progestérone). Ma et al. ont identifié sur 60 cancers RE + traités par tamoxifène [
20] deux gènes associés à la rechute métastatique, HOXB13 et IL17BR, dont le ratio d’expression était un facteur pronostique fort et indépendant. Paik et al. ont travaillé par RT-PCR quantitative sur des ARN extraits de tumeurs incluses en paraffine [
21]. Ils ont analysé 250 gènes candidats sur 447 tumeurs traitées par tamoxifène en adjuvant, et identifié 16 gènes associés à la rechute métastatique. Combinés à 5 témoins, ces gènes définissent un score de rechute qui classe les patients en 3 niveaux de risque : faible, intermédiaire et élevé. Ce score a été validé sur 668 tumeurs indépendantes N - RE +. Un test (Oncotype DX), est commercialisé aux États-unis par Genomic Health (Redwood City, CA). L’échantillon tumoral en paraffine est adressé à Genomic Health qui réalise le test. Il s’agit d’une RT-PCR quantitative type TaqMan qui mesure l’expression des 16 gènes (trois répliquats) normalisée par les 5 gènes de référence. Le résultat est rendu sous la forme d’appréciation du risque - faible, intermédiaire ou élevé - de rechute à 10 ans. Ce test est actuellement incorporé dans un essai clinique prospectif lancé par le NIH, TAILORx ( Trial assigning individualized options for treatment [Rx]) chez les patientes N - RE +. |
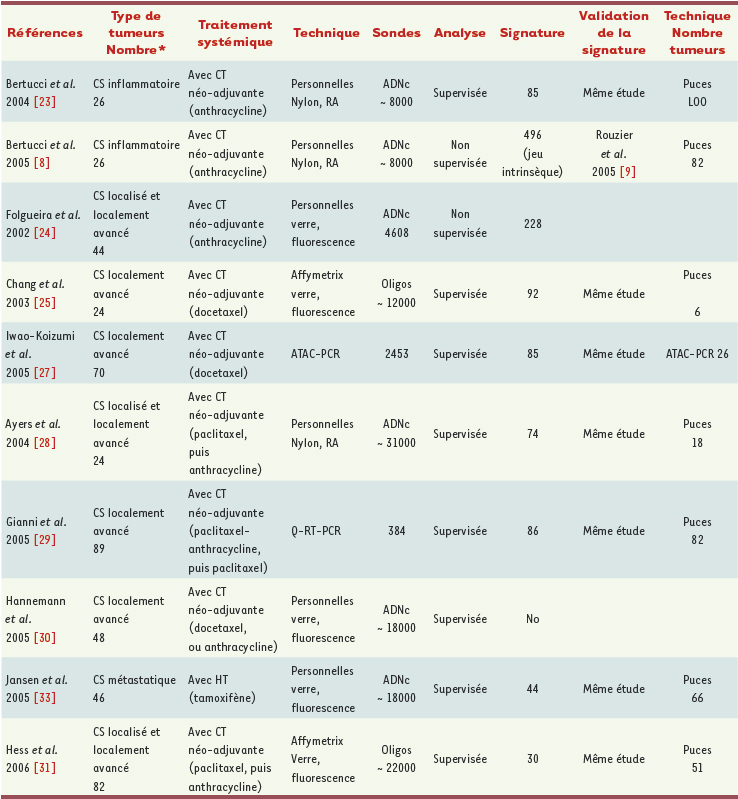
Signatures moléculaires et réponse aux traitements systémiques L’identification des patientes ayant un risque de rechute et nécessitant un traitement systémique adjuvant doit être complétée par l’identification des patientes susceptibles de répondre le mieux à un traitement donné. Bien qu’imparfaite, la réponse clinique ou histologique aux traitements systémiques est un facteur pronostique favorable [
22]. Plusieurs études ont abordé le thème de la résistance/sensibilité à la chimiothérapie ou à l’hormonothérapie en confrontant les profils d’expression à la réponse thérapeutique (Tableau II).  | Tableau II.
Profils d’expression génique du cancer du sein et réponse au traitement systémique. Nombre* : nombre de tumeurs du jeu d’apprentissage ; LOO : leave-one-out cross-validation. |
Réponse à la chimiothérapie Aujourd’hui, aucun paramètre ne prédit de façon fiable la réponse à un régime donné de chimiothérapie néo-adjuvante, conduisant toutes les patientes à recevoir le même protocole. Le contexte du néo-adjuvant est propice à l’identification de tels marqueurs car le critère de jugement (réponse thérapeutique) est rapidement atteint (après 4 ou 6 cures de chimiothérapie). Il permet de comparer les profils moléculaires avant, pendant et après traitement. Trois études ont concerné une chimiothérapie à base d’anthracycline [8,
23,
24]. Nous avons identifié dans le cancer du sein inflammatoire [23] une signature de 85 gènes définissant deux groupes de patientes, ayant respectivement 70 % et 0 % de réponse complète histologique après chimiothérapie. Trois études ont concerné une chimiothérapie à base de docetaxel (classe des taxanes) [
25–
27]. L’équipe du Baylor College de Houston [25] a identifié sur 24 tumeurs localement avancées un jeu de 92 gènes dont l’expression corrélait à la réponse clinique. Cinq études ont été réalisées autour de l’association anthracycline-taxane [9,
28–
31]. Hess et al. ont rapporté la plus grande série avec 133 patientes traitées par paclitaxel hebdomadaire suivi d’un protocole à base d’anthracycline [31]. La méthode d’analyse est intéressante. Après avoir ordonné les gènes en fonction de la corrélation de leur expression à la réponse histologique (complète versus non complète) dans le jeu d’apprentissage (82 tumeurs), et dans la mesure où aucun consensus n’existe sur la meilleure façon d’identifier un prédicteur, les auteurs ont testé 780 prédicteurs différents représentant 20 méthodes de classification sur 39 jeux de gènes allant de 1 à 22 823 gènes. La performance de chaque prédicteur était évaluée par courbes ROC (receiver operating characteristic) en validation croisée. Le prédicteur optimal contenait 30 gènes, et a été validé sur 51 tumeurs indépendantes avec une sensibilité supérieure à celle d’un prédicteur clinique. Réponse à l’hormonothérapie L’expression protéique des récepteurs hormonaux (RH) est le meilleur prédicteur de réponse à l’hormonothérapie. Mais la corrélation est imparfaite. Au stade métastatique, une réponse au tamoxifène est rapportée chez environ 50 % des patientes RE +, mais aussi 5-10 % des patientes RE -. Après une réponse initiale, la plupart des patientes développent une résistance [
32]. L’identification de marqueurs fiables de réponse et une compréhension des phénomènes de résistance sont cruciales. L’analyse de 112 tumeurs RE + issues de patientes présentant une rechute métastatique traitée par tamoxifène (52 sensibles, 60 résistantes) [
33] a identifié une signature de 44 gènes qui prédisait mieux que les RH la survie sans progression. |
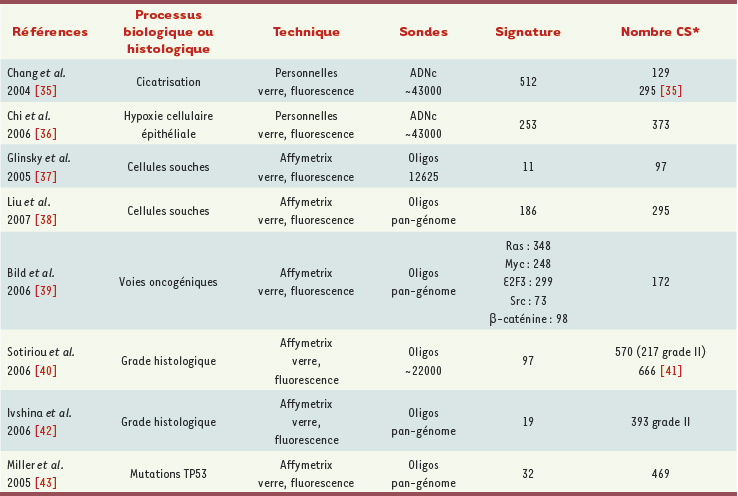
Signatures moléculaires identifiées par analyses supervisées fondées sur une hypothèse biologique Une troisième approche, plus récente, est fondée sur une hypothèse biologique. Elle consiste à identifier de manière supervisée à partir d’un modèle expérimental ou d’échantillons tumoraux cliniques, une signature moléculaire associée à un processus biologique ou à un paramètre histologique ayant un sens dans l’évolution métastatique, puis à évaluer son impact pronostique sur des tumeurs (Tableau III).
 | Tableau III.
Signatures moléculaires basées sur une hypothèse biologique et impact pronostique dans le cancer du sein. CS : cancer du sein ; * : effectif tumoral dans lequel l’impact pronostique a été démontré. |
En se fondant sur les similitudes existant entre cicatrisation et métastase (deux processus impliquant angiogenèse, prolifération, migration), Chang et al. [
34,
35] ont identifié à partir de fibroblastes stimulés par du sérum, une signature « de cicatrisation » ayant un impact pronostique dans plusieurs séries de cancers du sein. Les patients dont les tumeurs expriment la signature de cicatrisation ont une survie défavorable. La même approche a été réalisée avec des signatures de réponse à l’hypoxie cellulaire épithéliale [
36], de cellules souches cancéreuses mammaires [
37,
38], ou de l’activation de voies oncogéniques [
39]. Des signatures moléculaires associées à des facteurs pronostiques connus tels que le grade histologique [
40–
42] ou suspectés, comme le statut des mutations de TP53 [
43], présentent un impact pronostique. Sotiriou et al. ont identifié une signature de 97 gènes différentiellement exprimés entre tumeurs de grade I et de grade III. La majorité des gènes surexprimés dans les tumeurs de haut grade sont impliqués dans la progression du cycle cellulaire et la prolifération. Cette signature sépare les tumeurs de grade intermédiaire II en deux groupes de survie différente. |
Conclusion et perspectives Plusieurs études rétrospectives ont identifié des signatures pronostiques définissant de nouvelles classes du cancer du sein dans des groupes de tumeurs d’apparence histo-clinique homogène, mais hétérogènes sur le plan évolutif. Même si plusieurs études présentent encore des imperfections qu’il faudra éviter à l’avenir [
44], les résultats sont encourageants. Les signatures de prédiction de la réponse thérapeutique sont actuellement les moins avancées avec plusieurs faiblesses méthodologiques : effectif tumoral faible, validation indépendante rare, traitements hétérogènes. Ces résultats prometteurs révèlent une réalité qui peut sembler déconcertante : le faible chevauchement des signatures multigéniques décrites, que les plus sceptiques ne manquent pas de souligner [
51]. La fiabilité et la reproductibilité des puces à ADN ne sont plus en cause après les résultats du projet Microarray Quality Control (MAQC) réalisé sous l’égide de la FDA [
45]. En réalité, des différences méthodologiques à de multiples niveaux expliquent en partie ce faible chevauchement entre les études : tumeurs analysées, formes cliniques représentées, traitements systémiques délivrés, plates-formes de puces à ADN, jeux de gènes testés, erreurs d’annotation des sondes, méthodes d’hybridations, de pré-traitement et surtout d’analyse des données, critères de jugement, seuils statistiques différents… Mais les raisons majeures sont conceptuelles et méthodologiques. La complexité et l’hétérogénéité moléculaire des cancers du sein font que plusieurs voies sont dérégulées en même temps et que pour aboutir à un même effet biologique et à la progression, ce ne sont pas toujours les mêmes gènes qui sont touchés, mais parfois des gènes différents appartenant pourtant à une même voie de signalisation. Au niveau méthodologique, les fréquentes corrélations d’expression des gènes font que plusieurs gènes co-exprimés corrèlent au devenir clinique. Le pouvoir statistique appliqué à la sélection des gènes discriminants est souvent limité en raison de la faiblesse des effectifs tumoraux, qui coïncide fréquemment avec la faiblesse de la corrélation entre expression et devenir clinique, corrélation qui elle-même varie d’un jeu d’apprentissage à un autre. Pour ces raisons, les listes de gènes obtenues sont instables et le chevauchement entre signatures est faible. Ein-Dor et al. [
46] ont ainsi démontré sur le jeu de données d’Amsterdam [10] qu’il existait plusieurs signatures différentes à 70 gènes avec le même impact pronostique. L’information contenue dans ces signatures divergentes est donc très similaire. Cela a été confirmé par l’équipe de Perou par l’approche inverse [
47]. Les auteurs ont appliqué cinq signatures publiées différentes au même jeu de données publiques et montré que les classifications résultantes étaient similaires en termes de répartition des tumeurs et d’évaluation pronostique. Ces signatures contiennent donc la même information pronostique et reflètent probablement des voies moléculaires sous-jacentes communes, confirmant l’idée qu’Il n’existe pas une seule signature pronostique, mais plusieurs signatures très proches. En réalité, l’objectif n’est pas d’identifier « la meilleure signature », mais d’améliorer les classifications pronostiques actuelles. Des études de validation sur de plus grandes séries rétrospectives sont en cours. Le succès de ces études dépendra de la volonté réelle et de collaborations étroites entre chercheurs de différentes disciplines (cliniciens, anatomopathologistes, biologistes, bio-informaticiens, mathématiciens…), de la qualité et la quantité des tumeurs disponibles, de l’amélioration des outils d’analyse, et bien sûr du bon design des essais. Des recommandations concernant le design et l’analyse statistique des études pronostiques ont été suggérées [44], et la FDA a lancé la phase 2 du projet MAQC dans ce but. Concernant l’application clinique des signatures, la FDA a déjà approuvé l’utilisation de deux signatures sous la forme des tests Mammaprint et Oncotype. Mais pour atteindre un niveau de preuve suffisant et optimal quant à leur utilité réelle, ces signatures sont actuellement testées dans deux essais cliniques prospectifs : MINDACT et TAILORx. Un objectif complémentaire, encore peu approfondi, est de trouver un sens biologique aux signatures pronostiques afin de révéler les différentes voies moléculaires à cibler par de nouvelles thérapies. Parmi toutes les signatures publiées, celle qui est associée à la prolifération cellulaire est probablement le dénominateur commun le plus fréquemment retrouvé. Les puces à ADN marquent certainement un tournant dans l’approche moléculaire du cancer du sein, offrant un outil sans précédent pour disséquer la complexité moléculaire des tumeurs, et probablement améliorer la prise en charge des patientes [50].

|
1. The Chipping Forecast. Nat Genet 1999; 21 (suppl) : 1–60. 2. Bertucci F, Houlgatte R, Nguyen C, et al. Gene expression profiling of cancer by use of DNA arrays : how far from the clinic ? Lancet Oncol 2001; 2 : 674–82. 3. Perou CM, Sorlie T, Eisen MB, et al. Molecular portraits of human breast tumours. Nature 2000; 406 : 747–52. 4. Sorlie T, Perou CM, Tibshirani R, et al. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications. Proc Natl Acad Sci USA 2001; 98 : 10869–74. 5. Carey LA, Perou CM, Livasy CA, et al. Race, breast cancer subtypes, and survival in the Carolina breast cancer study. JAMA 2006; 295 : 2492–502. 6. Sorlie T, Tibshirani R, Parker J, et al. Repeated observation of breast tumor subtypes in independent gene expression data sets. Proc Natl Acad Sci USA 2003; 100 : 8418–23. 7. Yu K, Lee CH, Tan PH, Tan P. Conservation of breast cancer molecular subtypes and transcriptional patterns of tumor progression across distinct ethnic populations. Clin Cancer Res 2004; 10 : 5508–17. 8. Bertucci F, Finetti P, Rougemont J, et al. Gene expression profiling identifies molecular subtypes of inflammatory breast cancer. Cancer Res 2005; 65 : 2170–8. 9. Rouzier R, Perou CM, Symmans WF, et al. Breast cancer molecular subtypes respond differently to preoperative chemotherapy. Clin Cancer Res 2005; 11 : 5678–85. 10. van ’t Veer LJ, Dai H, van de Vijver MJ, et al. Gene expression profiling predicts clinical outcome of breast cancer. Nature 2002; 415 : 530–6. 11. Wang Y, Klijn JG, Zhang Y, et al. Gene-expression profiles to predict distant metastasis of lymph-node-negative primary breast cancer. Lancet 2005; 365 : 671–9. 12. van de Vijver MJ, He YD, van’t Veer LJ, et al. A gene-expression signature as a predictor of survival in breast cancer. N Engl J Med 2002; 347 : 1999–2009. 13. Buyse M, Loi S, van’t Veer L, et al. Validation and clinical utility of a 70-gene prognostic signature for women with node-negative breast cancer. J Natl Cancer Inst 2006; 98 : 1183–92. 14. Foekens JA, Atkins D, Zhang Y, et al. Multicenter validation of a gene expression-based prognostic signature in lymph node-negative primary breast cancer. J Clin Oncol 2006; 24 : 1665–71. 15. Desmedt C, Piette F, Loi S, et al. Strong time dependence of the 76-gene prognostic signature for node-negative breast cancer patients in the TRANSBIG multicenter independent validation series. Clin Cancer Res 2007; 13 : 3207–14. 16. Piccart-Gebhart MJ. Moving away from the « one shoe fits all » strategy : the key to future progress in chemotherapy. J Clin Oncol 2005; 23 : 1611–3. 17. Bertucci F, Houlgatte R, Benziane A, et al. Gene expression profiling of primary breast carcinomas using arrays of candidate genes. Hum Mol Genet 2000; 9 : 2981–91. 18. Bertucci F, Nasser V, Granjeaud S, et al. Gene expression profiles of poor-prognosis primary breast cancer correlate with survival. Hum Mol Genet 2002; 11 : 863–72. 19. Pawitan Y, Bjohle J, Amler L, et al. Gene expression profiling spares early breast cancer patients from adjuvant therapy : derived and validated in two population-based cohorts. Breast Cancer Res 2005; 7 : R953–64. 20. Ma XJ, Wang Z, Ryan PD, et al. A two-gene expression ratio predicts clinical outcome in breast cancer patients treated with tamoxifen. Cancer Cell 2004; 5 : 607–16. 21. Paik S, Shak S, Tang G, et al. A multigene assay to predict recurrence of tamoxifen-treated, node-negative breast cancer. N Engl J Med 2004; 351 : 2817–26. 22. Mamounas EP, Fisher B. Preoperative (neoadjuvant) chemotherapy in patients with breast cancer. Semin Oncol 2001; 28 : 389–99. 23. Bertucci F, Finetti P, Rougemont J, et al. Gene expression profiling for molecular characterization of inflammatory breast cancer and prediction of response to chemotherapy. Cancer Res 2004; 64 : 8558–65. 24. Folgueira MA, Carraro DM, Brentani H, et al. Gene expression profile associated with response to doxorubicin-based therapy in breast cancer. Clin Cancer Res 2005; 11 : 7434–43. 25. Chang JC, Wooten EC, Tsimelzon A, et al. Gene expression profiling for the prediction of therapeutic response to docetaxel in patients with breast cancer. Lancet 2003; 362 : 362–9. 26. Chang JC, Wooten EC, Tsimelzon A, et al. Patterns of resistance and incomplete response to docetaxel by gene expression profiling in breast cancer patients. J Clin Oncol 2005; 23 : 1169–77. 27. Iwao-Koizumi K, Matoba R, Ueno N, et al. Prediction of docetaxel response in human breast cancer by gene expression profiling. J Clin Oncol 2005; 23 : 422–31. 28. Ayers M, Symmans WF, Stec J, et al. Gene expression profiles predict complete pathologic response to neoadjuvant paclitaxel and fluorouracil, doxorubicin, and cyclophosphamide chemotherapy in breast cancer. J Clin Oncol 2004; 22 : 2284–93. 29. Gianni L, Zambetti M, Clark K, et al. Gene expression profiles in paraffin-embedded core biopsy tissue predict response to chemotherapy in women with locally advanced breast cancer. J Clin Oncol 2005; 23 : 7265–77. 30. Hannemann J, Oosterkamp HM, Bosch CA, et al. Changes in gene expression associated with response to neoadjuvant chemotherapy in breast cancer. J Clin Oncol. 2005; 23 : 3331–42. 31. Hess KR, Anderson K, Symmans WF, et al. Pharmacogenomic predictor of sensitivity to preoperative chemotherapy with paclitaxel and fluorouracil, doxorubicin, and cyclophosphamide in breast cancer. J Clin Oncol 2006; 24 : 4236–44. 32. Ring A, Dowsett M. Mechanisms of tamoxifen resistance. Endocr Relat Cancer 2004; 11 : 643–58. 33. Jansen MP, Foekens JA, van Staveren IL, et al. Molecular classification of tamoxifen-resistant breast carcinomas by gene expression profiling. J Clin Oncol 2005; 23 : 732–40. 34. Chang HY, Sneddon JB, Alizadeh AA, et al. Gene expression signature of fibroblast serum response predicts human cancer progression : similarities between tumors and wounds. PLoS Biol 2004; 2 : E7. 35. Chang HY, Nuyten DS, Sneddon JB, et al. Robustness, scalability, and integration of a wound-response gene expression signature in predicting breast cancer survival. Proc Natl Acad Sci USA 2005; 102 : 3738–43. 36. Chi JT, Wang Z, Nuyten DS, et al. Gene expression programs in response to hypoxia : cell type specificity and prognostic significance in human cancers. PLoS Med 2006; 3 : e47. 37. Glinsky GV, Berezovska O, Glinskii AB. Microarray analysis identifies a death-from-cancer signature predicting therapy failure in patients with multiple types of cancer. J Clin Invest 2005; 115 : 1503–21. 38. Liu R, Wang X, Chen GY, et al. The prognostic role of a gene signature from tumorigenic breast-cancer cells. N Engl J Med 2007; 356 : 217–26. 39. Bild AH, Yao G, Chang JT, et al. Oncogenic pathway signatures in human cancers as a guide to targeted therapies. Nature 2006; 439 : 353–7. 40. Sotiriou C, Wirapati P, Loi S, et al. Gene expression profiling in breast cancer : understanding the molecular basis of histologic grade to improve prognosis. J Natl Cancer Inst 2006; 98 : 262–72. 41. Loi S, Haibe-Kains B, Desmedt C, et al. Definition of clinically distinct molecular subtypes in estrogen receptor-positive breast carcinomas through genomic grade. J Clin Oncol 2007; 25 : 1239–46. 42. Ivshina AV, George J, Senko O, et al. Genetic reclassification of histologic grade delineates new clinical subtypes of breast cancer. Cancer Res 2006; 66 : 10292–301. 43. Miller LD, Smeds J, George J, et al. An expression signature for p53 status in human breast cancer predicts mutation status, transcriptional effects, and patient survival. Proc Natl Acad Sci USA 2005; 102 : 13550–5. 44. Dupuy A, Simon RM. Critical review of published microarray studies for cancer outcome and guidelines on statistical analysis and reporting. J Natl Cancer Inst 2007; 99 : 147–57. 45. Shi L, Reid LH, Jones WD, et al. The microarray quality control (MAQC) project shows inter- and intraplatform reproducibility of gene expression measurements. Nat Biotechnol 2006; 24 : 1151–61. 46. Ein-Dor L, Kela I, Getz G, Givol D, Domany E. Outcome signature genes in breast cancer : is there a unique set ? Bioinformatics 2005; 21 : 171–8. 47. Fan C, Oh DS, Wessels L, et al. Concordance among gene-expression-based predictors for breast cancer. N Engl J Med 2006; 355 : 560–9. 48. Blay JY. Le futur des thérapeutiques ciblées en oncologie : trouver les cibles, traiter tôt et au long cours. Med Sci (Paris) 2007; 23 : 1073–4. 49. Charafe-Jauffret E, Chaffanet M, Bertucci F, et al. Les cancers du sein : vers un modèle cellulaire et moléculaire intégré. Med Sci (Paris) 2007; 23 : 626–32. 50. Jordan B. Chroniques génomiques : des puces ADN en clinique ? Med Sci (Paris) 2007; 23 : 210–4. 51. Michiels S, Hill C. Défis statistiques posés par les biopuces : autant d’espoir que de faux positifs ? Med Sci (Paris) 2008; 24 : 317–9. |