La lèpre, ou maladie de Hansen, est une maladie infectieuse chronique due à Mycobacterium leprae, une mycobactérie qui présente un tropisme particulier pour la peau et les nerfs humains [ 1], et dont la culture en milieu artificiel est aujourd’hui encore impossible. Endémique en Europe jusqu’au XVIIIè siècle, et même jusqu’au début du XXe siècle en Norvège, la lèpre y a été éradiquée avant l’avènement de son traitement par les antibiotiques, pour des raisons encore obscures (amélioration des conditions sanitaires ? acquisition d’une immunité croisée avec la tuberculose ?). En revanche, elle persiste encore dans de nombreux pays en voie de développement. En effet, malgré une campagne d’élimination menée depuis plus de 15 ans par l’OMS, qui a permis de réduire considérablement la prévalence de la maladie par la mise à disposition gratuite d’une antibiothérapie efficace, l’incidence de la lèpre a peu diminué et environ 300 000 cas sont encore diagnostiqués chaque année dans le monde [ 2]. Les manifestations cliniques de la lèpre se distribuent suivant un spectre clinique qui va d’un pôle tuberculoïde (formes localisées à la peau et aux nerfs dont les lésions contiennent peu ou pas de bacilles, reflétant une immunité à médiation cellulaire spécifique efficace) à un pôle lépromateux (formes disséminées dont les lésions sont très riches en bacilles en raison d’une réponse immunitaire cellulaire déficiente envers M. leprae) [ 3]. Parmi les maladies transmissibles, la lèpre est la principale cause de handicap physique, et les difformités terribles dues à la forme avancée de la maladie ont largement contribué aux préjugés profonds à l’origine du rejet des malades et de leurs proches par la société.
À la suite de la découverte de M. leprae par Armauer Hansen en 1873, la lèpre fut considérée par la communauté scientifique comme une maladie exclusivement infectieuse, après des siècles de croyances diverses au sujet de son origine, parmi lesquelles la transmission héréditaire de la maladie. Au milieu du siècle dernier, l’idée d’une prédisposition génétique au développement de la maladie ressurgit peu à peu, suggérée notamment par la grande diversité des réponses cliniques à l’exposition par M. leprae. Il est certes observé que le risque de transmission de la lèpre après contact avec un patient atteint est plus élevé lorsque le patient index est porteur de lésions riches en bacilles (formes lépromateuses), mais les cohortes d’études vaccinales [ 4– 6] montrent que, globalement, seuls 5 % des individus exposés à M. leprae développent une lèpre. De plus, chez les personnes développant la maladie, la présentation clinique est très hétérogène, certaines ne développant qu’une forme tuberculoïde et d’autres une forme disséminée. À ce jour, ni l’épidémiologie traditionnelle ni la microbiologie ne sont parvenues à expliquer cette hétérogénéité clinique. En particulier, aucune différence de virulence n’a jamais été mise en évidence entre les différentes souches de la mycobactérie, et les récentes avancées de la génétique moléculaire ont démontré que le génome de M. leprae était pratiquement invariant [ 7]. À l’inverse, il existe maintenant des éléments indiscutables (détaillés plus loin) en faveur d’une contribution génétique de l’hôte au contrôle de l’infection par M. leprae. Dans la mesure où il n’existe aucun modèle expérimental satisfaisant de l’infection par M. leprae, seuls les outils de la génétique épidémiologique ont permis ces avancées significatives dans la compréhension des mécanismes en jeu.
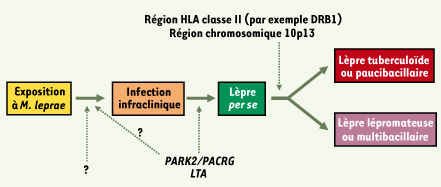
Les études d’épidémiologie génétique reposent sur l’utilisation conjointe d’informations de nature épidémiologique, telles que la mesure de facteurs de risque connus pour influencer l’expression de la maladie étudiée, et de nature génétique, comme les liens familiaux entre individus de l’échantillon ou encore le typage de marqueurs génétiques. On distingue schématiquement deux types d’approche selon qu’elles intègrent ou non l’étude de marqueurs génétiques (Tableau I) [ 8]. Dans le cas de la lèpre, il est probable que certains facteurs génétiques vont influencer le risque d’infection lors d’une exposition prolongée à M. leprae (hypothèse difficile à valider en l’absence de mesure suffisamment sensible de l’infection asymptomatique), d’autres la susceptibilité à la lèpre per se (c’est-à-dire la maladie indépendamment de sa forme clinique), et d’autres encore la polarisation vers une forme clinique particulière (Figure 1). Les études d’agrégation familiale ont observé une prévalence accrue de lèpre chez les individus apparentés à des patients lépreux [ 9] et des études de jumeaux ont retrouvé un taux de concordance pour la lèpre de 60 à 85 % chez les jumeaux monozygotes contre seulement 15-20 % chez les jumeaux dizygotes [ 10– 12]. Plusieurs analyses de ségrégation ont ensuite mis en évidence une forte composante familiale influençant la lèpre per se et ses sous-types [ 13, 14]. Depuis, la majorité des études ont cherché à localiser et à identifier les gènes et les variants de ces gènes qui influencent la susceptibilité à la maladie selon différentes stratégies exposées dans la Figure 2. Si l’étude de gènes candidats par hypothèse s’est avérée décevante, l’identification de gènes candidats par expérience (en particulier par clonage positionnel) a ouvert des voies totalement nouvelles dans la compréhension de l’histoire naturelle de la maladie.
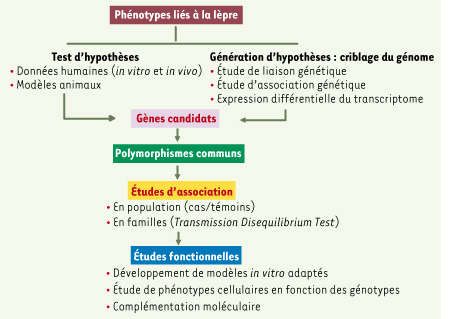
| Figure 2.
Stratégies d’identification de facteurs de susceptibilité génétique dans le cadre des maladies à hérédité complexe. L’approche « test d’hypothèse » consiste à sélectionner des gènes candidats a priori en se fondant sur des données issues d’études humaines in vitro ou in vivo ou de modèles animaux (peu contributifs dans le cas de la lèpre), de connaissances théoriques concernant la maladie ou des maladies proches (par exemple la tuberculose). L’approche « génération d’hypothèse » définit les gènes candidats sur la base des résultats d’une analyse de liaison génétique explorant le génome complet. Depuis peu, il est également possible de réaliser un criblage complet du génome par analyse d’association, ou d’utiliser l’analyse d’expression différentielle d’ARN messagers pour identifier des gènes d’intérêt. Le rôle des gènes candidats ainsi identifiés par l’une ou l’autre approche est ensuite étudié en testant l’association entre des polymorphismes communs de ces gènes et le phénotype d’intérêt, en utilisant des études d’association en population (cas-témoins) ou intrafamiliales (transmission disequilibrium test) [8]. Il est difficile de valider fonctionnellement les polymorphismes associés car l’effet attendu est subtil (comme dans toute autre maladie à déterminisme complexe) et le développement d’un modèle in vitro approprié difficile (M. leprae n’étant pas cultivable in vitro). |
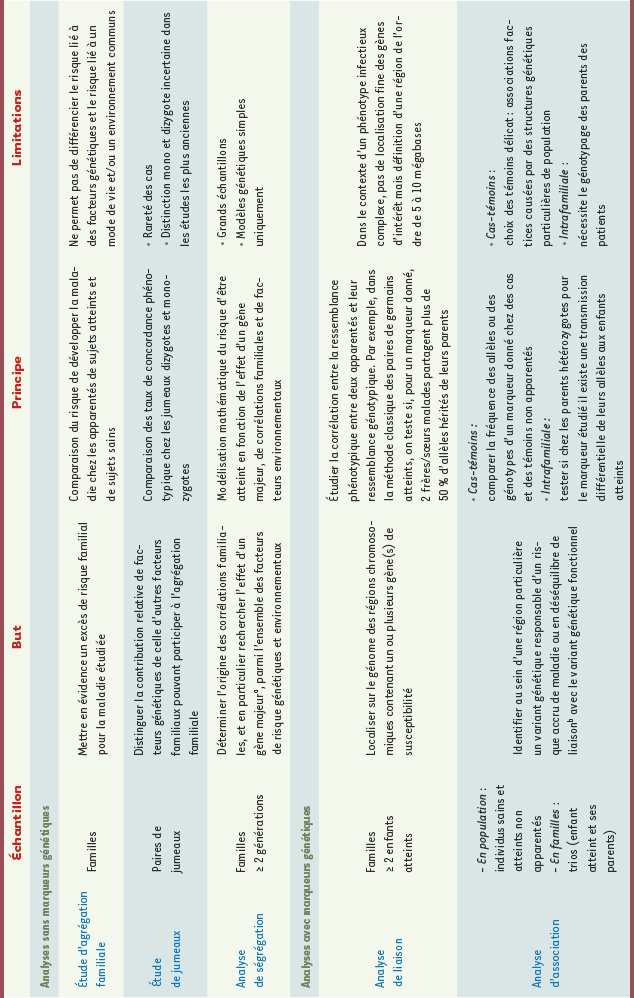
| Tableau I.
Principaux types d’analyses utilisés en épidémiologie génétique.
aLe terme « gène majeur » ne signifie pas qu’il s’agit du seul gène intervenant dans le phénotype étudié mais que, parmi l’ensemble des gènes impliqués, il en existe un dont l’effet est suffisamment important pour être distingué des autres. bIl existe un déséquilibre de liaison entre deux locus lorsqu’il existe à la fois une liaison génétique forte et une association allélique, c’est-à-dire que non seulement les locus coségrègent d’une génération à l’autre au sein de la famille, mais aussi leurs allèles s’associent préférentiellement au niveau de la population. |