À l’âge vénérable de près d’un siècle, la cartographie génétique reste une approche centrale en biologie, à laquelle on a recours dans des contextes variés, et avec des objectifs très différents : analyse de l’organisation des génomes (duplications, inversions, etc.), cartographie comparée (étude de la conservation de la synténie entre différentes espèces), cartographie de QTL (quantitative trait loci) et de eQTL (QTL d’expression), génétique d’association, isolement de gènes (positional cloning), SAM (sélection assistée par marqueurs), etc. Les populations génétiques utilisées, dont le choix répond à des objectifs spécifiques, se sont diversifiées, surtout depuis que l’on dispose de marqueurs moléculaires en nombre quasiment illimité. Il n’est pas toujours facile de s’y retrouver parce que les croisements mis en œuvre sont variés et que, ces populations ayant été développées indépendamment chez les végétaux et les rongeurs de laboratoires, le vocabulaire associé diffère souvent entre les deux domaines. Cet article fait le point sur la terminologie des populations expérimentales les plus courantes, leur mode d’obtention, leurs particularités et leurs intérêts.
Nous considérerons les descendances dont le génome est fixé à l’état homozygote, et qui peuvent donc être perpétuées à l’identique par croisements frères-sœurs ou autofécondation. Comme elles sont issues de croisements entre des populations homozygotes à tous leurs locus (lignées pures, ou fixées, ou consanguines), il n’y a que deux allèles par locus.
Les populations expérimentales peuvent être classées selon :
- la région génomique étudiée : certaines populations sont développées dans le but d’analyser la fonction d’un gène ou d’une région chromosomique, alors que d’autres visent à étudier l’ensemble du génome ;
- le type de croisement ayant permis de les obtenir. On distingue ici les populations expérimentales selon qu’elles sont issues d’une descendance F2 ou d’un rétro-croisement. C’est ce second critère que nous avons préféré, et qui est utilisé dans le Tableau I et les Figures 1 et 2.
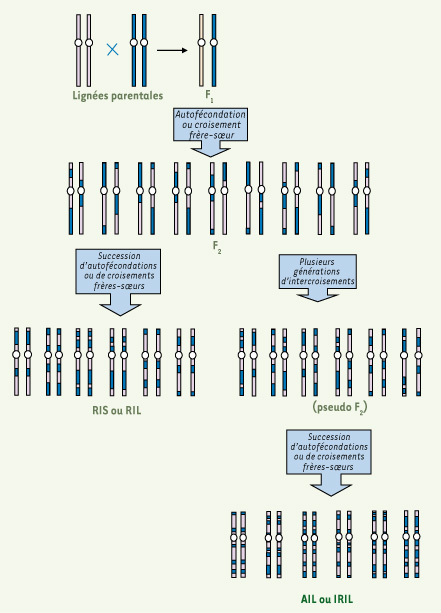
| Figure 1.
Populations de lignées recombinantes dérivables d’une descendance F2. Une seule paire de chromosomes est représentée sous la forme de « génotypes graphiques ». La taille moyenne des fragments dépend du nombre de méioses intervenues : elle est plus grande pour les AIL (ou IRIL) que pour les RIS (ou RIL), car les générations d’intercroisements ont permis davantage de recombinaisons. |
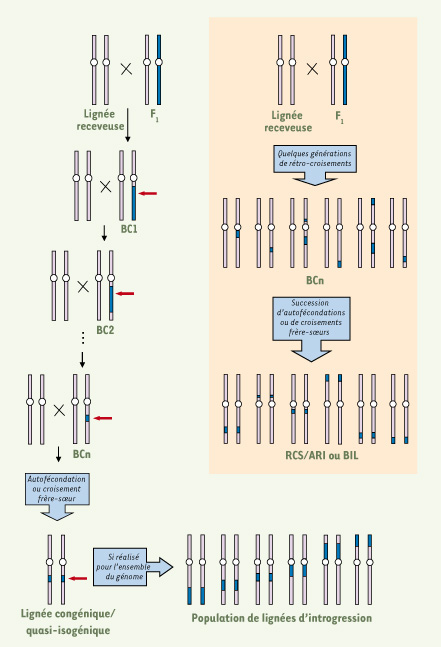
| Figure 2.
Matériels dérivés de rétro-croisements(BC : back-cross
). À gauche, développement de lignées congéniques (ou quasi-isogéniques). La flèche rouge indique le locus d’intérêt dont l’allèle doit être fixé dans le fonds génétique de la lignée receveuse. À droite, développement de lignées recombinantes « déséquilibrées ». Ce matériel diffère des lignées d’introgression (en bas à droite) par le fait que l’on n’a pas systématiquement cherché à obtenir une représentation exhaustive du génome donneur dans le génome receveur. |
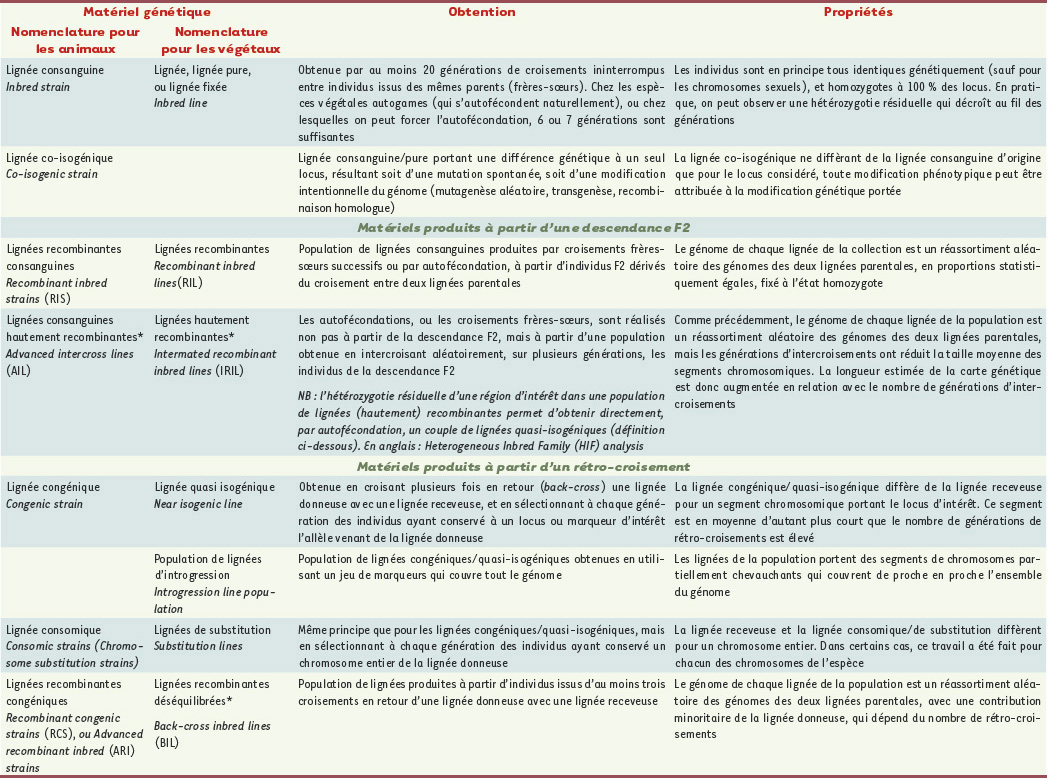
| Tableau I.
Nomenclature, mode d’obtention et propriétés des populations de cartographie génétique. *Proposition des auteurs, en l’absence de nomenclature établie. |
D’une façon générale, l’intérêt de travailler sur des lignées génétiquement homogènes et non sur des populations en ségrégation est de pouvoir effectuer des mesures phénotypiques non sur des individus isolés, mais sur un groupe d’individus isogéniques, afin d’accroître la précision de ces mesures, de pondérer l’influence des facteurs d’environnement, d’évaluer la variance intra-groupe et de comparer l’effet du sexe à génotype constant. Elles permettent également à différents laboratoires de travailler sur le même matériel génétique, pour comparer, corréler, voire compiler leurs résultats.
La plus petite variation génétique qu’il est possible d’étudier concerne un seul gène (ou toute séquence susceptible d’avoir un effet phénotypique). Elle peut avoir pour origine une mutation spontanée, une mutation produite par mutagenèse aléatoire (par un agent physique ou chimique) ou une modification induite par transgenèse ou par recombinaison homologue. Dans ce cas, la lignée obtenue ne diffère de la lignée consanguine de départ qu’au niveau de ce gène, et toute différence phénotypique observée peut donc être lui être attribuée. Ces lignées sont des modèles de choix pour démontrer le rôle d’un gène ou autre séquence dans un caractère, par exemple à l’issue d’une étape de cartographie génétique.
Des croisements frères-sœurs (ou des autofécondations) réalisés à partir d’une descendance F2 conduisent, à terme, à la production d’une série de lignées consanguines dites recombinantes (RIS ou RIL). Chacune porte, à l’état homozygote, une contribution égale des génomes des deux lignées parentales sous la forme de segments chromosomiques qui diffèrent d’une lignée à l’autre. Plus deux locus sont proches sur le même chromosome, plus ils tendent à être transmis dans un même segment. La capacité de séparer deux locus, qui est à la base de la cartographie génétique, croît avec le nombre de lignées recombinantes. Pour augmenter cette capacité résolutive, on peut réaliser plusieurs générations de croisements aléatoires à partir de la descendance F2, avant de commencer les croisements consanguins. On augmente d’autant le nombre de méioses informatives, donc la survenue d’événements de recombinaison qui réduisent la taille des segments. Cela constitue l’intérêt majeur des lignées hautement recombinantes (AIL ou IRIL). Ces différents types de populations permettent d’étudier l’ensemble du génome dans des populations possédant une contribution égale des deux lignées parentales.
Les croisements en retour (ou rétro-croisement, ou backcross) permettent de produire des lignées auxquelles les deux lignées parentales ont contribué de façon déséquilibrée. On distingue alors une lignée « receveuse », celle dont le génome se retrouve en proportion majoritaire et qui constitue le fonds génétique des lignées, et une lignée « donneuse ». Les lignées congéniques/quasi-isogéniques résultent de croisements en retour répétés avec la lignée receveuse, accompagnés, à chaque génération, de la sélection pour la génération suivante d’individus hétérozygotes à un locus (ou une région chromosomique) d’intérêt. À l’issue de ce processus, qui comprend généralement une dizaine de rétro-croisements, le génome de la lignée produite ne diffère de celui de la lignée receveuse que pour un segment chromosomique contenant le locus d’intérêt mais également quelques dizaines ou centaines de gènes qui lui sont génétiquement liés. L’utilisation de marqueurs génétiques permet de réduire le nombre de générations nécessaires (protocole dit speed congenic) ou de fixer les bornes du segment chromosomique transféré. Ce protocole peut être utilisé pour produire une série de lignées (dites d’introgression) portant des segments chromosomiques partiellement chevauchants qui, de proche en proche, couvrent l’ensemble du génome. Lorsque chaque segment correspond à un chromosome entier et lui seul, on parle de lignée consomique ou lignée de substitution.
Enfin, des croisements frères-sœurs (ou des autofécondations) réalisés à partir d’une descendance issue d’un ou de plusieurs rétro-croisements permettent de produire des lignées recombinantes congéniques/recombinantes déséquilibrées. La contribution de la lignée donneuse dépend du nombre de générations de rétro-croisements qui précèdent la mise à l’état consanguin. Par comparaison avec les lignées recombinantes (consanguines), ces lignées permettent une meilleure dissection génétique des caractères à déterminisme complexe et offrent une plus grande puissance statistique pour mettre en évidence des interactions épistatiques entre gènes du fait que les différences génétiques entre lignées concernent une plus petite fraction du génome.
L’ensemble de ces matériels génétiques représente une richesse inestimable pour la cartographie des gènes et l’étude des caractères complexes dans les espèces expérimentales. Nous espérons que cet article aidera généticiens « végétalistes » et « animalistes » à mieux se comprendre.