| |
| Med Sci (Paris). 2007 August; 23(8-9): 729–734. Published online 2007 August 15. doi: 10.1051/medsci/20072389729.Les microsatellites des génomes eucaryotes De leur cycle de vie et de leur neutralité Patricia Balaresque* Department of genetics, University of Leicester, Adrian Building, University Road, Leicester, LE17RH, Royaume-Uni |
Caractéristiques des séquences microsatellites Les microsatellites1 appelés également simple sequences ou short tandem repeats, sont des séquences simples et courtes, composées d’un motif k répété n fois [
1]. La longueur du motif k varie de 1 à 6 bases répétées en tandem2 et les différentes catégories sont désignées en fonction du nombre de bases contiguës constituant k. On parle de mono-nucléotide lorsque k = 1 [ex. (A)26] ou d’hexa-nucléotide lorsque k = 6 [ex. (CTGTCA)10]. Les répétitions qui constituent le motif peuvent être uniques (microsatellites parfaits) ou multiples (microsatellites imparfaits pouvant être interrompus ou composés) (Tableau I). Ce nombre de répétitions varie d’un individu à l’autre et au cours des générations, on parle de polymorphisme de taille [ex. (CA)15, (CA)11 ou (CA)19]. Les microsatellites montrent également un polymorphisme de structure, qui correspond à un changement d’une base en une autre à l’intérieur du motif [(CA)16, ici A → T (CA)13 CT (CA)2]. Si les changements de taille (~10−3 mutation/génération/locus) sont en moyenne 1 000 000 fois plus fréquents que les changements de structure (~ 10−9 mutation/génération/locus comme taux de substitution moyen), ils sont intimement liés les uns aux autres. Nombreux dans l’ensemble des génomes eucaryotes, ils représentent environ 1,6 % du génome humain [
3] et 3 % de celui de la souris [
4]. Les dinucléotides représentent 0,5 % de notre ADN dont 50 % sont (CA)n et 35 % (AT)n. Chez les mammifères et les arthropodes, les dinucléotides (CA)n sont les plus fréquents, mais ils sont fortement sous-représentés chez les oiseaux [
5], démontrant une réelle hétérogénéité de la dynamique de chaque motif entre espèces, mais également au sein du génome d’une même espèce [
2].  | Tableau I.
Les différents types de motifs microsatellites.
|
|
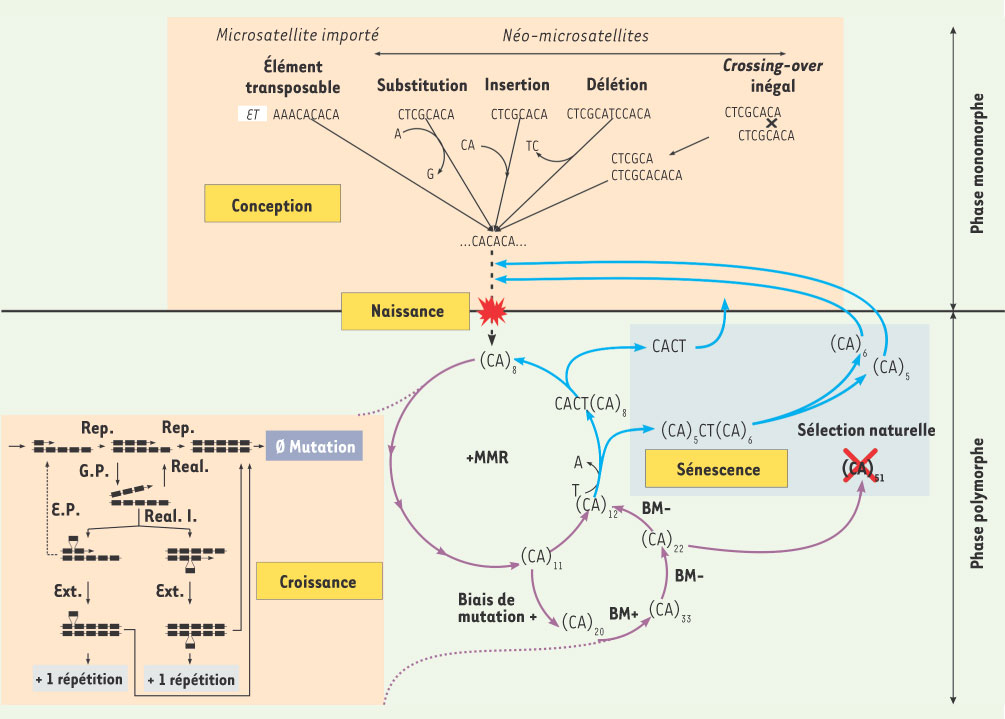
Le cycle de vie des microsatellites ou comment évolue le polymorphisme Les microsatellites sont des séquences possédant un réel potentiel évolutif voire adaptatif. Le fait que chaque locus ait la capacité de changer de taille au cours des générations, et se trouve aussi bien dans une région codante que non codante souligne leur caractère plastique. Mais cette plasticité repose essentiellement sur un facteur : leur taille. Faire référence à un microsatellite, c’est a priori désigner des unités répétées en tandem. On pourrait donc considérer que deux répétitions du même motif, par exemple (CA)2, constituent un microsatellite au même titre que (CA)8. Or il n’en est rien, puisque au terme microsatellite est également associée la notion de polymorphisme, cette dernière étant directement liée à une taille minimale en deçà de laquelle un microsatellite ne peut être considéré comme polymorphe. Comme nous allons le découvrir, le polymorphisme est transitoire, c’est un état temporaire caractéristique de la dynamique cyclique des microsatellites et de la variabilité du taux de mutation d’un locus donné au cours des générations. Les facteurs impliqués dans l’évolution des microsatellites peuvent être représentés (Figure 1) au sein d’un cycle vital à quatre phases, où le passage de l’une à l’autre est contrôlé par la taille : (1) la conception : mise en place de la matrice minimale (monomorphe par exemple (CA)3) ; (2) la naissance : le passage de la matrice initiale au microsatellite polymorphe ; (3) la croissance : phase de maturité caractérisée par le maintien d’un polymorphisme pendant plusieurs générations ; et (4) la sénescence ou renaissance : la disparition du locus ou le redémarrage d’un cycle.  | Figure 1.
Représentation sous forme de cycle de vie de la dynamique d’un locus microsatellite (par exemple CAn). Les quatre phases sont représentées avec conception, naissance, croissance et sénescence ou renaissance, et Rep : réplication, G.P. : glissement de la polymérase, Real :réalignement, Real I. : réalignement inégal, E.P. : exonucleolytic proofreading, Ext : Extension, et MMR : mismatch repair (système de réparation des mésappariements) pouvant intervenir à n’importe quel moment du cycle. |
La conception des microsatellites : la matrice minimale La genèse des microsatellites reste à l’heure actuelle une source de débats. C’est la notion de seuil de polymorphisme, en deçà duquel un microsatellite ne peut être polymorphe, qui suscite les plus grandes discussions. La majorité des auteurs s’accorde à dire que ce seuil est d’environ 8 répétitions (4 à 10) même si certains refusent l’idée même de son existence [
6,
7]. La question fondamentale est la suivante : comment expliquer la conception d’une matrice minimale (CA 2-3) qui serait susceptible d’évoluer en (CA) 8 ? Les études publiées jusqu’à ce jour n’ont pas clairement cherché à mettre en évidence les processus d’apparition des microsatellites, c’est pourquoi seules existent des preuves indirectes. Néanmoins, deux explications majeures se dégagent : soit la matrice minimale naît spontanément d’une séquence anonyme via différents mécanismes, on parlera alors de néo-proto-microsatellite, soit elle est copiée à partir d’une autre source via les éléments transposables ou les duplications, et l’on parlera de proto-microsatellite importé. Les néo-proto-microsatellites peuvent naître de différents événements : les substitutions ponctuelles, changement d’une base en une autre, peuvent, selon Messier et al. [
8] expliquer la création de quelques répétitions à partir d’une séquence anonyme. Néanmoins, elles ne peuvent expliquer la mise en place de longues matrices. Les insertions-délétions, en revanche, le pourraient très bien selon les travaux de Zhu et al. [
9], faisant de ces mécanismes des mécanismes-candidats sérieux pour la genèse des néo-proto-microsatellites. En fait, c’est probablement la combinaison de ces facteurs qui constitue le moyen le plus efficace d’obtenir une matrice minimale susceptible d’évoluer en microsatellite. La matrice minimale peut aussi provenir d’un néo-microsatellite importé via un élément transposable. Ces séquences abondantes structurent les génomes eucaryotes [
10], qu’il s’agisse des short et des long interspersed elements (SINE et LINE) ou des rétro-pseudogènes qui semblent en partie responsables de la dynamique spatiale des microsatellites [
11]. Ce sont les queues poly-A de ces éléments (SINE de type Alu notamment) qui expliqueraient la genèse de petits microsatellites riches en A comme A2-5NN [
12]. Mais alors les microsatellites riches en C, T ou G proviendraient-ils plutôt de néo-microsatellites que des microsatellites importés ? Cette question souligne la nécessité d’évaluer quelle proportion de microsatellites présents dans les génomes dériverait des néo-proto-microsatellites et des proto-microsatellites importés. La naissance Expliquer la mise en place de trois unités répétées successives est a priori simple comme le montre la Figure 1 (phase de conception). Mais la naissance d’un microsatellite reste la phase la plus obscure de ce cycle : il faut (1) montrer s’il existe ou non un nombre minimal d’unités contiguës nécessaire à l’apparition d’un polymorphisme (c’est-à-dire un nombre minimal d’unités répétées nécessaire à la mise en place des facteurs de croissance), (2) déterminer quels facteurs expliqueraient la transition séquence anonyme → proto-microsatellite → microsatellite polymorphe (soumis à de nouveaux mécanismes gouvernant les changements de taille). Le défi aujourd’hui est de vérifier si ce seuil existe (et si oui de quel ordre est-il), ou bien si ce seuil n’existe pas et que l’apparition du polymorphisme est directement liée au contexte génomique, et fonction du locus. Lors d’une étude menée sur les microsatellites liés aux duplications segmentaires, nous avons montré qu’un polymorphisme pouvait s’observer autour de 6 répétitions suggérant que les séquences répétées peuvent se révéler d’autant plus polymorphes qu’elles se trouvent dans un environnement répété [
13]. L’environnement génomique serait donc fondamental pour comprendre comment un microsatellite a des chances d’atteindre son état polymorphe, sans exclure néanmoins l’existence d’un seuil. La croissance : maturité et régime « yoyo » des microsatellites La phase de croissance des microsatellites constitue la période de leur vie pendant laquelle ils sont polymorphes, et, de ce fait informatifs et utiles dans de nombreuses disciplines. Les facteurs capables d’expliquer les variations de ce polymorphisme se répartissent en cinq groupes : les mécanismes de mutations impliqués, la nature même du microsatellite (par exemple, la longueur du motif), le contexte génomique (le contenu en GC), le contexte individuel (par exemple le sexe) et enfin les influences sélectives [
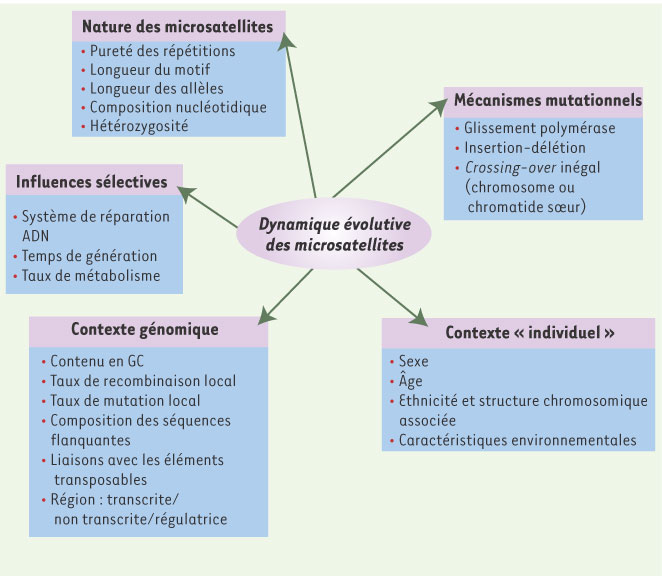
14]. Je m’attacherai à décrire ici les mécanismes mutationnels capables d’expliquer les changements de taille (et donc responsables du polymorphisme de ces séquences dans les populations humaines) et schématiserai les autres facteurs susceptibles d’expliquer les variations de ce polymorphisme entre individus (Figure 2). 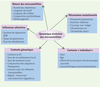 | Figure 2.
Polymorphisme des microsatellites. Les cinq catégories de facteurs capables d’expliquer les variations de polymorphisme d’un locus microsatellite à l’autre : les mécanismes mutationnels, la nature des microsatellites, le contexte génomique, le contexte individuel et les influences sélectives. |
Différents mécanismes mutationnels non exclusifs peuvent être à l’origine des changements de taille à partir d’un microsatellite minimum (CA)8. La recombinaison inégale entre chromosomes et/ou chromatides sœurs pourrait expliquer les variations de taille, l’échange inégal de segments d’ADN provoquant la perte d’unités sur un brin et le gain d’unités sur l’autre. Néanmoins, Klintschar et al. [
15] ont étudié 4 900 transmissions d’allèles microsatellites entre parents et enfants, mettant en évidence, par l’étude de marqueurs flanquant ces locus, l’absence d’événements de recombinaison inégale. Cette étude confirmerait donc la suprématie d’un seul mécanisme moléculaire capable d’expliquer les changements de taille des microsatellites et leur fort taux de mutations : le glissement de la polymérase ou slippage [
16]. Ce dernier serait en effet responsable des gains et pertes d’unités répétées. Le principe en est simple : lors de la polymérisation du néo-brin d’ADN, la polymérase effectue des pauses pendant lesquelles des mésappariements locaux peuvent survenir. Il se forme alors une/des boucle(s) en raison de la présence d’unités répétées (le microsatellite lui-même ou des répétitions environnantes). Il en résulte un ré-appariement imparfait, et donc une synthèse non fidèle de la matrice initiale. Selon que la boucle se forme sur le brin matrice ou le néo-brin, il y aura perte (contraction) ou gain (expansion) d’une unité répétée. Deux modèles d’évolution ont été proposés pour traduire ces changements de taille au cours des générations : le modèle SMM (single mutation model), impliquant le gain ou la perte d’une seule unité k et le TPM (two phase model) supposant majoritairement le gain ou la perte d’une unité k et plus rarement de plusieurs (pour revue, voir [
17]). Précisons que jusqu’à présent, ce phénomène a été observé dans de nombreuses ADN polymérases mais uniquement in vitro (pour revue, voir [
18]). L’ensemble des mécanismes mutationnels impliqués dans les changements de structure ou de longueur est assujetti aux mécanismes de réparation de l’ADN, corrigeant la plupart du temps les néo-mutations. De fait, l’apparition d’un nouvel allèle à un locus donné peut être considérée comme le résultat d’un équilibre subtil entre une mutation (substitution, insertions-délétions, crossing-over inégal ou glissement de la polymérase) et la non réparation de cette erreur [
19]. Je n’évoquerai que brièvement les deux voies principales de correction de ces erreurs : (1) exonucleolytic proofreading
3, qui répare essentiellement les mono/dinucléotides et prévient (partiellement) les incorporations de bases, influençant en priorité l’effet des insertions-délétions (voir phase de conception) ; (2) la réparation des mésappariements de bases (mismatch repair) qui corrige les mésappariements (voir phase de croissance). Ces deux mécanismes sont sensibles à de très nombreux facteurs comme la nature du microsatellite, sa composition nucléotidique, sa localisation sur l’ADN nucléaire ou celui des organites, le contenu en GC, l’état de méthylation, le niveau de transcription, l’espèce ainsi que les conditions environnementales associées à son milieu de vie [18]. Des altérations de ces systèmes de réparation entraînent l’accroissement du nombre de répétitions responsables de certains cancers (pour revue voir [
20]). En conséquence, évaluer précisément l’impact de ces systèmes de réparation sur les variations de taille au cours des générations permettrait de prédire l’évolution des structures microsatellites et plus particulièrement celles qui sont dans des régions dites chaudes (intra-génique ou dans des zones régulatrices). Vu la complexité même de la dynamique de chacun des facteurs influençant ces systèmes, l’exercice pourrait se révéler difficile. La sénescence ou renaissance Les microsatellites une fois dans leur phase de croissance semblent tout désignés pour atteindre de grandes tailles. Car même s’il semble s’établir une sorte d’équilibre entre expansion/contraction (glissement de la polymérase) et réparation des erreurs, il n’en reste pas moins que plus la longueur est importante plus elle rend les séquences sensibles aux mésappariements et formation de boucles. Ainsi, les répétitions semblent favoriser l’apparition de nouvelles répétitions. Les systèmes de réparations inaptes à réparer les larges insertions-délétions seraient donc moins efficaces dans des régions très répétées, favorisant l’accès à des tailles quasi-infinies. Pourtant les microsatellites dépassent rarement une taille supérieure à 100 bases [
21], les allèles longs étant qualifiés de rares [
22]. Kruglyak et al. ont rapporté un nombre de répétitions maximal pour les di-, tri- et tétranucléotides respectivement de 30, 15 et 15 répétitions chez l’homme, et de 25, 9 et 5 chez la drosophile [
23]. Trois explications sont possibles [
24] : (1) un biais de mutation expliquerait la contraction préférentiellement des allèles longs maintenant les microsatellites dans une gamme bien déterminée ; (2) la sélection naturelle préviendrait les expansions pouvant causer des problèmes structuraux lors de la synthèse de l’ADN ; (3) enfin, les substitutions et/ou les insertions-délétions seraient responsables de leur sénescence ou du démarrage d’un nouveau cycle. Les substitutions et les insertions peuvent interrompre un motif pur, entraîner la perte du potentiel polymorphique [
25] et la dégénérescence des microsatellites en séquences anonymes [
26] : c’est le processus de sénescence ; notons néanmoins que celui-ci surviendra d’autant plus probablement que l’interruption se produit au centre de la séquence. Si elle est dissymétrique, elle raccourcira le motif principal mais ce dernier pourra continuer son cycle : c’est la renaissance. |
Les microsatellites sont-ils des séquences neutres ? Les microsatellites sont qualifiés de séquences sélectivement neutres. Cette définition implique qu’ils sont a priori distribués de façon aléatoire dans les génomes, se transmettent selon un mode mendélien et sont capables d’enregistrer la trace des événements stochastiques passés [
27]. C’est pour cette raison qu’ils sont utilisés en médecine légale, combinés les uns aux autres afin de constituer des empreintes génétiques uniques. Également très employés en génétique des populations, ils servent à retracer l’histoire des populations contemporaines de n’importe quelle espèce. De telles utilisations impliquent que les mécanismes mutationnels régissant leur dynamique soient purement stochastiques. Pourtant, la littérature nous montre clairement qu’il n’en est rien. Par exemple, les distributions alléliques de certains microsatellites présents sur le chromosome X sont différentes entre hommes et femmes suggérant l’existence d’une sélection spécifique du sexe [
28] ; l’amplification d’un même locus chez des espèces ayant divergé de 3 à 470 mA laisse présumer d’une forte conservation de certaines séquences flanquantes [
29–
31] ; l’absence de tri-nucléotides dans des régions codantes, qui pourrait potentiellement décaler les cadres de lecture, souligne la contre-sélection de ces microsatellites dans des régions intra- mais également inter-géniques [
32] ; le polymorphisme de longueur d’un microsatellite (T)8 localisé en 3’ du gène CEACAM1 (carcinoembryonic antigen-related cell-adhesion molecule-1 biliary glycoprotein) affectant fortement l’expression de ce gène dans les cellules cancéreuses laisse présumer du rôle potentiel des microsatellites comme agents régulateurs [
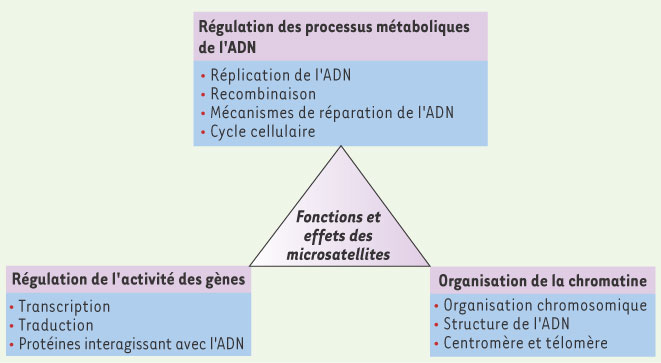
33] (voir Figure 3 [
34]). Enfin, substitutions et insertions-délétions seraient plus fréquentes au niveau des séquences flanquant les répétitions, laissant entrevoir un possible bais de mutation [
35].  | Figure 3
Fonctions et effets potentiels des microsatellites dans les génomes. Sous forme de trois champs d’action : régulation des processus métaboliques de l’ADN, régulation de l’activité des gènes et organisation de la chromatine. |
Ces observations isolées, parfois contradictoires, mais parfaitement informatives, soulignent : (1) la forte influence de l’environnement génomique et (2) la spécificité des processus de mutation agissant à proximité des microsatellites. Ce dernier point vient d’être analysé en détails [
36]. En analysant les séquences flanquant plusieurs milliers d’(AC)n dans les génomes humain et chimpanzé, ces auteurs ont mis en évidence un taux de mutation élevé au niveau des 20-50 bases flanquant les (AC)n, une forte asymétrie des processus de mutations 5’ versus 3’, et une évolution convergente des séquences flanquant des motifs de même taille (par exemple (AC)9). De plus, ils montrent que les biais de mutation (lorsque par exemple les transitions dominent les transversions) seraient observés dès le stade (CA)2 mais s’accentueraient avec la taille du microsatellite. Au final ces biais de mutation pourraient bien affecter plus de 30 % du génome humain, confirmant ainsi la difficulté qu’il y a à extrapoler les patterns de mutations. Néanmoins, cela ne résout pas le principal problème qui est de savoir si c’est l’environnement génomique qui influence les microsatellites ou bien le contraire. Dès lors, il apparaît très difficile de statuer sur le fait qu’ils soient neutres ou pas. Une certitude néanmoins : ils sont suffisamment neutres pour certaines disciplines. Ainsi, les variations de taux de mutations n’affectent en rien une recherche de paternité. En revanche, le caractère cyclique des processus de mutation pourrait bien rendre complexe la calibration de certains paramètres d’estimation en génétique des populations (coalescence) et invalider l’utilisation de certains systèmes type SNPSTR (single nucleotid polymorphisms short tandem repeats) (voir la discussion du concept dans [
37]). Continuer à clamer la parfaite neutralité de ces séquences, du moins sur l’ensemble de leur cycle vital, apparaît peu raisonnable. Néanmoins, les microsatellites ne sont pas tous soumis à des contraintes sélectives - à moins que les coefficients sélectifs ne soient très bas - car il existe un nombre très important d’études de génétique des populations ayant trouvé des réponses cohérentes à des questions biologiques spécifiques. La réponse pourrait être intermédiaire, entre neutralité et sélection, certaines phases de leur cycle de vie étant plus soumises au biais de mutation (conception, naissance, sénescence) que d’autres (croissance et second cycle). Bien que très populaires, les microsatellites restent donc au centre de nombreux débats. Ce sont des séquences complexes, dont la dynamique évolutive dépend de multiples facteurs aux dynamiques elles-mêmes relativement obscures (substitution, recombinaison, insertions-délétion, mécanisme de réparation de l’ADN, sélection, etc.). |
Je tiens à remercier le Dr Celia May pour nos échanges sur les crossing-over inégaux ainsi que le Dr Nicolas Poulet pour nos discussions sur l’évolution de ces séquences et sa précieuse assistance dans la réalisation des illustrations.
|
Footnotes |
1. Tautz D. Hypervariability of simple sequences as a general source for polymorphic DNA markers. Nucleic Acids Res 1989; 17 : 6463–71. 2. Lagercrantz U, Ellegren H, Andersson L. The abundance of various polymorphic microsatellite motifs differs between plants and vertebrates. Nucleic Acids Res 1993; 21 : 1111–5. 3. ISHGC, Lander ES, Linton LM, et al. Initial sequencing and analysis of the human genome. Nature 2001; 409 : 860–921. 4. Mouse Genome Sequencing Consortium. Initial sequencing and comparative analysis of the mouse genome. Nature 2002; 420 : 520–62. 5. Primmer CR, Raudsepp T, Chowdhary BP, et al. Low frequency of microsatellites in the avian genome. Genome Res 1997; 7 : 471–82. 6. Weber JL. Informativeness of human (Dc-Da)N.(Dg-Dt)N polymorphisms. Genomics 1990; 7 : 524–30. 7. Rose O, Falush D. A threshold size for microsatellite expansion. Mol Biol Evol 1998; 15 : 613–5. 8. Messier W, Li SH, Stewart CB. The birth of microsatellites. Nature 1996; 381 : 483-. 9. Zhu Y, Strassmann JE, Queller DC. Insertions, substitutions, and the origin of microsatellites. Genet Res 2000; 76 : 227–36. 10. Kazazian HH. Mobile elements : drivers of genome evolution. Science 2004; 303 : 1626–32. 11. Wilder J, Hollocher H. Mobile elements and the genesis of microsatellites in dipterans. Mol Biol Evol 2001; 18 : 384–92. 12. Nadir E, Margalit H, Gallily T, Ben-Sasson SA. Microsatellite spreading in the human genome : evolutionary mechanisms and structural implications. Proc Natl Acad Sci USA 1996; 93 : 6470–5. 13. Balaresque P, Toupance B, Heyer E, Crouau-Roy B. Evolutionary dynamics of duplicated microsatellites shared by sex chromosomes. J Mol Evol 2003; 57 : S128-S37. 14. Buschiazzo E, Gemmell NJ. The rise, fall and renaissance of microsatellites in eukaryotic genomes. Bioessays 2006; 28 : 1040–50. 15. Klintschar M, Dauber EM, Ricci U, et al. Haplotype studies support slippage as the mechanism of germline mutations in short tandem repeats. Electrophoresis 2004; 25 : 3344–8. 16. Levinson G, Gutman GA. Slipped-strand mispairing : a major mechanism for DNA sequence evolution. Mol Biol Evol 1987; 4 : 203–21. 17. Estoup A, Cornuet JM. Microsatellite evolution : inferences from population data. In : Goldstein DB, Schlotterer C, eds. Microsatellites : evolution and applications. Oxford : Oxford University Press, 1999 : 330. 18. Sia EA, Jinks-Robertson S, Petes TD. Genetic control of microsatellite stability. Mutat Res 1997; 383 : 61–70. 19. Tautz D, Schlotterer C. Simple sequences. Curr Opin Genet Dev 1994; 4 : 832–7. 20. Shibata D. Microsatellite analysis of human tumours. In : Goldstein DB, Schlotterer C, eds. Microsatellites : evolution and applications. Oxford : Oxford University Press, 1999: 330. 21. Schlotterer C. The evolution of molecular markers : just a matter of fashion ? Nat Rev Genet 2004; 5 : 63–9. 22. Goldstein DB, Pollock DD. Launching microsatellites : a review of mutation processes and methods of phylogenetic inference. J Heredity 1997; 88 : 335–42. 23. Kruglyak S, Durrett RT, Schug MD, Aquadro CF. Equilibrium distributions of microsatellite repeat length resulting from a balance between slippage events and point mutations. Proc Natl Acad Sci USA 1998; 95, 10774–8. 24. Amos W. A comparative approach to the study of microsatellite evolution. In : Goldstein DB, Schlotterer C, eds. Microsatellites : evolution and applications. Oxford : Oxford University Press, 1999 : 330. 25. Angers B, Bernatchez L. Complex evolution of a salmonid microsatellite locus and its consequences in inferring allelic divergence from size information. Mol Biol Evol 1997; 14 : 230–8. 26. Taylor JS, Durkin JM, Breden F. The death of a microsatellite : a phylogenetic perspective on microsatellite interruptions. Mol Biol Evol 1999; 16 : 567–72. 27. Schlotterer C. Evolutionary dynamics of microsatellite DNA. Chromosoma 2000; 109 : 365–71. 28. Balaresque P, Toupance B, Lluis QM, et al. Sex-specific selection on the human X chromosome ? Genet Res 2004; 83 : 169–76. 29. Schlotterer C, Amos B, Tautz D. Conservation of polymorphic simple sequence loci in cetacean species. Nature 1991; 354 : 63–5. 30. Rico C, Rico I, Hewitt G. 470 million years of conservation of microsatellite loci among fish species. Proc R Soc Lond B Biol Sci 1996; 263 : 549–57. 31. Blanquer-Maumont A, Crouau-Roy B. Polymorphism, monomorphism, and sequences in conserved microsatellites in primate species. J Mol Evol 1995; 41 : 492–7. 32. Metzgar D, Bytof J, Wills C. Selection against frameshift mutations limits microsatellite expansion in coding DNA. Genome Res 2000; 10 : 72–80. 33. Ruggiero T, Olivero M, Follenzi A, et al. Deletion in a (T)(8) microsatellite abrogates expression regulation by 3’-UTR. Nucleic Acids Res 2003; 31 : 6561–9. 34. Li YC, Korol AB, Fahima T, et al. Microsatellites : genomic distribution, putative functions and mutational mechanisms. Mol Ecol 2002; 11 : 2453–65. 35. Brohede J, Ellegren H. Microsatellite evolution : polarity of substitutions within repeats and neutrality of flanking sequences. Proc R Soc Lond B Biol Sci 1999; 266 : 825–33. 36. Vowles EJ, Amos W. Evidence for widespread convergent evolution around human microsatellites. Plos Biol 2004; 2 : 1157–67. 37. Mountain JL, Knight A, Jobin M, et al. SNPSTRs : empirically derived, rapidly typed, autosomal haplotypes for inference of population history and mutational processes. Genome Res 2002; 12 : 1766–72. |