| |
| Med Sci (Paris). 2007 August; 23(8-9): 705–706. Published online 2007 August 15. doi: 10.1051/medsci/20072389705.
Helicobacter pylori, notre fidèle compagnon gastrique François Balloux* Department of Genetics, University of Cambridge, Downing Street, Cambridge, CB2 3E Royaume-Uni MeSH keywords: Muqueuse gastrique, Variation génétique, Géographie, Infections à Helicobacter, Helicobacter pylori, Humains |
Ces dernières années ont vu l’émergence d’un quasi-consensus sur l’histoire de la colonisation du monde par l’homme anatomiquement moderne. Si de nombreux détails restent à clarifier, seule une minorité de chercheurs dans le domaine met en doute une origine africaine récente (~200 000 ans). Il y a 60 000 ans environ, une expansion démographique a entraîné une sortie hors de l’Afrique et la colonisation de tous les continents Ce scénario est fondé sur l’analyse de marqueurs génétiques haploïdes (ADN mitochondrial et chromosome Y) [
1,
2], de marqueurs autosomiques [
3–
5] ainsi que de données morphologiques [
6] et archéologiques [
7]. |
Dater l’association entre l’homme et ses pathogènes Ces mouvements migratoires depuis une population ancestrale africaine expliquent pourquoi la variabilité génétique a une distribution géographique caractéristique. Les populations génétiquement les plus diverses se trouvent sur le continent africain. Le polymorphisme décroît de façon linéaire au fur et à mesure que l’on s’éloigne de l’Afrique, si cette distance est estimée comme la distance la plus courte possible en restant sur la terre ferme autant que possible [3, 5]. Cela peut s’expliquer par la colonisation de proche en proche qui a entraîné, à chaque étape, une perte au hasard de certains des allèles présents dans la population parentale. Le phénomène peut être visualisé par analogie à la diffusion d’une goutte de colorant dans l’eau, qui, de façon transitoire, se traduira par une densité de pigments diminuant lorsque l’on s’éloigne du point d’impact de la goutte. Ces progrès dans la description de la distribution géographique de la diversité génétique humaine ouvrent de nouvelles possibilités d’inférences sur l’âge de l’association entre notre espèce et les maladies infectieuses qui nous affectent. Schématiquement, plus l’association entre un pathogène et l’homme est ancienne, plus grande sera la similitude des distributions géographiques de leurs diversités génétiques. À l’exception de certaines maladies émergentes (SRAS ou syndrome respiratoire aigu sévère, VIH), on ne sait que très peu de choses sur l’âge des associations entre les humains et leurs pathogènes. Une hypothèse souvent avancée est que l’origine de la plupart des maladies infectieuses humaines est postérieure à la découverte de l’agriculture, il y a environ 10 000 ans, dans la mesure où cette transition culturelle a entraîné des densités de population élevées. |
L’exemple d’ Helicobacter pylori
Un pathogène dont l’âge est particulièrement controversé est Helicobater pylori, une bactérie ne colonisant que l’estomac humain. Bien qu’infectant plus de la moitié de l’humanité, H. pylori n’a été décrite qu’en 1982 par deux chercheurs australiens, Robin Warren et Barry Marshall. D’emblée, Warren et Marshall étaient convaincus que H. pylori était impliquée dans la majorité des ulcères et des cancers de l’estomac. Ce point de vue n’étant pas partagé par la majorité de la communauté médicale, Marshall ingurgita une culture de ces bactéries, tomba fort malade puis se soigna avec des antibiotiques. Ce geste joua un rôle important dans la décision d’attribuer le prix Nobel de physiologie et de médecine 2005 aux deux chercheurs [
8]. Il a été suggéré que H. pylori n’infecte l’homme que depuis très récemment [
9]. Cette hypothèse a été remise en question lorsqu’il a été démontré que les populations sibériennes et amérindiennes étaient porteuses de souches apparentées, suggérant que les premiers hommes à s’être établi en Amérique après avoir traversé le Détroit de Béring il y a 10 000 à 20 000 ans étaient potentiellement déjà infectés [
10]. Afin de résoudre cette controverse nous avons récemment analysé 769 souches de H. pylori provenant de 51 populations humaines [
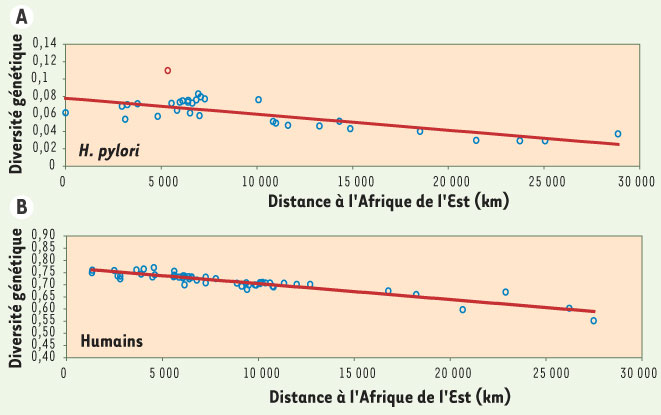
11]. Celles-ci ont été génotypées à plus de 1 500 sites variables distribués à travers le génome. La plus grande diversité génétique est observée en Afrique. En particulier, certains Xhosas d’Afrique du Sud sont porteurs de lignées très différenciées qui, à ce jour, n’ont été observées nulle part ailleurs. Comme c’est le cas pour les populations humaines, la diversité génétique de ces bactéries décroît de façon presque linéaire lorsque la distance géographique à l’Afrique s’accroît (Figure 1). Cette réduction de la diversité depuis l’Afrique devient plus difficile à mettre en évidence à une échelle géographique plus réduite. Au sein des continents, la distribution de la diversité est fortement influencée par des événements migratoires plus récents. L’Europe est la seule zone du globe pour laquelle nous disposions de suffisamment d’échantillons pour une analyse régionale. À cette échelle, nous avons à nouveau observé un parallèle frappant entre la distribution géographique des polymorphismes de H. pylori et des populations humaines. Finalement nous avons procédé a des simulations intensives sur ordinateur afin d’estimer l’origine géographique la plus probable de H. pylori ainsi que l’âge de l’expansion depuis cette source. Ces analyses suggèrent qu’une population ancestrale située en Afrique de l’Est est plus vraisemblable qu’une origine Sud Africaine ou asiatique. L’âge de l’expansion a été estimé à environ 58 000 ans, une date en concordance parfaite avec les datations les plus récentes pour l’expansion de l’homme [1, 2, 5].  | Figure 1.
Diversité génétique de populations de Helicobacter pylori (A) et de leur hôte, l’homme (B) représentée en fonction de la distance géographique à l’Afrique de l’Est en suivant les masses terrestres. La population de H. pylori mise en évidence en rouge (o en A) représente les bactéries isolées des Xhosas d’Afrique du Sud (version modifiée d’après [ 11]). |
Ces résultats démontrent que H. pylori infectait déjà nos ancêtres avant leur sortie d’Afrique, il y a 60 000 ans environ, voire depuis plus longtemps. Les différentes lignées bactériennes sont restées associées de façon étroite avec leurs populations hôtes lors de la colonisation du globe et depuis lors. L’intimité de cette association est étonnante dans la mesure où H. pylori se transmet aisément, comme l’indique le taux de réinfection élevé après traitement aux antibiotiques. Une hypothèse pouvant expliquer ce paradoxe, mais totalement spéculative à ce stade, serait que les lignées bactériennes sont adaptées localement à leurs hôtes et ne se maintiennent que dans les populations humaines avec lesquelles elles ont co-évolué. Dans ce contexte, il serait particulièrement intéressant d’étudier si les infections par des bactéries « allochtones » influencent le risque d’ulcères et de cancers de l’estomac. |
1. Underhill PA, Shen PD, Lin AA, et al. Y chromosome sequence variation and the history of human populations. Nat Genet 2000; 26 : 358–61. 2. Macaulay V, Hill C, Achilli A, et al. Single, rapid coastal settlement of Asia revealed by analysis of complete mitochondrial genomes. Science 2005; 308 : 1034–6. 3. Prugnolle F, Manica A, Balloux F. Geography predicts neutral genetic diversity of human populations. Curr Biol 2005; 15 : R159–60. 4. Prugnolle F, Manica A, Charpentier M, et al. Pathogen-driven selection and worldwide HLA class I diversity. Curr Biol 2005; 15 : 1022–7. 5. Liu H, Prugnolle F, Manica A, Balloux F. A geographically explicit genetic model of worldwide human settlement history. Am J Hum Genet 2006; 79 : 230–7. 6. Manica A, Amos W, Balloux F, Hanihara T. The effect of ancient bottlenecks on human phenotypic variation. Nature 2007; 448 : 346–8. 7. Mellars P. Going East: new genetic and archaeological perspectives on the modern human colonization of Eurasia. Science 2006; 313 : 796–800. 8. Mignon M. The Nobel Prize in Medicine, 2005. Barry J. Marshall and J. Robin Warren. Helicobacter pylori honored]. Med Sci (Paris) 2005; 21 :993–4. 9. Kersulyte D, Mukhopadhyay AK, Velapatino B, et al. Differences in genotypes of Helicobacter pylori from different human populations. J Bacteriol 2000; 182 : 3210–8. 10. Falush D, Wirth T, Linz B, et al. Traces of human migrations in Helicobacter pylori populations. Science 2003; 299 : 1582–5. 11. Linz B, Balloux F, Moodley Y, et al. An african origin for the intimate association between humans and Helicobacter pylori. Nature 2007; 445 : 915–8. |