| |
| Med Sci (Paris). 2007 June; 23(6-7): 633–639. Published online 2007 June 15. doi: 10.1051/medsci/20072367633.Lost in translation Le déblocage des ribosomes bactériens par le mécanisme de trans-traduction Reynald Gillet* and Brice Felden* Université de Rennes I, Inserm U835, UPRES JE 2311, Biochimie Pharmaceutique, 2, avenue du Professeur-Léon-Bernard, 35043 Rennes, France |
Le ribosome est la machinerie enzymatique permettant la synthèse protéique La biosynthèse des protéines est un mécanisme complexe, indispensable à la vie de toute cellule et conservé chez l’ensemble des organismes vivants. Le code génétique est construit à partir de quatre types de nucléotides différents regroupés en triplets (les codons) au sein des acides ribonucléiques messagers (ARNm). Grâce aux adaptateurs que sont les ARN de transfert (ARNt), l’information génétique portée par les ARNm est fidèlement traduite en une séquence comportant vingt acides aminés différents (la protéine), permettant ainsi de passer d’un alphabet de quatre à vingt lettres. Au cœur de ce système de traduction se trouvent les ribosomes, énormes particules constituées d’ARN et de protéines, assurant à la fois la fidélité du décodage de l’information génétique ainsi que la formation des liaisons entre acides aminés lors de la fabrication de la protéine naissante. La synthèse protéique est un processus complexe, en constante adaptation par rapport aux besoins cellulaires. À titre d’exemple, une bactérie en phase de croissance peut mobiliser l’essentiel de son énergie à la synthèse de nouvelles protéines. Le poids sec en ribosomes peut dès lors atteindre 45 % du poids de la cellule ! Sachant que chaque ribosome ajoute en moyenne vingt acides aminés à la seconde, ce sont des milliers de nouvelles protéines qui voient le jour en quelques instants. Ce n’est que depuis quelques années, grâce à l’acquisition de nombreuses données expérimentales biochimiques et génétiques ainsi qu’à l’extraordinaire avancée de la biologie structurale (notamment la cristallographie et la cryo-microscopie électronique), que le fonctionnement des ribosomes commence à être compris à l’échelle atomique [
1–
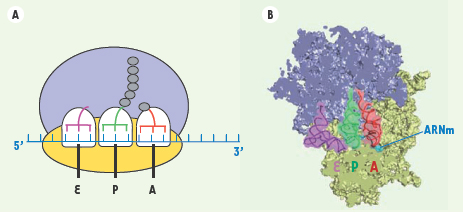
6]. Les ribosomes sont universellement constitués de deux sous-unités, respectivement dénommées 50S et 30S chez les bactéries en raison de leurs coefficients de sédimentation exprimés en unités Svedberg (S). La petite sous-unité (Figure 1AB, jaune) permet le décodage de l’information génétique portée par l’ARNm en assurant la reconnaissance de ses codons par les anticodons des ARNt complémentaires. La grande sous-unité (Figure 1AB, bleu) est responsable de la formation des liaisons peptidiques entre les acides aminés apportés par les ARNt aminoacylés. L’assemblage des deux sous-unités donne naissance à la particule complète 70S, dont les trois principaux sites actifs au sein desquels les ARNt vont évoluer sont le site A (ou site de liaison de l’aminoacyl-ARNt), le site P (ou site de liaison du peptidyl-ARNt), et le site E (ou site de sortie « exit » des ARNt) (Figure 1AB). Le processus de traduction est orchestré par de nombreux partenaires dont la majorité sont des facteurs protéiques dépendants d’une activation par la guanosine triphosphate ou GTP [
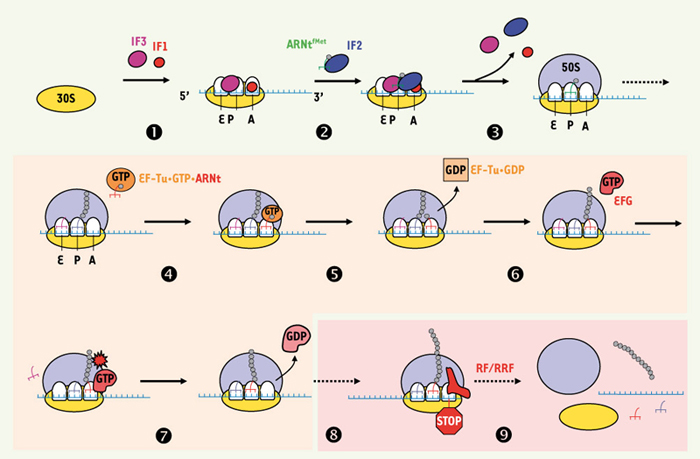
7]. Il peut se décomposer en trois étapes essentielles que sont le démarrage, l’allongement de la chaîne peptidique et la terminaison (Figure 2). Le démarrage de la traduction prend place sur la petite sous-unité du ribosome. Après liaison de l’ARN messager par un contact « ARNm-ARN ribosomique », le codon de démarrage AUG est placé dans le site P.  | Figure 1.
A. Vue schématique d’un ribosome en cours de traduction. La grande sous-unité (50S) est représentée en bleu, la petite sous-unité (30S) en jaune. De droite à gauche les trois alvéoles blanches représentent le site A (ou site de liaison de l’aminoacyl-ARNt), le site P (ou site de liaison du peptidyl-ARNt), et le site E (ou site de sortie « exit » des ARNt). Les ARN de transfert sont schématisés par des tridents de différentes couleurs et l’ARN messager est en bleu foncé. B. Structure tridimensionnelle d’un ribosome, obtenue par cristallographie aux rayons X [32]. Les couleurs attribuées à chaque composant sont identiques à celles de la Figure 1A. |
 | Figure 2.
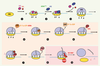
Représentation schématique de la synthèse protéique bactérienne (en vert pâle l’étape de démarrage (ou initiation) ; en orange pâle l’étape d’allongement de la chaîne peptidique ; en rose l’étape de terminaison). ❶ Après liaison de l’ARN messager, le codon de démarrage AUG est placé dans le site P de la petite sous-unité. IF1 (rouge) se lie au site A afin de favoriser la dissociation des deux sous-unités et bloquer temporairement l’accès aux ARNt. IF3 (violet) empêche l’association prématurée du 50S et favorise la sélection de l’ARNt initiateur (ARNt formylméthionine) au site P. ❷ IF2 (bleu) se lie à l’ARNt initiateur ❸ Les trois facteurs sont éjectés, le 50S rejoint le 30S pour former le complexe d’initiation 70S ❹ Les ARNt aminoacylés sont amenés au site A sous forme d’un complexe ternaire EF-Tu/GTP/ARNt (orange et rouge). ❺ En cas de reconnaissance « codon-anticodon », EF-Tu (orange) quitte le ribosome après hydrolyse du GTP. L’extrémité de l’ARNt porteuse de l’acide aminé bascule dans le site catalytique de la grande sous-unité du ribosome afin d’y subir le transfert peptidique. ❻ Le facteur d’élongation EF-G (rouge), dépendant du GTP pénètre dans le site A et entraîne le décalage de l’ARNt du site P vers le site E ainsi que de l’ARNt du site A au site P. ❼ Un nouveau codon se retrouve prêt à être reconnu par un ARNt dans le site A de la petite sous-unité. ❽ Le processus se poursuit de manière cyclique, jusqu’à ce qu’un codon de terminaison soit atteint. Les facteurs de terminaison RF1, RF2 et RF3 (rouge) entraînent l’hydrolyse puis le départ de la chaîne peptidique nouvellement synthétisée. ❾ Le facteur de recyclage du ribosome ou RRF (ribosome recycling factor) en association avec EFG permet de dissocier le ribosome afin de débuter un nouveau cycle de traduction (❶). |
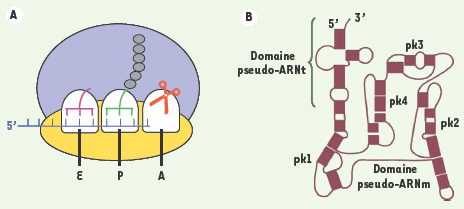
Trois facteurs protéiques d’initiation, IF1, IF2 et IF3 (initiation factors), interviennent. Une fois le système en place, ces trois facteurs se retirent, la grande sous-unité s’assemble à la petite. L’allongement proprement dit de la chaîne peptidique débute grâce aux facteurs d’élongation (elongation factors) EF-Tu et EF-G. Dans un processus itératif, les ARNt porteurs des acides aminés correspondant au codon de l’ARNm situé dans le site A du ribosome (selon les règles dictées par le code génétique) sont apportés par le facteur EF-Tu sous forme d’un complexe ternaire EF-Tu/GTP/ARNtaa. En cas de reconnaissance entre l’anticodon de l’ARNt aminoacylé et le codon de l’ARNm en place, un signal est transmis à la protéine EF-Tu qui quitte le ribosome après hydrolyse du GTP en GDP. L’extrémité de l’ARNt portant l’acide aminé bascule dans le site catalytique de la grande sous-unité du ribosome afin d’y subir le transfert peptidique. Cette réaction permet le transfert du peptide en cours de synthèse sur l’acide aminé de l’ARNt entrant. Elle est catalysée par l’ARN 23S, suggérant que le ribosome est un ARN catalytique (un ribozyme). Afin de préparer le ribosome pour l’étape suivante d’élongation de la chaîne polypeptidique, un second facteur d’élongation EF-G, capable lui aussi d’hydrolyser le GTP, intervient. En pénétrant dans le site A du ribosome, ce facteur entraîne le décalage de l’ARNt déacylé du site P vers le site E ainsi que de l’ARNt portant la chaîne peptidique du site A au site P. Un nouveau codon de l’ARNm se retrouve prêt à être reconnu par un ARNt dans le site A de la petite sous-unité. Ce processus se poursuit de manière itérative, jusqu’à ce que le codon situé dans le site A soit un codon de terminaison (UGA, UAG ou UAA). La reconnaissance de ce codon « stop » fait appel à trois facteurs de terminaison RF1, RF2 et RF3 (release factors). Leur liaison entraîne l’hydrolyse et le départ de la protéine nouvellement synthétisée. Enfin, le ribosome encore lié à l’ARNm et aux ARNt est dissocié grâce à l’intervention du facteur de recyclage du ribosome ou RRF (ribosome recycling factor), en association avec EF-G. Un cycle de traduction peut être à nouveau réalisé sur un nouvel ARNm. Cependant, il arrive que le processus de traduction ne parvienne pas à son terme, notamment en cas d’absence de codon « stop » sur l’ARNm (Figure 3A). Cette situation se présente lorsque l’ARNm est incomplet, endommagé par des ribonucléases par exemple. L’ARNm sera alors traduit jusqu’à son extrémité tronquée sans que les facteurs de terminaison puissent mettre un terme à la synthèse de la protéine. Cela a pour conséquence : (1) de bloquer le ribosome (ainsi que le chapelet de ribosomes en amont sur le message en cours de traduction), empêchant ainsi leur recyclage sur d’autres ARNm à traduire ; (2) de produire un polypeptide incomplet qui reste bloqué sur l’ARNt dans le site P du ribosome (Figure 3A). Afin de pallier ces défauts traductionnels, les bactéries ont développé un système de contrôle qualité de la synthèse protéique appelé trans-traduction, porté par un ARN particulier, l’ARN transfert-message ou ARNtm, hybride entre un ARN de transfert et un ARN messager (Figure 3B) [
8–
10]. 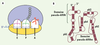 | Figure 3.
A. Ribosome bloqué en cours de traduction après coupure de l’ARN messager. Noter l’absence de codon dans le site A. B. Structure secondaire schématique de l’ARN transfert-message. Les domaines principaux de l’ARNtm sont le domaine pseudo-ARNt, le domaine pseudo-ARNm ainsi que les quatre pseudo-nœuds, annotés de PK1 à PK4. |
|
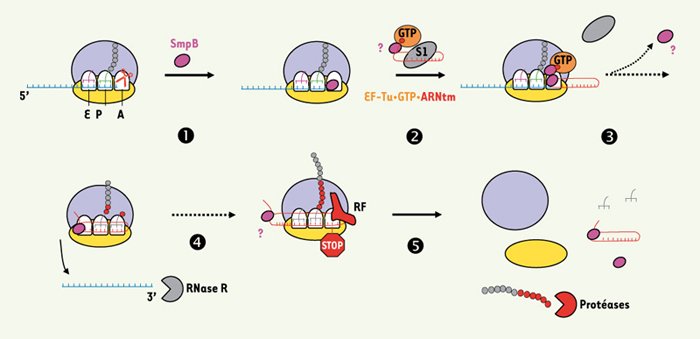
La trans-traduction est gouvernée par un ARN hybride et par des protéines associées La Figure 4 illustre le mécanisme de trans-traduction. Lorsque des ribosomes en cours de traduction parviennent à l’extrémité d’ARN messagers défectueux, une petite protéine basique appelée petite protéine B ou SmpB [
11], possédant certaines analogies avec des protéines du ribosome, s’associe avec les ribosomes bloqués [
12,
13]. Ce positionnement prépare l’arrivée de l’ARNtm. L’ARNtm aminoacylé avec une alanine à son extrémité 3’-terminale, en complexe avec le facteur d’élongation Tu, pénètre dans le site A du ribosome. Lors de cette étape initiale de reconnaissance, deux molécules de SmpB sont associées à chaque ribosome bloqué, l’une sur la grande sous unité au contact du centre GTPase nécessaire au départ d’EF-Tu, et l’autre sur la petite sous unité, dans le site de décodage [
14,
15]. Le mode d’arrivée de la seconde protéine SmpB (liée à la grande sous-unité) est encore débattu : soit directement sur le ribosome, soit en complexe avec l’ARNtm. Il s’ensuit l’étape d’accommodation, c’est-à-dire l’entrée de la portion pseudo-ARNt de l’ARNtmAla au sein du centre catalytique de la grande sous unité du ribosome. Ensuite s’effectue un transfert inhabituel, indépendant d’une reconnaissance codon-anticodon : le peptide porté par l’ARNt bloqué au site P du ribosome est transféré sur l’alanine de l’ARNtm. On suppose qu’à ce stade les deux molécules de SmpB entourant l’ARNtm ont chacune joué un rôle distinct au niveau de chaque unité ribosomique, en participant à la reconnaissance du ribosome bloqué au niveau de la petite sous-unité et à la transmission du signal de départ d’EF-Tu au niveau de la grande. La traduction reprend alors, non pas sur l’ARNm tronqué qui est rejeté du ribosome puis rapidement dégradé, mais sur la partie ARNm de l’ARNtm jusqu’au codon « stop » qu’elle contient. Trompé, le ribosome se dissocie et libère la protéine partielle à laquelle sont maintenant associés les acides aminés codés par l’ARNtm. Cette séquence est un étiquetage qui identifie la protéine ; ainsi marquée, elle est détruite par des enzymes spécifiques de la cellule. L’ARNm problématique est quant à lui dégradé par des exonucléases telles que la RNase R, évitant ainsi le démarrage d’une nouvelle traduction abortive [
16]. Il est à noter que si la trans-traduction est généralement activée sur des ARNm abîmés, il arrive également que, lorsque le ribosome est ralenti ou bloqué sur un ARNm intact (au niveau de codons rares, par exemple, impliquant un ralentissement temporaire de la traduction), ce dernier soit directement clivé dans le site de décodage par un facteur encore inconnu, induisant la trans-traduction. Ce mécanisme permet de réguler négativement l’expression d’ARNm spécifiques [
17]. 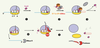 | Figure 4.
Représentation schématique de la trans-traduction. ❶ La protéine SmpB (violet) se lie au site A vide de la petite sous-unité d’un ribosome bloqué. ❷ Après activation par la protéine ribosomique S1 (gris) l’ARNtm rejoint le ribosome bloqué sous forme d’un complexe ternaire avec EF-Tu/GTP (orange). Deux protéines SmpB font partie du complexe initial. Le mode d’arrivée de la seconde protéine SmpB est encore débattu : soit directement au ribosome, soit en complexe avec l’ARNtm. ❸ Après transpeptidation, le ribosome bloqué bascule sur la portion messager de l’ARNtm. L’ARNm tronqué est éliminé et dégradé par les ribonucléases cellulaires (RNase R chez E. coli). ❹ La traduction se poursuit jusqu’à atteindre le codon de terminaison de l’ARNtm, reconnu par les facteurs de terminaison (RFs). ❺ Le peptide tronqué est libéré, marqué par un signal de dégradation par les protéases cellulaires (rouge). Le ribosome est ensuite recyclé. Les couleurs attribuées à chaque composant du ribosome bloqué sont identiques à celles de la Figure 1. |
|
La trans-traduction sous l’œil du microscope Récemment, des données structurales ont été collectées sur les acteurs de la trans-traduction à une échelle atomique, permettant de progresser considérablement dans la compréhension du mécanisme. Les données obtenues par cristallographie aux rayons X de la portion pseudo-ARNt de l’ARNtm en complexe avec l’une des deux molécules de SmpB [
18] ont permis de déterminer le mode de reconnaissance entre les deux partenaires. SmpB se lie à la région coudée flexible du domaine pseudo-ARNt (Figure 3B) de l’ARNtm, formé par la boucle D et la petite région variable (Figure 5A). L’ARN possède une conformation en L ouvert dans laquelle la boucle D, ne possédant pas de structure interne, est stabilisée par la protéine. Des contacts spécifiques entre l’ARN et la protéine sont situés à l’extrémité 3’ de la boucle D et dans la boucle joignant les tiges H2a et T. Un angle d’environ 120° se forme entre H2a et le bras accepteur. Par cryo-microscopie électronique, nous avons pu visualiser le mode de fixation de SmpB seul ainsi que celui du complexe ARNtm/SmpB/EF-Tu·GTP sur un ribosome bloqué [13, 15,
19]. Les données obtenues confirment tout d’abord la liaison de la protéine SmpB à un ribosome bloqué indépendamment de la présence de l’ARNtm. L’interaction a lieu dans le centre de décodage vide de la petite sous-unité (Figure 5B). Ces images confirment l’existence d’une étape précoce, indépendante de l’ARN, au cours de laquelle une première protéine SmpB agit en tant que sentinelle, en reconnaissant les ribosomes bloqués avant que l’ARNtm ne vienne exercer son action salvatrice, avec l’aide de facteurs protéiques associés comme la protéine ribosomique S1. L’étape suivante, appelée pré-accomodation, consiste en la présentation de l’ARNtm au ribosome par EF-Tu, juste avant le départ de ce dernier sous sa forme GDP (Figure 5C). L’analogie est forte entre l’interaction initiale de l’ARNtm et d’un ARNt avec le ribosome, malgré l’absence d’interaction de type codon-anticodon dans le cas de l’ARNtm, et malgré sa taille, qui est cinq fois supérieure à un ARNt. EF-Tu se positionne de la même manière dans les deux cas et permet à la portion pseudo-ARNt de l’ARNtm de pénétrer dans le ribosome à la manière d’un ARNt canonique. À ce niveau, la protéine SmpB, déjà en place sur la petite sous-unité, servirait d’intermédiaire entre la petite sous-unité du ribosome et l’ARNtm, en palliant l’absence de décodage. Ce positionnement permet en outre de préparer le redémarrage de la traduction en plaçant le début de la portion ARNm de l’ARNtm à proximité du site de décodage. Un autre avantage, compte tenu de la taille imposante de l’ARNtm, est de rediriger trois pseudo-nœuds (PK2-PK4) en dehors des sites actifs du ribosome afin de ne pas perturber le processus. Ceux-ci forment une grande boucle en lasso autour du bec de la petite sous-unité (Figure 5C). À ce stade, deux molécules de SmpB sont mises en évidence. Une molécule relie le domaine pseudo-ARNt à la grande sous-unité ribosomique (SmpB1, Figure 5CD) ; ce positionnement à proximité du centre GTPasique suggère l’implication de cette protéine dans le départ d’EF-Tu après que le ribosome bloqué ait accueilli l’ARNtm. La deuxième molécule est située dans le centre de décodage de la petite sous-unité comme le suggère l’étude cristallographique (Figure 5A) ; ce site correspond au positionnement de la protéine avant l’arrivée de l’ARNtm (SmpB2). Ainsi, deux protéines SmpB stabilisent la conformation de l’ARNtm interagissant avec le ribosome bloqué en se liant aux deux sous unités [15]. Si l'étape de démarrage est désormais bien caractérisée d'un point de vue structural, les étapes ultérieures (Figure 4, étapes 4 et 5) restent encore largement méconnues. 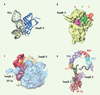 | Figure 5.
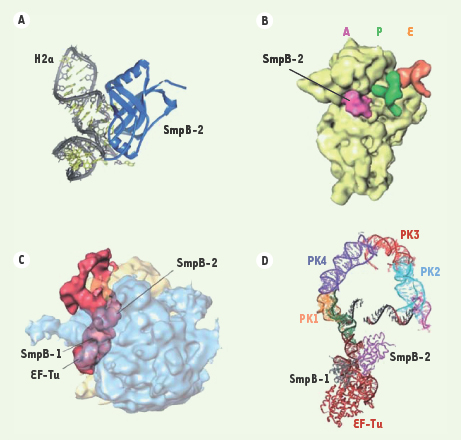
A. Complexe entre la protéine SmpB et le domaine pseudo-ARNt de l’ARNtm résolu par cristallographie aux rayons X. La protéine SmpB se lie à la partie coudée de la portion ARNt de l’ARNtm. L’une des deux faces basiques de la protéine (du côté de son extrémité amino-terminale) fait face à l’ARN tandis que l’autre (proche de l’extrémité carboxy-terminale) reste disponible pour interagir avec d’autres portions de l’ARNtm ou avec les ARN ribosomiques [18]. B. Localisation de l’une des deux protéines SmpB au sein de la petite sous-unité 30S d’un ribosome bloqué en cours de traduction par cryo-microscopie électronique. La protéine SmpB-2 est visualisée en rose dans le site de décodage. Les ARNt placés dans les sites P et E sont en vert et orange respectivement. La grande sous-unité a été volontairement omise par souci de clarté [13]. C. Vue tridimensionnelle d’un ribosome bloqué lié au complexe quaternaire ARNtm/2SmpB/EF-Tu•GTP obtenue par cryo-microscopie électronique. La densité du ribosome est représentée sous forme semi-transparente. Le complexe quaternaire est en rouge. SmpB1 interagit avec la grande sous-unité ribosomique ; SmpB-2 avec le centre de décodage de la petite sous-unité [15]. D. Modèle atomique du complexe représenté en C. Les protéines SmpB-1 et SmpB-2, respectivement colorées en gris et violet, sont placées de part et d’autre de la portion ARNt de l’ARNtm. La protéine EF-Tu (rouge) se situe dans la même position que lors de la traduction canonique tandis que les pseudo-nœuds forment un grand arc qui place le cadre ouvert de lecture de l’ARNtm à proximité du centre de décodage du ribosome [15] (Figures 5B et 5C et D reproduites avec la permission de The American society for biochemistry and molecular biology et The National Academy of Sciences, USA, respectivement). |
|
La trans-traduction est un mécanisme exclusivement procaryote À l’exception de quelques rares organismes primitifs (algues, diatomées), la trans-traduction est exclusivement retrouvée dans le monde bactérien. Bien que confrontées à la même nécessité de contrôle qualité de la synthèse protéique, les cellules eucaryotes sont dépourvues d’ARNtm et de SmpB. Les systèmes de surveillance mis à leur disposition ont évolué différemment, faisant appel à au moins trois mécanismes indépendants en fonction du problème rencontré :
-
Lorsque les ARNm portent des codons de terminaison prématurés, ils sont pris en charge, de manière coordonnée avec la machinerie d’épissage, par le système NMD (nonsense-mediated mRNA decay), qui permet à la cellule de rapidement les repérer puis de les dégrader, [
20].
-
Lorsque les ARNm sont dépourvus de codon stop, leur dégradation intervient via le système NSD (non-stop mRNA decay) [
21]. Ce système utilise l’exosome, pendant fonctionnel du « dégradosome » bactérien, doté de propriétés 3’ exonucléasiques. Le devenir des protéines tronquées libérées reste un mystère mais on peut supposer, par analogie avec le mécanisme à l’œuvre chez les procaryotes, qu’elles seraient également marquées pour être dégradées. L’un des systèmes susceptibles d’effectuer ce ciblage est l’ubiquitinylation qui dirige les protéines marquées vers le protéasome.
-
Enfin, tout comme chez les procaryotes, le blocage d’un ARN messager eucaryote dans le ribosome en cours de traduction entraîne sa coupure. Ce processus récemment découvert [
22] est dénommé no-go decay ou NGD. Il permettrait, grâce à l’action de plusieurs partenaires protéiques capables d’être recrutés par le ribosome, de couper spécifiquement l’ARNm dans le site de décodage afin d’entraîner par la suite sa dégradation par l’exosome.
|
Utilisation de la trans-traduction à des fins thérapeutiques et biotechnologiques Chez certaines bactéries impliquées dans des maladies humaines et/ou animales comme la salmonellose ou autres infections digestives (agents responsables : S. typhimurium et Y. pseudotuberculosis respectivement), un lien a été établi entre la trans-traduction et la virulence bactérienne. Parfois même, la bactérie est incapable de survivre lorsque la trans-traduction est affectée, lors d’infections comme la blennorragie par exemple (agent responsable : N. gonorrhae), tandis que d’autres espèces tolèrent l’absence de trans-traduction (E. coli ou B. subtilis par exemple) [
23–
27]. Bien que la trans-traduction ne soit pas systématiquement indispensable à la survie bactérienne, son absence, par inactivation génétique du couple ARNtm-SmpB, rend cependant les cellules plus sensibles à la présence de certains antibiotiques, même à des concentrations sub-létales [
28]. Cela n’est pas étonnant pour les antibiotiques inhibant la synthèse protéique comme les aminoglycosides, les macrolides, le chloramphénicol ou la spectinomycine. Ces molécules provoquent des erreurs de lecture ou des pauses prolongées en cours de traduction. On peut supposer que le couple ARNtm-SmpB permet à la bactérie de recycler les ribosomes bloqués par ces antibiotiques, entraînant la réutilisation de ces ribosomes sur d’autres messages et accroissant ainsi la survie bactérienne. Il est intéressant de noter que l’absence de trans-traduction a également un effet sur l’activité d’antibiotiques n’agissant pas sur la synthèse protéique, comme les inhibiteurs de la synthèse de la paroi bactérienne dont l’ampicilline et la phosphomycine sont deux représentants. Ce lien fonctionnel inattendu serait probablement consécutif à un stress général subi par la bactérie en présence de ces antibiotiques. Ce stress serait jugulé plus efficacement lorsque la trans-traduction est fonctionnelle [
29]. L’ensemble de ces données permet d’envisager les acteurs moléculaires essentiels de la trans-traduction comme des cibles thérapeutiques. En effet, des molécules capables de se lier spécifiquement à l’ARNtm, à SmpB ou au complexe ARNtm-SmpB pourraient être utilisées afin d’interférer avec le mécanisme d’entrée de l’ARNtm au ribosome bloqué et de potentialiser l’action d’antibiotiques existants. Cette association permettrait probablement de diminuer les concentrations d’antibiotiques utilisées afin d’en limiter les effets secondaires. Dans les quelques cas décrits où la trans-traduction est liée à la virulence et/ou à la survie bactérienne, des inhibiteurs de ce mécanisme pourraient s’avérer actifs en monothérapie. Aussi, le fait que les organismes eucaryotes soient dépourvus de ce mécanisme permet d’envisager la conception d’antibactériens interférant le moins possible avec les cellules et les métabolismes du malade. Une autre mise à profit du mécanisme de trans-traduction réside dans le fait que le complexe ARNtm-SmpB est présent et conservé chez l’ensemble des bactéries par rapport à la séquence des ARN ribosomiques. La séquence de l’ARNtm est nettement plus courte et présente l’avantage d’être fortement conservée aux extrémités et hautement variable ailleurs. Cela permet d’amplifier aisément sa séquence par réaction de polymérisation en chaîne (PCR) et de le séquencer ensuite afin de discriminer avec précision les espèces les unes des autres. Les études actuelles de génotypage bactérien font appel à des comparaisons d’ARN ribosomiques pourtant limitées par leurs taux élevés de conservation nucléotidique entre espèces ainsi que par leur grande taille (de 1 500 à 3 000 nucléotides). La forte divergence de la séquence primaire de la partie interne de l’ARNtm, associée à une forte conservation des deux extrémités 5’ et 3’, peut donc avantageusement être mise à profit pour ce type d’analyses. Des études ont permis de démontrer que l’utilisation d’amorces universelles complémentaires des deux extrémités de l’ARNtm permettait ainsi d’amplifier les séquences nucléotidiques de bactéries différentes puis, par comparaison des séquences internes, d’identifier les espèces bactériennes présentes [
30,
31]. |
Découvert en 1996, le processus de trans-traduction n’a eu de cesse de livrer les secrets d’un mécanisme d’action pour le moins atypique au sein des ribosomes nécessitant un sauvetage. Dix ans plus tard, le modèle proposé continue régulièrement de s’affiner, grâce à la collecte de données à la fois génétiques, biochimiques et structurales. Ces avancées laissent déjà entrevoir un certain nombre d’applications futures, tant d’un point de vue thérapeutique que biotechnologique. |
Les auteurs remercient les Drs Frank Murphy IV (Cambridge, UK), Nenad Ban (Zurich, Suisse) et Joachim Frank (Albany, NewYork, USA) pour les figures 1B ; 5B et 5A/C/D respectivement. Nous remercions également Rennes Métropole, la Région Bretagne (PRIR 691 et CRB 20041483), l’Action Concertée Initiative BCMS 136 et l’ANR programme MIME 2006 pour leur soutien financier.
|
1. Ramakrishnan V. Ribosome structure and the mechanism of translation. Cell 2002; 108 : 557–72. 2. Frank J. Toward an understanding of the structural basis of translation. Genome Biol 2003; 4 : 237. 3. Wimberly BT, Brodersen DE, Clemons WM Jr, et al. Structure of the 30S ribosomal subunit. Nature 2000; 407 : 327–39. 4. Ban N, Nissen P, Hansen J, Moore PB, Steitz TA. The complete atomic structure of the large ribosomal subunit at 2.4 Å resolution. Science 2000; 289 : 905–20. 5. Schluenzen F, Tocilj A, Zarivach R, et al. Structure of functionally activated small ribosomal subunit at 3.3 angstroms resolution. Cell 2000; 102 : 615–23. 6. Yusupov MM, Yusupova GZ, Baucom A, et al. Crystal structure of the ribosome at 5.5 A resolution. Science 2001; 292 : 883–96. 7. Nierhaus KH, Wilson DN ed. Protein synthesis and ribosome structure. Translating the genome. Weinheim : Wiley-VCH, 2004 : 580 p. 8. Keiler KC, Waller PR, Sauer RT. Role of a peptide tagging system in degradation of proteins synthesized from damaged messenger RNA. Science 1996; 271 : 990–3. 9. Karzai AW, Roche ED, Sauer RT. The SsrA-SmpB system for protein tagging, directed degradation and ribosome rescue. Nat Struct Biol 2000; 7 : 449–55. 10. Saguy M, Gillet R, Metzinger L, Felden B. tmRNA and associated ligands : a puzzling relationship. Biochimie 2005; 87 : 897–903. 11. Karzai AW, Susskind MM, Sauer RT. SmpB, a unique RNA-binding protein essential for the peptide-tagging activity of SsrA (tmRNA). EMBO J 1999; 18 : 3793–9. 12. Hallier M, Ivanova N, Rametti A, et al. Pre-binding of small protein B to a stalled ribosome triggers trans-translation. J Biol Chem 2004; 279 : 25978–85. 13. Gillet R, Kaur S, Li W, et al. Scaffolding as an organizing principle in trans-translation : the roles of proteins SmpB and S1. J Biol Chem 2007; 282 : 6356–63. 14. Hallier M, Desreac J, Felden B. Small protein B interacts with the large and the small subunits of a stalled ribosome during trans-translation. Nucleic Acids Res 2006; 34 : 1935–43. 15. Kaur S, Gillet R, Li W, Gursky R, Frank J. Cryo-EM visualization of transfer-messenger RNA with SmpB in a stalled ribosome as revealed at higher resolution. Proc Natl Acad Sci USA 2006; 103 : 16484–9. 16. Richards J, Mehta P, Karzai W. RNase R degrades non-stop mRNAs selectively in an SmpB-tmRNA-dependent manner. Mol Microbiol 2006; 62 : 1700–12. 17. Hayes CS, Sauer, RT. Cleavage of the A site mRNA codon during ribosome pausing provides a mechanism for translational quality control. Mol Cell 2003; 12 : 903–11. 18. Gutmann S, Haebel PW, Metzinger L, et al. Crystal structure of the transfer-RNA domain of transfer-messenger RNA in complex with SmpB. Nature 2003; 424 : 699–703. 19. Valle M, Gillet R, Kaur S, et al. Visualizing tmRNA entry into a stalled ribosome. Science 2003; 300 : 127–30. 20. Maquat LE. Nonsense-mediated mRNA decay : A comparative analysis different species. Curr Genomics 2004; 5 : 175–90. 21. Van Hoof A, Frischmeyer PA, Dietz HC, Parker R. Exosome mediated recognition and degradation of mRNAs lacking a termination codon. Science 2002; 295 : 2262–4. 22. Doma MK, Parker R. Endonucleolytic cleavage of eukaryotic mRNAs with stalls in translation elongation. Nature 2006; 440 : 561–4. 23. Komine Y, Kitabatake M, Yokogawa T, et al. 1994A tRNA-like structure is present in 10Sa RNA, a small stable RNA from Escherichia coli. Proc Natl Acad Sci USA 1994; 91 : 9223–7. 24. Muto A, Fujihara A, Ito KI, et al. Requirement of transfer-messenger RNA for the growth of Bacillus subtilis under stresses. Genes Cells 2000; 8 : 627–35. 25. Julio SM, Heithoff, DM, Mahan, MJ. ssrA (tmRNA) plays a role in Salmonella enterica Serovar Typhimurium pathogenesis. J Bacteriol 2000; 182 : 1558–63. 26. Huang C, Wolfgang MC, Withey J, et al. Charged tmRNA but not tmRNA-mediated proteolysis is essential for Neisseria gonorrhoeae viability. EMBO J 2000; 19 : 1098–107. 27. Okan NA, Bliska JB, Karzai AW. A role for the SmpB-SsrA system in Yersinia pseudotuberculosis Pathogenesis. PLoS Pathog 2006; 2 : 50–62. 28. Vioque A, de la Cruz J. Trans-translation and protein synthesis inhibitors. FEMS Microbiol Lett 2003; 218 : 9–14. 29. Luidalepp H, Hallier M, Felden B, Tenson T. tmRNA decreases the bactericidal activity of aminoglycosides and the susceptibility to inhibitors of cell wall synthesis. RNA Biol 2005; 2 : 70–4. 30. Felden B, Gesteland RF, Atkins JF. Eubacterial tmRNAs : everywhere except the alpha-proteobacteria ? Biochim Biophys Acta 1999; 1446 : 145–8. 31. Schonhuber W, Le Bourhis G, Tremblay J, Amann R, Kulakauskas S. Utilization of tmRNA sequences for bacterial identification. BMC Microbiol 2001; 1 : 20. 32. Selmer M, Dunham CM, Murphy FV 4th, et al. Structure of the 70S ribosome complexed with mRNA and tRNA. Science 2006; 313 : 1935–42. |