| |
| Med Sci (Paris). 2007 March; 23(3): 245–247. Published online 2007 March 15. doi: 10.1051/medsci/2007233245.Réplication, développement et pluripotence Jean-Marc Lemaitre,1,2 Damien Grégoire, and Marcel Méchali1* 1Institut de Génétique Humaine, CNRS, 141, rue de la Cardonille, 34980, Montpellier, France 2Responsable Équipe; Avenir Inserm, Plasticité du Génome et Vieillissement, Institut de Génomique Fonctionnelle, 141, rue de la Cardonille, 34089 Montpellier Cedex 05, France MeSH keywords: Animaux, Différenciation cellulaire, Noyau de la cellule, Chromosomes, Clonage d'organisme, Réplication de l'ADN, Épigenèse génétique, Régulation de l'expression des gènes au cours du développement, Microinjections, Mitose, Cellules souches multipotentes, Ovocytes, Cellules souches totipotentes, Xenopus laevis |
Le développement se caractérise par le passage d’un état cellulaire totipotent à un état différencié, qui s’accompagne d’une restriction dans les programmes de transcription et de réplication [
1–
5]. Ce processus, longtemps supposé irréversible, est à l’origine de l’identité cellulaire, et donc à la base de l’état multicellulaire. Ce n’est que dans la deuxième partie du XXe siècle que le dogme d’une différenciation irréversible est tombé avec les premières expériences de clonage animal. Le transfert du noyau d’une cellule issue d’un organisme différencié, dans un oeuf énucléé, conduit dans un certain nombre de cas à un individu viable. Ces expériences pionnières, réalisées dans les années 1950 aux États-Unis par R. Briggs et T.J. King chez la grenouille [
6] et poursuivies par la suite par J. Gurdon en Angleterre [
7], démontraient qu’un noyau de cellule en différenciation terminale était capable d’être déprogrammé par l’œuf, puis reprogrammé au cours d’un processus de développement. Leur taux de réussite était cependant faible, de l’ordre de 3 à 5 %, et si le résultat n’avait pas été aussi spectaculaire, il est probable que cette expérience scientifique particulièrement inefficace fut tombée aux oubliettes. Pourtant, elle mit en évidence l’extrême plasticité de l’expression du génome et ouvrit la voie de la reprogrammation cellulaire, qui est sans aucun doute à la base de la médecine régénératrice de demain. |
Totipotence et réplication Plusieurs espèces animales ont été maintenant clonées et un ensemble de manipulations combinant l’état de la cellule «donneuse »(type cellulaire, phase du cycle) ainsi que celui de la cellule réceptrice (ovocyte, œuf, cellule ES), visèrent à améliorer l’efficacité du clonage, sans pour autant lui donner le succès escompté. L’efficacité du clonage animal ne dépasse pas 10 % dans le meilleur des cas [
8]. Comment déprogrammer un noyau provenant d’un tissu différencié ? Comment rétablir un état de totipotence ? Comment une cellule totipotente s’engage t-elle dans la détermination d’une lignée spécifique ? La réponse à ces questions passe probablement par notre compréhension de la nature moléculaire des équilibres entre prolifération et différenciation au cours du développement. Une bactérie ou un organisme unicellulaire doit coordonner sa croissance à sa division, alors qu’un organisme multicellulaire doit également coordonner sa croissance et sa division à la différenciation. À chaque transmission du matériel génétique au cours des divisions, la réplication ne doit pas seulement reproduire le texte ADN mais également la manière dont ce texte est formaté pour la différenciation en cours. Ainsi, la distribution spatiale des origines de la réplication de l’ADN le long des chromosomes pourrait être impliquée dans l’organisation des chromosomes en domaines structurels et fonctionnels [
9,
10]. La notion de plasticité dans l’usage des origines de réplication a émergé avec des études réalisées sur des modèles d’embryogenèse chez le xénope et la drosophile. Chez le xénope, la fécondation déclenche un rythme de division accéléré, sans transcription et ce n’est qu’à partir du 13e cycle de division que la transcription et l’organogenèse de l’embryon démarrent. Un phénomène similaire a lieu chez la drosophile. Or, dans les deux cas, il a été démontré que les origines de réplication n’étaient pas fixées au cours du développement précoce, et ne se positionnent spécifiquement que lorsque les transcriptions démarrent dans l’embryon [
2,
11,
12]. Ainsi, de manière inattendue, le positionnement des origines de réplication apparaissait soumis à régulation au cours du développement. La micro-injection de plasmides recombinants dans un embryon précoce a ensuite permis de reproduire expérimentalement le positionnement d’une origine spécifique lors de la détermination d’un domaine génique par sa transcription [
13]. |
Cellule souche et réplication Une cellule souche, comme une cellule d’embryon précoce, garde sa pluripotence ; elle est capable de se différencier en plusieurs types cellulaires. Dès lors, on peut se demander si l’organisation de la réplication dans des cellules souches est une des caractéristiques conférant les propriétés particulières de ces cellules (stemness). Par ailleurs, comme chez l’embryon précoce du xénope ou de la drosophile, les cellules embryonnaires présentent un niveau élevé d’expression des membres du complexe pré-réplicatif, qui baisse de manière drastique lors de la différenciation [
14]. Une hypothèse séduisante est que l’état de totipotence des cellules souches puisse refléter un programme de réplication encore peu contraint. Les locus dont le profil de réplication a été analysé dans ces cellules, ceux de la β-globine et du X-inactivation center (Xic), sont répliqués à partir de nombreuses origines de réplication [1,
15]. De même, dans leur état indifférencié, les cellules pluripotentes P191 utilisent un grand nombre de sites pour initier la réplication du locus HoxB [
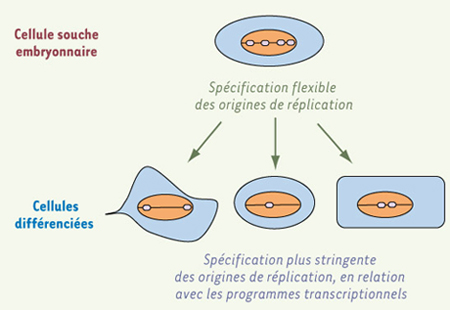
4]. En revanche, après induction de la différenciation cellulaire et de la transcription des gènes HoxB, le profil de réplication du locus HoxB est alors tout à fait différent, le domaine étant répliqué à partir d’une origine majeure localisée en aval du locus. Au niveau du génome entier, un plus grand nombre d’origines de réplication semble être activé à chaque phase S lorsque ces cellules sont dans un état indifférencié (D. Grégoire, M. Méchali, communication personnelle). Ainsi, il est possible que les origines de réplication soient positionnées de manière plus flexible dans les cellules souches totipotentes, un sous-ensemble de ces origines pouvant alors être activé dans les cellules différenciées en relation avec leurs programmes transcriptionnels spécifiques (Figure 1).
 | Figure 1.
Spécification des origines de réplication et différenciation.
|
Même si les conditions de cycle cellulaire sont bien différentes, on retrouve ainsi l’association entre pluripotence et flexibilité de position des origines de réplication lors du développement précoce et dans les divisions des cellules souches pluripotentes. |
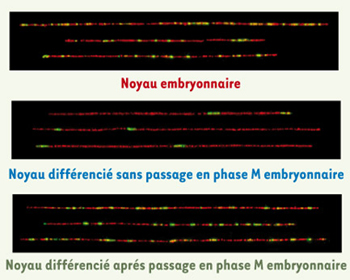
Reprogrammation du génome pour sa réplication Si l’organisation du génome pour sa réplication contribue à organiser l’architecture des chromosomes pour leur expression, alors une restructuration globale devrait être nécessaire pour que, lors du transfert d’un noyau somatique dans un œuf, les origines de réplication d’un noyau de cellule différenciée soient remises dans un état correspondant à un état de multi-ou totipotence. Nous avons démontré que la structure même du chromosome jouait un rôle déterminant dans cette réadaptation nucléaire et que la mitose était l’étape clé de cette restructuration. Le noyau d’une cellule somatique présente des marques « épigénétiques » qui permettent d’organiser le génome de sorte qu’il garde en mémoire les programmes de transcription et réplication caractéristiques de son identité cellulaire. Ce noyau mis dans le contexte d’un œuf, cellule bloquée en mitose, réacquiert la structure des chromosomes mitotiques de l’embryon, et peut alors au cours de la phase S suivante, répliquer son génome comme un noyau embryonnaire [
16]. En revanche, si ce noyau est incubé dans un contexte de phase S embryonnaire, il se contente de révéler les marques de structuration caractéristiques d’une cellule somatique différenciée (Figure 2). En effet, les origines de réplication d’une cellule somatique sont espacées en moyenne de 120 kb et sont organisées en domaines d’une taille similaire. Il semble indispensable pour permettre de positionner les origines de réplication tous les 20 kb de restructurer au préalable le génome en domaines de taille identique, et seule la mitose semble capable de reprogrammer le génome de la sorte, la phase S embryonnaire n’en étant pas capable. Cette reprogrammation des origines de réplication est étroitement liée à la topoisomérase II, impliquée par son activité enzymatique dans le métabolisme de la chromatine mais aussi dans la structuration du chromosome.  | Figure 2.
Reprogrammation des origines de réplication par la mitose. L’ADN des noyaux a été extrait et peigné sur lames silanisées. Les molécules individuelles d’ADN sont colorées en rouge tandis que les origines de réplication apparaissent en vert. M : mitose. |
Cette réorganisation mitotique des domaines de réplication suggère des améliorations possibles des méthodes de clonage animal. Elle démontre que la structuration du génome en mitose est une étape clé du couplage entre réplication et expression du génome, entre division et différenciation. |
Footnotes |
1. Aladjem MI, Rodewald LW, Lin CM, et al., Replication initiation patterns in the beta-globin loci of totipotent and differentiated murine cells: evidence for multiple initiation regions. Mol Cell Biol 2002; 22 : 442–52. 2. Hyrien O, Maric C, Mechali M. Transition in specification of embryonic metazoan DNA replication origins. Science 1995; 270 : 994–7. 3. Norio P, Kosiyatrakul S, Yang Q, et al. Progressive activation of DNA replication initiation in large domains of the immunoglobulin heavy chain locus during B cell development. Mol Cell 2005; 20 : 575–87. 4. Grégoire D, Brodolin K, Mechali M. HoxB domain induction silences DNA replication origins in the locus and specifies a single origin at its boundary. EMBO Rep 2006; 7 : 812–6. 5. Dazy S, Gandrillon O, Hyrien O, Prioleau MN. Broadening of DNA replication origin usage during metazoan cell differentiation. EMBO Rep 2006; 7 : 806–11. 6. Briggs R, King TJ. Transplantation of living nuclei from blastula cells into enucleated frogs’ eggs. Proc Natl Acad Sci USA 1952; 38 : 455–63. 7. Gurdon JB, Laskey RA, Reeves OR. The developmental capacity of nuclei transplanted from keratinized skin cells of adult frogs. J Embryol Exp Morphol 1975; 34 : 93–112. 8. Gurdon JB, Byrne JA. The first half-century of nuclear transplantation. Proc Natl Acad Sci USA 2003; 100 : 8048–52. 9. Mechali M. DNA replication origins: from sequence specificity to epigenetics. Nat Rev Genet 2001; 2 : 640–5. 10. Lemaitre JM, Méchali M. Organisation and dynamics of the cell nucleus for DNA replication. In : Hemmerich P, Diekmann S, eds. Vision of the nucleus. New York : American Scientific Publisher, 2005 : 37–51. 11. Sasaki T, Sawado T, Yamaguchi M, Shinomiya T. Specification of regions of DNA replication initiation during embryogenesis in the 65-kilobase DNApolalpha-dE2F locus of Drosophila melanogaster. Mol Cell Biol 1999; 19 : 547–55. 12. Lunyak VV, Ezrokhi M, Smith HS, Gerbi SA. Developmental changes in the Sciara II/9A initiation zone for DNA replication. Mol Cell Biol 2002; 22 : 8426–37. 13. Danis E, Brodolin K, Menut S, et al. Specification of a DNA replication origin by a transcription complex. Nat Cell Biol 2004; 6 : 721–30. 14. Fujii-Yamamoto H, Kim JM, Arai K, Masai H. Cell cycle and developmental regulations of replication factors in mouse embryonic stem cells. J Biol Chem 2005; 280 : 12976–87. 15. Rowntree RK, Lee JT. Mapping of DNA replication origins to noncoding genes of the X-inactivation center. Mol Cell Biol 2006; 26 : 3707–17. 16. Lemaitre JM, Danis E, Pasero P, et al. Mitotic remodeling of the replicon and chromosome structure. Cell 2005; 123 : 787–801. |