| |
| Med Sci (Paris). 2007 October; 23(10): 873–874. Published online 2007 October 15. doi: 10.1051/medsci/20072310873.Infection par le virus Chikungunya : les leçons de l’analyse génomique Paul-Henri Consigny* Consultation de Pathologie Infectieuse, Tropicale et de Médecine des Voyages, Centre Médical, Institut Pasteur, Paris Institut Pasteur, 28, rue du Docteur Roux, 75674 Paris Cedex 15, France MeSH keywords: Aedes, Afrique, Infections à alphavirus, Animaux, Asie de l'Ouest, Virus du chikungunya, Flambées de maladies, Génome viral, Glycoprotéines, Humains, Îles de l'Océan Indien, Vecteurs insectes, Italie, Voyage, Protéines de l'enveloppe virale, Virologie |
L’épidémie d’infections par le virus Chikungunya, classiquement responsable d’un tableau arthralgique fébrile, a sévi avec une ampleur particulièrement importante durant la période 2005-2006, atteignant de façon successive divers territoires géographiquement distincts, les îles de l’Océan Indien, puis le sous-continent indien. L’année 2007 marque cependant un retour vers une situation « inter-épidémique » dans l’Océan Indien, avec des cas sporadiques, indiquant malgré tout la présence persistante du virus. Les chiffres pour 2007 ne plaident en effet pas en faveur d’une reprise épidémique sur l’un au moins des territoires fortement touchés en 2006, à l’exception de l’Inde [
1]. |
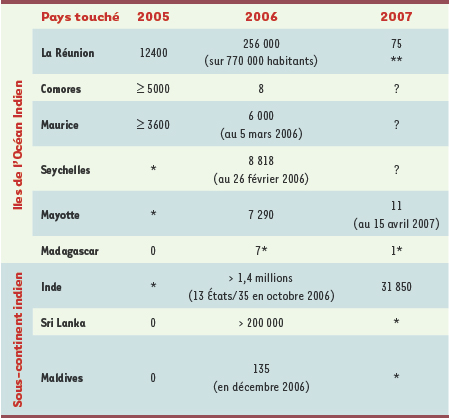
L’histoire de l’épidémie de 2005-2006 Dans l’Océan Indien, l’épidémie a démarré début 2005 dans les îles des Comores, puis a diffusé aux autres îles avoisinantes courant 2005, touchant l’île de la Réunion dès mars 2005. La diffusion de l’épidémie a pu y être suivie en temps réel par la suite, particulièrement début 2006, quand l’épidémie a connu ses pics d’incidence les plus importants (Tableau I). Si les chiffres paraissent moindres pour les autres îles de l’Océan Indien, il est probable que les taux d’incidence y ont été équivalents. L’exemple-type en est l’île de Mayotte, avec un nombre de cas déclarés relativement limité, contrastant avec une incidence importante estimée par des enquêtes de séroprévalence effectuées après l’épidémie [1]. Le différentiel observé est lié aux modalités d’accès aux soins sur cette île, où le recours à la médecine traditionnelle a été important, même en première intention.  | Tableau I.
É
pidémie de Chikungunya dans l’Océan Indien en 2005-2006 au 25 mai 2007. Les chiffres indiqués correspondent au nombre de cas suspects ou estimés (pour la Réunion). Ils sont tirés des sites Internet de l’Institut de Veille Sanitaire (InVS) et de Promed. *Données non disponibles (cas décrits, non quantifiés précisément). **Aucun cas autochtone à la Réunion depuis le 19 avril 2007. Un cas importé de Madagascar sur le dernier relevé de l’InVS du 30 juin 2007. |
L’analyse moléculaire en temps réel des souches isolées dans les territoires de l’Océan Indien a permis de mettre en évidence la diffusion d’une souche virale originaire, sur le plan phylogénétique, d’Afrique Centrale et d’Afrique de l’Est [
2–
4], par opposition aux souches ouest-africaine et asiatique. Cette souche a cependant subi, au cours de son expansion dans l’Océan Indien, des modifications génomiques ayant possiblement influé sur sa capacité de diffusion [2]. Ces résultats phylogénétiques sont corroborés par la précession de l’épidémie survenue dans l’Océan Indien par une flambée épidémique au Kénya sur la bande côtière (Mombasa, Lamu) au cours de l’année 2004 [
5]. L’extension ultérieure de l’épidémie au sous-continent indien, touchant une grande moitié inférieure du territoire indien (principalement les États du Karnataka, Maharashtra et Andhra Pradesh) ainsi que le Sri Lanka, et responsable de plus d’un million de cas, est, là encore, liée à une souche phylogénétiquement proche du sous-type viral est-africain, après séquençage des gènes NS4 et E1, contrairement aux épidémies antérieures impliquant le sous-type asiatique [
6]. Il est tout à fait plausible que cette souche ait trouvé son origine dans les îles de l’Océan Indien. |
Les raisons de l’ampleur de cette épidémie Au cours de l’épidémie de 2005-2006, un certain nombre d’hypothèses avaient été proposées pour expliquer son ampleur importante : des modifications du génome viral avaient-elles entraîné : (1) une meilleure adaptation à un hôte « théoriquement inhabituel », l’Aedes albopictus, et donc une meilleure capacité vectorielle ? (2) une plus grande pathogénicité de la souche virale, responsable d’un taux d’attaque particulièrement élevé et de formes cliniques sévères ? La première étude génomique disponible, émanant du Centre National de Référence des Arbovirus [2], concernait le séquençage complet de 6 souches et celui, partiel (séquences de la glycoprotéine de surface E1), de 121 souches de virus Chikungunya isolées dans les îles de l’Océan Indien (La Réunion, Seychelles, Maurice, Madagascar, Mayotte). Une mutation A226V, avec substitution d’une alanine par une valine, a été mise en évidence sur la glycoprotéine d’enveloppe E1 des souches virales « tardives », alors qu’elle était absente sur les souches virales « précoces ». La datation précise de son apparition la fait remonter à la période de l’hiver austral 2005, puisque si aucune souche séquencée avant juin 2005 ne présentait cette mutation, 90 % des souches isolées à partir de septembre 2005 la contenaient. L’accélération de l’épidémie qui a suivi indique que l’acquisition de cette mutation a correspondu à un « succès évolutif » pour la souche virale, possiblement lié à une meilleure adaptation du virus à son moustique vecteur, ce d’autant que ce vecteur, Aedes albopictus, était largement présent. Une hypothèse explicative, déjà évoquée pour d’autres alphavirus, suggère que cette mutation pourrait faire perdre au virus sa dépendance vis-à-vis du cholesterol, élément souvent déficitaire chez les moustiques, avec pour conséquence une meilleure adaptation à un environnement peu favorable chez le moustique [2]. Dans le même temps, le nombre de souches virales disponibles et responsables de formes sévères est resté limité, ne permettant pas pour le moment de répondre à la question de la plus grande virulence. Il a cependant été constaté que les virémies observées au cours des infections aiguës étaient élevées (jusqu’à 109 copies/ml), au regard de celles observées dans d’autres arboviroses [
3]. La capacité pour la souche épidémique de virus Chikungunya d’infecter les moustiques Aedes aegypti et Aedes albopictus s’est avérée équivalente en laboratoire [
7], ce qui conforte l’hypothèse du rôle majeur de ce dernier vecteur dans cette épidémie récente. Il est donc à craindre, avec l’extension de l’aire de répartition d’Aedes albopictus, en particulier dans le Sud de l’Europe, que des épidémies d’infections au virus Chikungunya n’émergent dans ces nouvelles régions, particulièrement dans le contexte actuel de réchauffement climatique [
8]. La poursuite d’une surveillance entomologique est donc capitale, de même que le dépistage précoce de toute infection importée (désormais à déclaration obligatoire), susceptible de diffuser à la population de moustiques, particulièrement en cas de virémie élevée chez un patient [4].

|
2. Schuffenecker I, Iteman I, Michault A, et al. Genome microevolution of Chikungunya viruses causing the Indian Ocean outbreak. PLoS Med 2006; 3 : 1058–70. 3. Parola P, de Lamballerie X, Jourdan J, et al. Novel Chikungunya virus variant in travelers returning from Indian Ocean islands. Emerg Infect Dis 2006; 12 : 1493–9 4. Bessaud M, Peyrefitte CN, Pastorino BAM, et al. Chikungunya virus strains, Reunion Island outbreak. Emerg Infect Dis 2006; 12 : 1604–6 5. Chrétien JP, Anyamba A, Bedno SA, et al. Drought-associated Chikungunya emergence along coastal East Africa. Am J Trop Med Hyg 2007; 76 : 405–7 6. Yergolkar PN, Tandale BV, Arankalle VA, et al. Chikungunya outbreaks caused by african genotype, India. Emerg Infect Dis 2006; 12 : 1580–3 7. Tsetsarkin K, Higgs S, McGee CE, et al. Infectious clones of Chikungunya virus (La Réunion isolate) for vector competence studies. Vector Borne Zoonotic Dis 2006; 6 : 325–37 8. Epstein PR. Chikungunya fever resurgence and global warming. Am J Trop Med Hyg 2007; 76 : 403–4. |