| |
| Med Sci (Paris). 2006 December; 22(12): 1087–1094. Published online 2006 December 15. doi: 10.1051/medsci/200622121087.ADN hélicases et maladies associées Muriel Uhring and Arnaud Poterszman* Institut de génétique et de biologie moléculaire et cellulaire, UMR 7104 CNRS/ Inserm/ULP, 1, rue Laurent-Fries, BP 10142, 67404 Illkirch Cedex, France |
La double hélice d’ADN constitue une structure stable bien adaptée à la transmission et à la sauvegarde de l’information génétique. Cependant, de nombreux aspects du métabolisme de l’ADN demandent l’accès à une forme simple brin dans laquelle les bases nucléotidiques sont accessibles, permettant l’intervention de protéines de remodelage, parmi lesquelles figurent hélicases et topo-isomérases. Les hélicases constituent une grande famille d’enzymes qui permettent le déroulement de doubles brins d’ADN ou d’ARN et utilisent l’énergie libérée par l’hydrolyse de l’ATP pour catalyser la rupture des liaisons hydrogènes entre paires de bases nucléotiques (pour revue, voir [
1]). Cette famille de protéines est caractérisée par un ensemble de séquences signatures, connues sous le nom de motifs hélicases [
2]. La fonction hélicase est assurée par un module catalytique composé de deux domaines α/β parallèles dont la topologie est identique à celle de RecA, une protéine bactérienne impliquée dans les processus de recombinaison [
3]. Si l’on inclut le motif Q récemment mis en évidence [
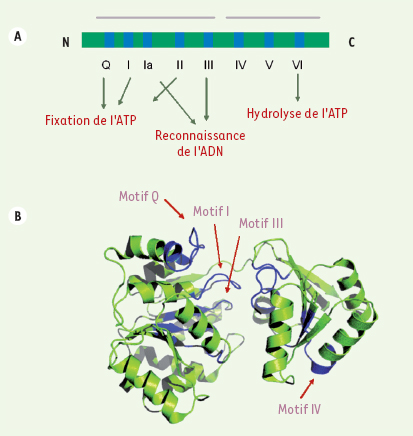
4], les motifs conservés, au nombre de 8 pour les hélicases des superfamilles SF1 et SF2, sont localisés à l’interface des deux domaines (Figure 1). On peut schématiquement distinguer les motifs intervenant dans la fixation de l’ATP/ADP-Mg, ceux dont la fonction est principalement de reconnaître l’oligonucléotide substrat et, enfin, ceux qui participent au mécanisme de couplage entre hydrolyse de l’ATP et changement conformationel, couplage conduisant au déroulement de la double hélice et au déplacement de brins (pour revue, voir [
5]). Les hélicases n’opèrent pas de manière isolée dans la cellule, mais leur action est couplée à celle d’autres enzymes du métabolisme des acides nucléiques (polymérase, ligase, endonucléase…), et bon nombre d’entre elles sont intégrées au sein de complexes multiprotéiques [
6].  | Figure 1.
Organisation et structure des hélicases. A. Organisation du module catalytique d’une hélicase : les domaines structuraux (traits gris), ainsi que les motifs hélicase (boîtes bleues), sont indiqués. B. Structure du domaine hélicase de la protéine RecQ d’ E. coli [
43]. Les motifs hélicase (en bleu) correspondent pour la plupart à des boucles, sont localisés à l’interface des deux lobes et constituent le site actif. |
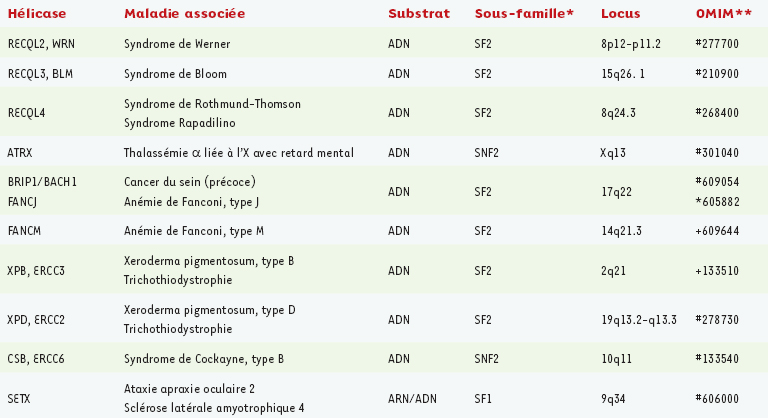
Les hélicases sont essentielles à de nombreux aspects du métabolisme des acides nucléiques, et participent aux processus de réplication, recombinaison, réparation de l’ADN, transcription, traduction et épissage de l’ARN. Chez l’homme, une cinquantaine d’hélicases ont été caractérisées par voie biochimique, et une dizaine d’entre elles sont associées à des pathologies (Encadré et Tableau I). Parmi celles-ci figurent la sénataxine, l’orthologue d’une hélicase à ARN de levure (Sen1p), qui joue un rôle dans la maturation de petits ARN nucléolaires, ainsi que six ADN hélicases qui font l’objet de cette revue : il s’agit des sous-unités XPB et XPD du facteur de transcription/réparation TFIIH, des hélicases de la famille RecQ, ainsi que de la protéine BRIP1/BACH1. Nous n’aborderons pas le cas des protéines CSB et ATRX, des membres de la famille SWI/SNF2 qui possèdent une activité ATPase dépendante de l’ADN, mais pas d’activité hélicase, et dont les dysfonctionnements peuvent être à l’origine du syndrome de Cockayne ou d’α-thalassémie.  | Tableau I.
Principales hélicases associées à des maladies rares.
|
|
XPB et XPD : réparation de l’ADN et transcription La réparation par excision-resynthèse de nucléotides (NER) permet de corriger les modifications de l’ADN à l’origine d’une distorsion de la double hélice, comme des adduits de cisplatine ou des photoproduits engendrés par les rayons UV qui bloquent la réplication de l’ADN et la transcription. Chez l’homme, une trentaine de protéines agissent de concert pour réaliser les différentes étapes de ce mécanisme de réparation : reconnaissance de la lésion, ouverture locale de la double hélice suivie de l’incision du brin endommagé et, enfin, re-synthèse du brin excisé (pour revue, voir [
7]). Des mutations dans les gènes codant pour ces protéines sont à l’origine de maladies génétiques autosomiques récessives comme le xeroderma pigmentosum (XP), la trichothiodystrophie (TTD), le syndrome de Cockayne (CS) ou le syndrome cérébro-oculo-facio-squelettique (COFS) (pour revue, voir [
8]). Dans le cas du XP, les patients ont une sensibilité de la peau au soleil augmentée, qui se traduit par des anomalies pigmentaires et une prédisposition élevée aux cancers de la peau. Des expériences de fusion cellulaire ont permis l’identification de huit groupes de complémentation pour les patients XP : de A à G, ainsi qu’un groupe XP-variant. À chacun de ces groupes correspond un gène codant pour un facteur indispensable au processus de réparation par NER (http://www.xpmutations.org/). Les groupes de complémentation XPB et XPD, pour lesquels 8 et 76 allèles ont respectivement été identifiés, ont fait l’objet de nombreux travaux. Les gènes XPB et XPD codent pour des protéines de 760 et 782 acides aminés présentant les 8 motifs conservés caractéristiques des enzymes de la famille SF2 [
9,
10]. Ces deux protéines, qui possèdent des activités ADN hélicases complémentaires (activités de polarité 3’→5’ pour XPB et 5’→3’ XPD), sont des sous-unités du facteur TFIIH [
11,
12], un complexe multiprotéique initialement identifié comme l’un des six facteurs généraux nécessaires à l’initiation de la transcription des gènes de classe II, et qui participe aussi aux processus cellulaires fondamentaux que sont l’apoptose, le cycle cellulaire et la transcription des gènes de classe I (pour revue, voir [
13,
14]). TFIIH est composé de 10 protéines, parmi lesquelles figurent la kinase dépendante des cyclines cdk7, intervenant dans la régulation du cycle cellulaire, ainsi que la protéine récemment identifiée TFB5/p8 [
15,
16], mutée chez les patients atteints d’une trichothiodystrophie appartenant au groupe de complémentation TTD-A. Ces sous-unités se répartissent en deux sous-complexes correspondant à des entités structurales et fonctionnelles : le cœur-TFIIH (composé des protéines p8, p34, p44, p52, p62 et de l’hélicase XPB) et le complexe CAK (comprenant la cdk7, la cycline H et MAT1) [
17–
19]. Le facteur XPD, qui peut être associé au cœur-TFIIH ou au complexe CAK, assure le lien entre les deux sous-complexes. XPB et XPD permettent la formation de structures ouvertes au cours de la réparation de l’ADN par NER, ainsi que lors de la transcription (Figure 2). Dans le cas de la réparation, les activités hélicase des protéines XPB et XPD sont essentielles [
20–
22] : elles assurent la formation d’une bulle de réparation qui s’étend sur une trentaine de paires de bases autour de la lésion. Cependant, pour assurer la formation d’un complexe ouvert sur lequel pourront agir les endonucléases XPG et XPF-ERCC1, TFIIH requiert l’assistance de facteurs participant à la reconnaissance de la lésion et à l’assemblage du complexe de réparation, tels que XPC-hR23B, XPA ou RPA. Lors de l’initiation de la transcription, TFIIH catalyse la formation d’un complexe ouvert, avant que débute la synthèse de l’ARN messager. TFIIH participe également à l’étape d’échappée du promoteur, lorsque le complexe de transcription passe de l’étape d’initiation à la phase d’élongation. Contrairement à ce qui est observé dans le cas de la réparation, où les activités catalytiques des protéines XPB et XPD sont toutes deux nécessaires, seule l’activité hélicase 3’→5’ de la protéine XPB est requise au cours de la transcription, la protéine XPD jouant essentiellement un rôle structural [
18,
23]. Par ailleurs, de récentes données suggèrent que seule l’activité ATPase de XPB est impliquée dans l’ouverture du promoteur, son activité hélicase étant ultérieurement utilisée lors de l’échappée du promoteur, pour le couplage entre transcription et maturation des ARN [
24].

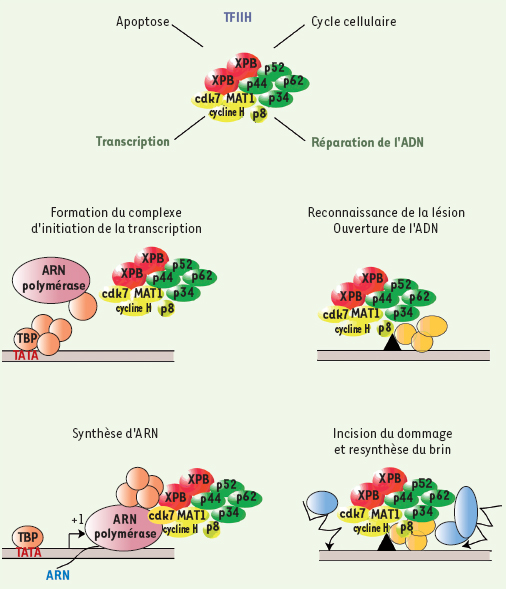
 | Figure 2.
Rôle des hélicases XPB et XBD. Les hélicases XPB et XPD sont des sous-unités du facteur de transcription/réparation TFIIH. Lors de l’initiation de la transcription, ces hélicases interviennent non seulement dans la formation de la bulle de transcription, mais également dans l’étape d’échappée du promoteur qui précède la phase d’élongation. Dans le cadre de la réparation de l’ADN par excision-resynthèse de nucléotides (NER), XPB et XPD assurent la formation d’un complexe de pré-incision ouvert sur lequel pourront agir les endonucléases XPG et XPF/ERCC1. |
Si la majorité des mutations dans les gènes XPB et XPD ont été identifiées chez des malades présentant un phénotype XP, de telles mutations sont également retrouvées chez des patients atteints de TTD (pour revue, voir [8,
25]). La mise en évidence du rôle de TFIIH dans la transcription et dans la réparation suggère que les phénotypes XP seraient une conséquence des mutations altérant la fonction de réparation de TFIIH, tandis que les symptômes observés chez les patients TTD ou CS reflèteraient un dérèglement de l’activité transcriptionelle de TFIIH [
26]. Ces hypothèses sont étayées par une analyse des mutations observées dans le gène XPD, qui montre que la grande majorité d’entre elles sont spécifiques d’un phénotype : la plupart des mutations TTD affectent les résidus R112, R658 et R577, alors que 70 % des mutations retrouvées chez les patients XP concernent l’acide aminé R683. Des études menées in vitro ont montré que toutes les mutations affectent la fonction hélicase de XPD, ce qui explique les défauts de réparation, et notamment la sensibilité des cellules des malades aux rayonnements UV. Alors que les mutations associées au phénotype XP (y compris les mutations qui inhibent totalement l’activité hélicase de XPD) n’ont pas d’effet sur la transcription, les mutations observées chez les patients TTD inhibent in vitro la transcription de base [
27] et diminuent in vivo la stabilité de TFIIH, dont la concentration intracellulaire peut être réduite de 70 % [
28]. Les effets des mutations TTD sur la transcription expliquent l’apparition de symptômes tels qu’une anomalie de la synthèse de protéines riches en soufre (les cheveux et ongles cassants, caractéristiques de la TTD, sont la conséquence d’une synthèse anormale de sulfures dans la kératine) ou de production de β-globine, des gènes devant être fortement exprimés étant insuffisamment transcrits [
29,
30]. De nombreuses mutations du gène XPD, et cela indépendamment du phénotype, ont également un effet sur la transcription activée [
31,
32]. Ainsi, en diminuant la stabilité du complexe et en fragilisant l’ancrage du CAK au cœur-TFIIH, elles affectent l’activité cdk7 kinase de TFIIH, ce qui a pour effet de réduire la phosphorylation de certains récepteurs nucléaires et donc de modifier la transactivation des gènes qui sont sous leur contrôle. Ce mécanisme fournit des pistes pour expliquer les déficiences neurologiques et les retards de développement observés chez des patients XP et TTD. Si notre compréhension des relations entre phénotype et génotype a considérablement progressé au cours de ces dernières années, de nombreuses questions subsistent. Elles concernent notamment les fondements moléculaires du vieillissement prématuré observé lorsque le gène XPD est muté [
33], ainsi que la relation entre prédisposition génétique au cancer et NER. La réponse à cette dernière interrogation permettrait de comprendre pourquoi les malades TTD des groupes de complémentation XPB et XPD ne développent pas de cancers cutanés, contrairement aux malades XP [
34]. |
Hélicases de la famille RecQ : recombinaison et réplication Les protéines de la famille RecQ ont été nommées ainsi en référence à la protéine bactérienne qui intervient dans la voie de recombinaison homologue RecF et dans la suppression de recombinaisons illégitimes. Chez l’homme, les gènes de cinq hélicases apparentées à RecQ ont été identifiés, et des mutations dans trois d’entre eux (WRN, BLM, RecQL4) sont à l’origine des syndromes de Werner (WS), de Bloom (BS) et de Rothmund-Thomson (RTS) [
35–
37]. Ces maladies autosomiques récessives rares sont caractérisées par un risque élevé de développer des tumeurs malignes et par des signes de vieillissement prématuré [
38]. Au niveau cellulaire, ces syndromes se caractérisent principalement par une instabilité génétique reliée à des anomalies de la réplication et de la recombinaison : recombinaisons illégitimes et grandes délétions chromosomiques dans le cas du syndrome de Werner, fréquence élevée de recombinaisons homologues, incluant des échanges réciproques entre chromatides sœurs et chromosomes homologues, pour le syndrome de Bloom (pour revue, voir [
39]). La fréquence élevée d’échanges entre chromatides sœurs (événements de croisement de brin) peut conduire à une perte d’hétérozygotie et à des aberrations chromosomiques comme des délétions, inversions ou translocations. Dans le cas du syndrome de Werner, l’instabilité génétique explique vraisemblablement la fréquence de cancers observée ; quant au large spectre clinique des troubles directement liés à la sénescence tels que l’athérosclérose, le diabète, l’ostéoporose ou la cataracte, il traduit les nombreuses implications de l’hélicase WRN dans la réplication de l’ADN et la ségrégation des chromosomes. Les protéines RecQ, et en particulier les protéines WRN, BLM et RecQL4, possèdent des activités hélicase de polarité 3’→5’ [
40–
42]. Outre leur domaine hélicase (dont une structure tridimensionnelle est représentée dans la Figure 1 [43]), les protéines RecQ présentent pour la plupart une région conservée composée de deux modules additionnels : le domaine RQC, qui intervient dans le recrutement de partenaires protéiques et dont un motif hélice-boucle-hélice est capable de fixer l’ADN double brin, et le domaine HRDC, dont la topologie est proche de celle trouvée dans d’autres protéines participant également au métabolisme de l’ADN, et qui permettrait la formation de contacts spécifiques avec des intermédiaires de recombinaison [
44]. Des membres de cette famille, dont la protéine WRN [
45] et la protéine FFA 1 de xénope, possèdent en outre un domaine exonucléase. Contrairement aux hélicases XPB et XPD, les fonctions moléculaires des protéines RecQ font encore l’objet de nombreuses interrogations. La caractérisation in vitro des substrats des hélicases RecQ a fourni des pistes pour comprendre le rôle de ces enzymes dans un contexte cellulaire. Toutes les protéines de cette famille possèdent une activité hélicase de polarité 3’→5’. L’analyse des cinétiques de réaction a montré que ces enzymes utilisent préférentiellement des substrats qui miment des fourches de réplication, des bulles ou des jonctions de Holliday (pour revue, voir [
46]). Leur activité hélicase participe également à la résolution de structures à 4 brins appelées G-quadruplex [
47] ; de telles structures, formées par des séquences riches en guanine et stabilisées par des liaisons hydrogène de type Hoogsteen, sont trouvées entre autres au niveau de l’ADN télomérique et de triplets CGG associés au syndrome de l’X-fragile. Des données, corrélées à l’étude des réseaux d’interactions protéine-protéine (Tableau II), suggèrent que les hélicases RecQ interviennent dans les mécanismes de réplication, où elles participeraient à la stabilisation de la fourche de réplication après un dommage (protéine bactérienne RecQ et hélicase de levure sgs1 p [
48,
49]), ainsi que dans la recombinaison, où elles permettraient de résoudre des intermédiaires réactionnels et la suppression des échanges de brins (chez l’homme, des intermédiaires de réplication aberrants sont ainsi accumulés dans les lignées cellulaires issues de patients WRN et BLM). Ces hélicases interagissent physiquement et fonctionnellement avec de nombreuses protéines dont le rôle dans la réplication est connu (topo-isomérases I et III, protéine RPA, PCNA, endonucléase FEN-1 (qui participe au traitement des fragments d’Okasaki), ADN polymérase δ [39,
50]. Les mécanismes de recombinaison jouent un rôle majeur dans la réparation des cassures double brin qui surviennent, notamment, lorsque la fourche de réplication est endommagée. De nombreux travaux menés sur les protéines BLM, WRN et l’hélicase de levure sgs1, illustrent la participation des hélicases RecQ aux mécanismes de recombinaison homologue [
51]. Des études réalisées in vitro ont montré que les actions combinées de l’hélicase BLM et de la topo-isomérase III permettent la résolution de doubles jonctions de Holliday sans créer de croisement de brin, et fournissent une explication moléculaire aux fréquences élevées d’échanges entre chromatides sœurs observées chez les patients atteints du syndrome de Bloom [
52]. Des évidences génétiques suggèrent un rôle similaire pour la protéine WRN, mais aucune fonction moléculaire précise n’a pu encore être mise en évidence (pour revue, voir [
53]).  | Tableau II.
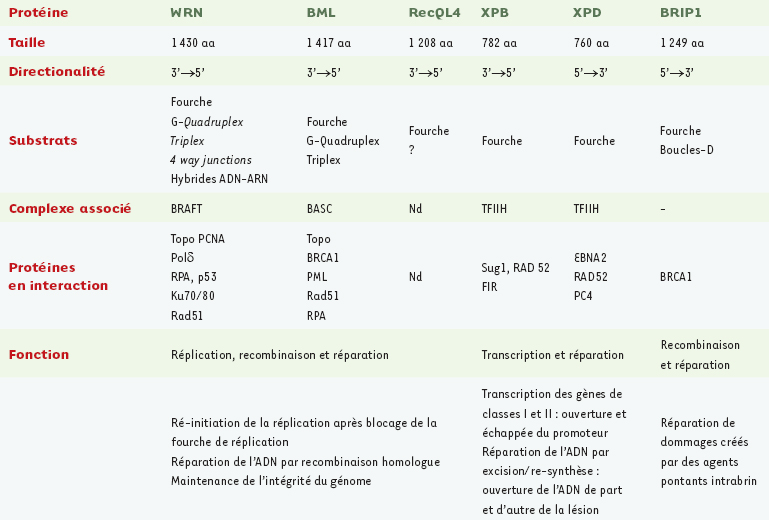
Caractéristiques biochimiques de quelques hélicases.
|
Un rôle dans les mécanismes de réparation par jonction non homologue, de stabilisation des télomères et de surveillance du génome a également été proposé pour ces hélicases de la famille RecQ. Concernant ce dernier point, il a été montré que la protéine BLM appartient au complexe BASC impliqué dans la reconnaissance de structures d’ADN aberrantes [
54]. De plus, les protéines BLM et WRN interagissent avec la protéine p53, et la formation de ce complexe inhibe leur activité hélicase ainsi que l’activité exonucléase de WRN [
55]. Il est possible que les protéines RecQ coordonnent diverses voies de réponse aux dommages et de réparation, et jouent le rôle de transducteur. Par exemple, elles participeraient à la reconnaissance de différents types de lésions (bases modifiées, coupure simple brin ou coupure double brin) et pourraient déclencher l’activité de la voie de réparation appropriée : réparation des coupures simple brin, réparation par excision de bases, recombinaison homologue ou réparation par jonction non homologue [46]. |
L’hélicase BRIP1/BACH1 et la protéine FANCJ ne font qu’un Le dernier exemple d’hélicase associée à une maladie rare est celui de la protéine BRIP1/BACH1, mutée chez des patients souffrant d’anémie de Fanconi. Cette pathologie, qui se manifeste par une insuffisance médullaire associée à des anomalies physiques et à une prédisposition au cancer, est caractérisée au niveau cellulaire par une sensibilité élevée aux agents pontants interbrins (mitomycine C, cis-platine) [
56]. Aujourd’hui, 11 groupes de complémentation, ayant pour origine des mutations dans des gènes (FANCA, C, D1 [BRCA2], D2, E, F, G, I, J, L et M) distincts, sont connus. Sept des protéines FANC forment un complexe nucléaire, auquel peuvent se trouver associées les protéines RPA, la topo-isomérase IIIα ainsi que l’hélicase BLM (voir plus haut). Les gènes FANCJ et FANCM, récemment identifiés, codent pour des protéines présentant des motifs hélicase. Le produit du gène FANCM, FAAP250, est homologue à d’autres protéines intervenant dans les mécanismes de réparation de l’ADN, comme la protéine d’archae bactérie Hef, la protéine de levure MPH1 ou la protéine humaine ERCC4/XPF [54]. FANCM ne possède pas d’activité hélicase avec des substrats double brin, mais peut dissocier une triple hélice, ce qui pourrait refléter une activité translocase [
57]. Le gène FANCJ code, quant à lui, pour une ADN hélicase, également connue sous le nom de BRIP1/BACH1, une protéine interagissant avec la protéine suppresseur de tumeur BRCA1 [
58]. FANCJ possède une activité hélicase de polarité 5’→3’ et utilise préférentiellement des fourches, ainsi que des substrats à 3 brins mimant des intermédiaires de recombinaison (boucles-D). Le lien entre le gène BRIP1/BACH1 et l’anémie de Fanconi a été établi par la découverte de mutations dans ce gène chez des patients souffrant de cette maladie [
59,
60]. D’autre part, chez le poulet, l’inactivation du gène brip1 conduit à une forte sensibilité au cis-platine, un agent pontant utilisé pour le diagnostic de l’anémie de Fanconi [
61]. Cette sensibilité peut être corrigée par une version tronquée, incapable d’interagir avec BRCA1, mais ne peut pas l’être avec un mutant ponctuel inactif, démontrant ainsi que BRIP1 fonctionne indépendamment de la voie BRCA1 et que l’activité hélicase de BRIP1/BACH1 est essentielle pour la réparation des dommages causés par des agents pontants. |
Les hélicases sont connues pour jouer un rôle essentiel dans presque tous les aspects du métabolisme des acides nucléiques et pour permettre l’accès à des formes simple brin. Elles sont généralement intégrées au sein de complexes multiprotéiques, ce qui permet une modulation de leurs activités catalytiques et l’orientation des spécificités de substrats. Les progrès réalisés en génétique humaine ont permis d’identifier un ensemble de syndromes ayant pour origine le dysfonctionnement d’hélicases à ADN, dont cette revue a fait l’objet. On peut maintenant entrevoir les fondements moléculaires de ces pathologies et, dans certains cas, comprendre pourquoi une mutation dans une hélicase peut causer une instabilité génétique ou être à l’origine d’une prédisposition au cancer. Cependant, bien des interrogations subsistent, concernant notamment les interactions avec leurs partenaires protéiques, les structures d’ADN créées par les différents types de dommages qui sont effectivement résolues in vivo, ainsi que les liens qui existent entre les mécanismes de réparation, la régulation du cycle cellulaire et l’apotose. Une étude approfondie des fonctions de ces hélicases devrait non seulement permettre d’identifier de nouveaux outils ou de nouvelles cibles pour prévenir ou diagnostiquer les maladies qui les accompagnent, mais aussi avoir des implications importantes pour aborder la question du vieillissement dans la population générale, ou mieux comprendre les processus de tumorigenèse.

|
Les auteurs remercient A.C. Dock-Bregeon, Jean-Marc Egly et Dino Moras pour les nombreuses et enrichissantes discussions partagées sur le sujet. Le travail de notre laboratoire est soutenu par le CNRS, l’Inserm et l’Université Louis-Pasteur. Muriel Uhring a bénéficié d’une bourse de l’Association pour la recherche sur le cancer.
|
Footnotes |
1. Tuteja N, Tuteja R. Prokaryotic and eukaryotic DNA helicases. Essential molecular motor proteins for cellular machinery. Eur J Biochem 2004; 271 : 1835–48. 2. Gorbalenya AE, Koonin EV, Donchenko AP, et al. Two related superfamilies of putative helicases involved in replication, recombination, repair and expression of DNA and RNA genomes. Nucleic Acids Res 1989; 17 : 4713–30. 3. Subramanya HS, Bird LE, Brannigan JA, et al. Crystal structure of a DExx box DNA helicase. Nature 1996; 384 : 379–83. 4. Cordin O, Tanner NK, Doere M, et al. The newly discovered Q motif of DEAD-box RNA helicases regulates RNA-binding and helicase activity. EMBO J 2004; 23 : 2478–87. 5. Caruthers JM, McKay DB. Helicase structure and mechanism. Curr Opin Struct Biol 2002; 12 : 123–33. 6. von Hippel PH, Delagoutte E. Macromolecular complexes that unwind nucleic acids. Bioessays 2003; 25 : 1168–77. 7. Dip R, Camenisch U, Naegeli H. Mechanisms of DNA damage recognition and strand discrimination in human nucleotide excision repair. DNA Repair (Amst) 2004; 3 : 1409–23. 8. Lehmann AR. DNA repair-deficient diseases, xeroderma pigmentosum, Cockayne syndrome and trichothiodystrophy. Biochimie 2003; 85 : 1101–11. 9. Weeda G, van Ham RC, Vermeulen W, et al. A presumed DNA helicase encoded by ERCC-3 is involved in the human repair disorders xeroderma pigmentosum and Cockayne’s syndrome. Cell 1990; 62 : 777–91. 10. Weber CA, Salazar EP, Stewart SA, et al. ERCC2 : cDNA cloning and molecular characterization of a human nucleotide excision repair gene with high homology to yeast RAD3. EMBO J 1990; 9 : 1437–47. 11. Schaeffer L, Roy R, Humbert S, et al. DNA repair helicase : a component of BTF2 (TFIIH) basic transcription factor. Science 1993; 260 : 58–63. 12. Schaeffer L, Moncollin V, Roy R, et al. The ERCC2/DNA repair protein is associated with the class II BTF2/TFIIH transcription factor. EMBO J 1994; 13 : 2388–92. 13. Egly JM. The 14th Datta lecture. TFIIH : from transcription to clinic. FEBS Lett 2001; 498 : 124–8. 14. Zurita M, Merino C. The transcriptional complexity of the TFIIH complex. Trends Genet 2003; 19 : 578–84. 15. Giglia-Mari G, Coin F, Ranish JA, et al. A new, tenth subunit of TFIIH is responsible for the DNA repair syndrome trichothiodystrophy group A. Nat Genet 2004; 36 : 714–9. 16. Coin F, Proietti DS, Nardo T, et al. p8/TTD-A as a repair-specific TFIIH subunit. Mol Cell 2006; 21 : 215–26. 17. Reardon JT, Ge H, Gibbs E, et al. Isolation and characterization of two human transcription factor IIH (TFIIH)-related complexes : ERCC2/CAK and TFIIH. Proc Natl Acad Sci USA 1996; 93 : 6482–7. 18. Tirode F, Busso D, Coin F, et al. Reconstitution of the transcription factor TFIIH : assignment of functions for the three enzymatic subunits XPB, XPD and cdk7. Mol Cell 1999; 3 : 87–95. 19. Schultz P, Fribourg S, Poterszman A, et al. Molecular structure of human TFIIH. Cell 2000; 102 : 599–607. 20. Araujo SJ, Tirode F, Coin F, et al. Nucleotide excision repair of DNA with recombinant human proteins : definition of the minimal set of factors, active forms of TFIIH, and modulation by CAK. Genes Dev 2000; 14 : 349–59. 21. Winkler GS, Araujo SJ, Fiedler U, et al. TFIIH with inactive XPD helicase functions in transcription initiation but is defective in DNA repair. J Biol Chem 2000; 275 : 4258–66. 22. Coin F, Auriol J, Tapias A, et al. Phosphorylation of XPB helicase regulates TFIIH nucleotide excision repair activity. EMBO J 2004; 23 : 4835–46. 23. Bradsher J, Coin F, Egly JM. Distinct roles for the helicases of TFIIH in transcript initiation and promoter escape. J Biol Chem 2000; 275 : 2532–8. 24. Lin YC, Choi WS, Gralla JD. TFIIH XPB mutants suggest a unified bacterial-like mechanism for promoter opening but not escape. Nat Struct Mol Biol 2005; 12 : 603–7. 25. Itin PH, Sarasin A, Pittelkow MR. Trichothiodystrophy : update on the sulfur-deficient brittle hair syndromes. J Am Acad Dermatol 2001; 44 : 891–920. 26. Bootsma D, Hoeijmakers JH. DNA repair. Engagement with transcription. Nature 1993; 363 : 114–5. 27. Dubaele S, Proietti DS, Bienstock RJ, et al. Basal transcription defect discriminates between xeroderma pigmentosum and trichothiodystrophy in XPD patients. Mol Cell 2003; 11 : 1635–46. 28. Botta E, Nardo T, Lehmann AR, et al. Reduced level of the repair/transcription factor TFIIH in trichothiodystrophy Hum Mol Genet 2002; 11 : 2919–28. 29. de Boer J, van Steeg H, Berg RJ, et al. Mouse model for the DNA repair/basal transcription disorder trichothiodystrophy reveals cancer predisposition. Cancer Res 1999; 59 : 3489–94. 30. Viprakasit V, Gibbons RJ, Broughton BC, et al. Mutations in the general transcription factor TFIIH result in beta-thalassaemia in individuals with trichothiodystrophy. Hum Mol Genet 2001; 10 : 2797–802. 31. Liu J, He L, Collins I, et al. The FBP interacting repressor targets TFIIH to inhibit activated transcription. Mol Cell 2000; 5 : 331–41. 32. Keriel A, Stary A, Sarasin A, et al. XPD mutations prevent TFIIH-dependent transactivation by nuclear receptors and phosphorylation of RARa. Cell 2002; 109 ; 125–35. 33. de Boer J, Andressoo JO, de Wit J, et al. Premature aging in mice deficient in DNA repair and transcription. Science 2002; 296 : 1276–9. 34. Da Costa RM, Riou L, Paquola A, et al. Transcriptional profiles of unirradiated or UV-irradiated human cells expressing either the cancer-prone XPB/CS allele or the noncancer-prone XPB/TTD allele. Oncogene 2005; 24 : 1359–74. 35. Yu CE, Oshima J, Fu YH, et al. Positional cloning of the Werner’s syndrome gene. Science 1996; 272 : 258–62. 36. Ellis NA, Groden J, Ye TZ, et al. The Bloom’s syndrome gene product is homologous to RecQ helicases. Cell 1995; 83 : 655–66. 37. Kitao S, Shimamoto A, Goto M, et al. Mutations in RECQL4 cause a subset of cases of Rothmund-Thomson syndrome Nat Genet 1999; 22 : 82–4. 38. Mohaghegh P, Hickson ID. DNA helicase deficiencies associated with cancer predisposition and premature ageing disorders Hum Mol Genet 2001; 10 : 741–6. 39. Hickson ID. RecQ helicases : caretakers of the genome. Nat Rev Cancer 2003; 3 : 169–78. 40. Gray MD, Shen JC, Kamath-Loeb AS, et al. The Werner syndrome protein is a DNA helicase. Nat Genet 1997; 17 : 100–3. 41. Karow JK, Chakraverty RK, Hickson ID. The Bloom’s syndrome gene product is a 3’→5’ DNA helicase. J Biol Chem 1997; 272 : 30611–4. 42. Macris MA, Krejci L, Bussen W, et al. Biochemical characterization of the RECQ4 protein, mutated in Rothmund-Thomson syndrome. DNA Repair (Amst) 2006 : 5 : 172–80. 43. Bernstein DA, Zittel MC, Keck JL. High-resolution structure of the E. coli RecQ helicase catalytic core. EMBO J 2003; 22 : 4910–21. 44. Bernstein DA, Keck JL. Conferring substrate specificity to DNA helicases : role of the RecQ HRDC domain. Structure (Camb) 2005; 13 : 1173–82. 45. Huang S, Li B, Gray MD, et al. The premature ageing syndrome protein, WRN, is a 3’→5’ exonuclease. Nat Genet 1998; 20 : 114–6. 46. Opresko PL, Cheng WH, Bohr VA. Junction of RecQ helicase biochemistry and human disease. J Biol Chem 2004; 279 : 18099–102. 47. Li JL, Harrison RJ, Reszka AP, et al. Inhibition of the Bloom’s and Werner’s syndrome helicases by G-quadruplex interacting ligands. Biochemistry 2001; 40 : 15194–202. 48. Courcelle J, Donaldson JR, Chow KH, et al. DNA damage-induced replication fork regression and processing in Escherichia coli. Science 2003; 299 : 1064–7. 49. Cobb JA, Bjergbaek L, Shimada K, et al. DNA polymerase stabilization at stalled replication forks requires Mec1 and the RecQ helicase Sgs1. EMBO J 2003; 22 : 4325–36. 50. Khakhar RR, Cobb JA, Bjergbaek L, et al. RecQ helicases : multiple roles in genome maintenance. Trends Cell Biol 2003; 13 : 493–501. 51. Laursen LV, Bjergbaek L, Murray JM, et al. RecQ helicases and topoisomerase III in cancer and aging. Biogerontology 2003; 4 : 275–87. 52. Wu L, Hickson ID. The Bloom’s syndrome helicase suppresses crossing over during homologous recombination. Nature 2003; 426 : 870–4. 53. Ozgenc A, Loeb LA. Current advances in unraveling the function of the Werner syndrome protein. Mutat Res 2005; 577 : 237–51. 54. Meetei AR, Medhurst AL, Ling C, et al. A human ortholog of archaeal DNA repair protein Hef is defective in Fanconi anemia complementation group M. Nat Genet 2005; 37 : 958–63. 55. Yang Q, Zhang R, Wang XW, et al. The processing of Holliday junctions by BLM and WRN helicases is regulated by p53. J Biol Chem 2002; 277 : 31980–7. 56. Papadopoulo D, Moustacchi E. L’anémie de Fanconi : gènes et fonction(s) revisités. Med Sci (Paris) 2005; 21 : 730–6. 57. Mosedale G, Niedzwiedz W, Alpi A, et al. The vertebrate Hef ortholog is a component of the Fanconi anemia tumor-suppressor pathway. Nat Struct Mol Biol 2005; 12 : 763–71. 58. Cantor SB, Bell DW, Ganesan S, et al. BACH1, a novel helicase-like protein, interacts directly with BRCA1 and contributes to its DNA repair function. Cell 2001; 105 : 149–60. 59. Levran O, Attwooll C, Henry RT, et al. The BRCA1-interacting helicase BRIP1 is deficient in Fanconi anemia. Nat Genet 2005; 37 : 931–3. 60. Levitus M, Waisfisz Q, Godthelp BC, et al. The DNA helicase BRIP1 is defective in Fanconi anemia complementation group J. Nat Genet 2005; 37 : 934–5. 61. Bridge WL, Vandenberg CJ, Franklin RJ, et al. The BRIP1 helicase functions independently of BRCA1 in the Fanconi anemia pathway for DNA crosslink repair. Nat Genet 2005; 37 : 953–7. |