| |
| Med Sci (Paris). 2006 December; 22(12): 1061–1068. Published online 2006 December 15. doi: 10.1051/medsci/200622121061.La Carte d’Haplotype du génome humain Une révolution en génétique des maladies à hérédité complexe Alexandre Montpetit* and Fanny Chagnon* Centre d’Innovation de Génome Québec et de l’Université McGill, 740 avenue Dr Penfield, Montréal, Québec, H3A 1A4 Canada |
Depuis la mise au point de la technique de clonage positionnel, vers la fin des années 80, et surtout depuis l’obtention de la séquence complète du génome humain, la découverte de gènes à l’origine de maladies génétiques rares ou mendéliennes a augmenté en flèche pour atteindre plus de mille en 2005 (base de données OMIM : http://www.ncbi.nlm.nih.gov/entrez/query.fcgi ?db=OMIM). Les tentatives pour adapter la technique aux maladies à hérédité complexe telles que l’asthme, le diabète, les maladies cardiovasculaires ou le cancer se sont avérées plus décevantes. Dans ces maladies, le bagage génétique, ainsi que d’autres facteurs tels que l’environnement, contribuent à divers degrés à l’étiologie. Des obstacles attribuables notamment à la faible puissance des analyses de liaison, à la trop grande étendue des régions chromosomiques à analyser ou à une difficulté à reproduire les observations, ont considérablement modéré les aspirations des généticiens [
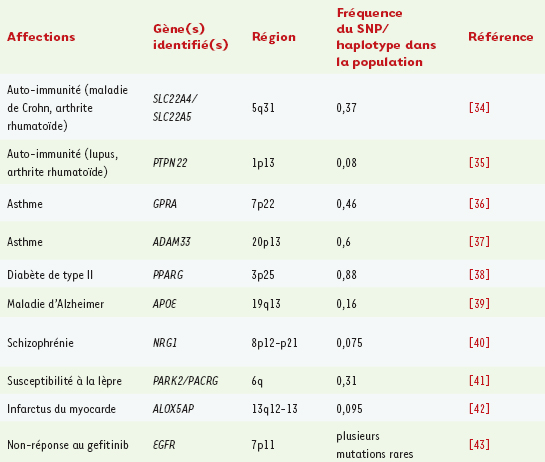
1]. L’autre approche employée, celle dite des gènes candidats, présente le désavantage de se limiter aux gènes dont la fonction est déjà établie. Tout de même, certains succès ont été obtenus selon ces modalités (Tableau I).  | Tableau I.
Exemples de gènes impliqués dans des maladies complexes trouvés à l’aide d’études d’association.
|
La majorité des chercheurs s’entendent pour dire que les maladies complexes sont souvent causées par des variations communes dans la population (ils adhèrent à l’hypothèse du common variant-common disease) [
2]. Les quelques gènes identifiés jusqu’ici tendent à confirmer cette hypothèse [
3] (Tableau 1). On estime qu’il existe dans le génome plus de sept millions de variations communes (fréquence de plus de 5 % dans la population), la plupart étant des variations d’une seule base, soit les polymorphismes nucléotidiques simples (SNP) [
4]. L’étude systématique de toutes les variations communes du génome dans une étude d’association permettrait donc, en principe, de découvrir tous les gènes de susceptibilité aux maladies complexes. En réalité, plusieurs facteurs vont limiter les succès d’une telle approche, notamment le risque relatif (RR) de celles-ci (c’est-à-dire le degré de la perturbation qu’elles causent), la fréquence de ces variations dans la population, et surtout, le coût prohibitif de telles études. Le déséquilibre de liaison (DL) mesure la fréquence avec laquelle les allèles de deux polymorphismes se retrouvent sur le même chromosome ou haplotype. Il est affecté par la recombinaison méiotique et diminue proportionnellement à la distance séparant les polymorphismes (pour revue, voir [
5]). Dans une étude d’association, même si la variation qui contribue à la maladie n’est pas testée, la présence d’un ou des SNP en DL avec le SNP causatif permettra d’identifier la région chromosomique comportant cette variation. En 1999, les modèles de population permettaient d’estimer que le DL utile ne devait pas dépasser 3 kb [
6]. Cependant, en 2001 et 2002, quelques études ont démontré qu’à petite échelle, le DL ne diminuait pas de façon graduelle comme on le croyait précédemment, mais plutôt par sauts brusques, suggérant que le génome est organisé en blocs. Ces blocs mesurent en moyenne 10 à 20 kb, mais varient de quelques kilobases à plusieurs centaines de kilobases et sont séparés par des petites régions d’environ 1-2 kb comportant des taux de recombinaison élevés, soit les points chauds de recombinaison (recombination hotspots). De plus, la diversité haplotypique (soit les différentes combinaisons possibles des allèles de chaque SNP) y est très faible et leur structure est similaire au sein de toutes les populations humaines [
7–
10]. Cette découverte impliquait que la recombinaison ne survenait pas de façon aléatoire dans le génome. Donc, en principe, si l’on connaissait la structure haplotypique sous-jacente (soit la structure des blocs d’haplotypes), il serait possible de réduire considérablement le nombre de SNP à utiliser pour les études d’association. Le projet International de la Carte d’Haplotype du Génome Humain (HapMap) est issu de ces observations.

|
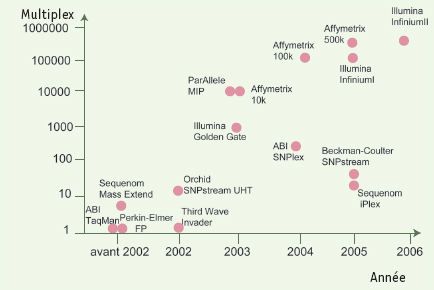
Technologies de génotypage L’avènement de nouvelles technologies permettant de génotyper plusieurs SNP en parallèle a grandement facilité la réalisation d’un tel projet. Pour sa part, le projet a contribué à l’augmentation du débit de ces technologies et à la diminution du coût des opérations de génotypage, de façon similaire à ce qui avait été observé lors du projet de séquençage du génome humain (Figure 1). Le coût par génotype est passé de près de 0,50 $US en 2002, lors de l’annonce du projet HapMap, à moins de 0,01 $ à son terme ouvrant la voie à l’idée d’envisager des études d’association à l’échelle du génome. Le Consortium International HapMap était constitué de neuf groupes répartis aux quatre coins du Globe : ils se sont partagés la tâche et ont eu recours à sept technologies différentes de génotypage (Tableau II). Trois exercices de contrôle de la qualité ont été effectués en cours de route. Ces exercices avaient pour but de s’assurer que la qualité des génotypes publiés était équivalente au sein de tous les Centres participant. Pour mener le projet à terme, le Consortium a aussi financé la découverte de plusieurs millions de SNP en reséquençant aléatoirement l’ADN de 16 personnes. Ainsi, entre 2001 et 2005, le nombre de SNP dans les bases de données est passé de moins de 1 million à plus de 9 millions, dont plus de la moitié ont été validés, la plupart grâce au projet HapMap (base de données dbSNP : http://www.ncbi.nlm.nih.gov/entrez/query.fcgi ?db=dbSNP).  | Figure 1.
Évolution des technologies de génotypage disponibles commercialement. La progression du nombre de SNP pouvant être analysés en parallèle (multiplex) des technologies de génotypage depuis 2002 est présentée. Au début du projet, en 2002, aucune technologie disponible commercialement ne permettait de génotyper plus d’une dizaine de SNP à la fois. La principale limitation des technologies à faible débit est la réaction de PCR qui ne peut être aisément multiplexée au-delà de 20 en raison des interactions entre amorces. Les technologies à haut débit n’ont pas cette limitation. La réaction permettant de discriminer les deux allèles d’un SNP est soit effectuée directement sur l’ADN génomique (Illumina, ParAllele) ou encore l’ADN génomique est coupé en petits morceaux qui sont ligués à des amorces (Affymetrix). Les molécules sont par la suite amplifiées par PCR grâce à des amorces universelles puis séparées sur un réseau et détectées par fluorescence. Pour l’approche de la compagnie Perlegen, tout le génome est initialement amplifié grâce à une série de longs PCR d’environ 10 kb (pour revue, voir [
33]). |
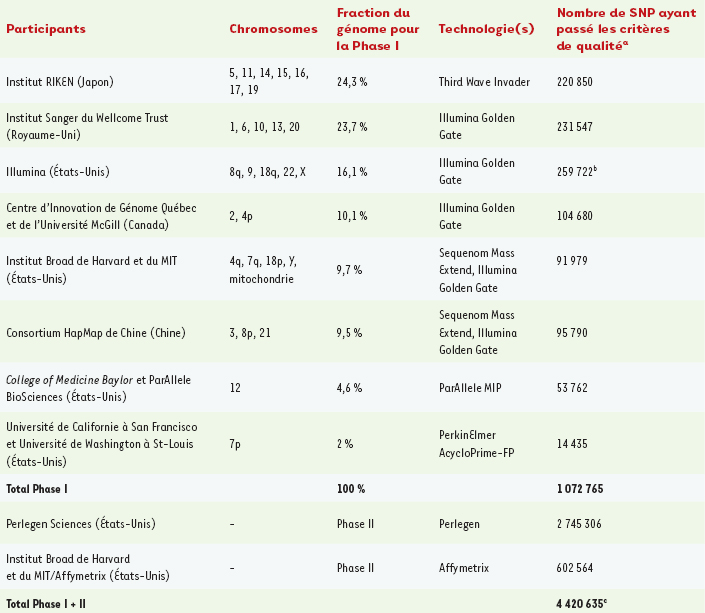
 | Tableau II.
Participants au projet HapMap et technologies utilisées.
|
|
 La Phase I contient un peu plus de un million de SNP polymorphiques dans au moins une population et ayant passé tous les contrôles de qualité (les SNP doivent avoir un taux de succès d’au moins 80 %, être en équilibre Hardy-Weinberg à p > 0,001 et avoir au plus une erreur de transmission mendélienne sur 30 familles ou une erreur de reproductibilité sur 5 réplicats) [
12]. Au total, 1,3 million de SNP ont été génotypés au cours du projet sur les 269 échantillons. Grâce aux exercices de contrôle de la qualité, on estime que le taux d’erreur ne dépasse pas les 0,3 %. Tous les résultats de génotypage ont été publiés sur le site internet du projet (http://www.hapmap.org). Lors de la Phase II, près de trois millions de SNP supplémentaires ont été génotypés au sein de tout le génome, principalement par la compagnie Perlegen, triplant ainsi la densité existante de la Carte. Cette entreprise avait précédemment publié une carte d’haplotype du génome de 1,5 million de SNP à partir de 71 personnes d’origine européenne, africaine-américaine et asiatique [
13]. Les résultats de cette carte sont très comparables à ceux obtenus par le Consortium International, et ce, malgré un plus faible nombre d’échantillons. En effet, un nombre plus restreint limite les interprétations susceptibles d’être tirées des haplotypes plus rares, soit ceux dont les fréquences sont inférieures à 10 %. La Phase I contient un peu plus de un million de SNP polymorphiques dans au moins une population et ayant passé tous les contrôles de qualité (les SNP doivent avoir un taux de succès d’au moins 80 %, être en équilibre Hardy-Weinberg à p > 0,001 et avoir au plus une erreur de transmission mendélienne sur 30 familles ou une erreur de reproductibilité sur 5 réplicats) [
12]. Au total, 1,3 million de SNP ont été génotypés au cours du projet sur les 269 échantillons. Grâce aux exercices de contrôle de la qualité, on estime que le taux d’erreur ne dépasse pas les 0,3 %. Tous les résultats de génotypage ont été publiés sur le site internet du projet (http://www.hapmap.org). Lors de la Phase II, près de trois millions de SNP supplémentaires ont été génotypés au sein de tout le génome, principalement par la compagnie Perlegen, triplant ainsi la densité existante de la Carte. Cette entreprise avait précédemment publié une carte d’haplotype du génome de 1,5 million de SNP à partir de 71 personnes d’origine européenne, africaine-américaine et asiatique [
13]. Les résultats de cette carte sont très comparables à ceux obtenus par le Consortium International, et ce, malgré un plus faible nombre d’échantillons. En effet, un nombre plus restreint limite les interprétations susceptibles d’être tirées des haplotypes plus rares, soit ceux dont les fréquences sont inférieures à 10 %. |
En appliquant la méthode de Gabriel [10] sur les données de la Phase I, on peut calculer que plus de 80 % du génome est caractérisé par des blocs dans les populations non-africaines ; cette proportion chute à 67 % chez les YRI. La longueur moyenne de ces blocs est de 7 kb chez les YRI et environ le double dans les autres populations [12]. Dans toutes les populations, les blocs d’haplotypes les plus grands sont situés près des centromères où l’on avait déjà observé un plus faible taux de recombinaison. La diversité haplotypique est en général plus grande chez les YRI comparativement aux populations ayant quitté le continent africain et qui ont subi un effet de goulot d’étranglement (bottleneck). Plusieurs algorithmes ont été développés afin de décrire les blocs d’haplotypes. Cependant, étant donné l’apparition de mutations récurrentes, de conversion génique ou de l’hétérogénéité dans la population, ces patrons de recombinaison sont rarement bien définis. Une des conséquence est que plus la densité de SNP augmente, plus le nombre de blocs augmente et plus les frontières prédites par les différents algorithmes varient [
14]. De nouveaux outils d’analyses comme les cartes LDU (LD units) [
15] ou les cartes d’estimation du taux de recombinaison avec des SNP [
16] sont plus robustes et moins sensibles à la variation de densité ou de fréquence allélique : elles permettent notamment de localiser plus précisément les points chauds de recombinaison. On peut ainsi observer que 50 % de la recombinaison totale survient dans 10 % de la séquence du génome. Dans la région du complexe majeur d’histocompatibilité (CMH), c’est 80 % de la recombinaison qui est observée dans moins de 10 % de la séquence [16]. À l’échelle du génome, on observe que la présence de gènes est intimement liée à la recombinaison puisque le taux de celle-ci augmente proportionnellement à la distance du début du gène le plus rapproché. En revanche, il est intéressant de constater que, si l’on divise le génome en quatre selon le taux de recombinaison local, un plus grand nombre de gènes sont associés aux régions extrêmes qu’aux sections moyennes [12]. Par exemple, les gènes liés à l’immunité ou aux processus neurologiques, qui ont une grande diversité, sont en général associés aux régions de haut taux de recombinaison tandis que les gènes liés au cycle cellulaire et à la réparation de l’ADN sont associés aux régions de faible taux de recombinaison et comptent une diversité plus faible. Des corrélations positives entre le taux de recombinaison et les taux de GC, de polymorphismes ou de séquences répétées ont également été observées. Toutes ces observations confortent la thèse selon laquelle la recombinaison contribue à l’évolution du génome [12]. Une analyse de Myers et collaborateurs [
17] a pu démontrer que certains motifs étaient associés de façon significative aux points chauds de recombinaison. Par exemple, les éléments répétés THE1A et 1B ayant un motif CCTCCCT au lieu de CCTCCCC sont cinq fois plus représentés dans les points chauds de recombinaison, étant présents dans environ 11 % des quelque 25 000 qui ont été répertoriés. En revanche, il est surprenant de constater que la localisation des points chauds de recombinaison chez le chimpanzé est très différente de celle de l’humain, malgré plus de 98 % d’identité entre les deux génomes [
18]. De plus, il a récemment été montré que les points chauds de recombinaison observés par le typage de spermatozoïdes diffèrent significativement de ceux observés dans la population générale [
19]. Cela impliquerait que l’activité et la position des points chauds est en constante évolution. D’ailleurs, il a récemment été suggéré que la recombinaison était associée directement à des événements de conversion génique et que cela pourrait contribuer à l’évolution du génome en transformant graduellement la séquence composant le point chaud [
20,
21]. Cependant, il faut aussi noter que le taux de recombinaison diffère beaucoup entre les hommes et les femmes [15]. Des analyses plus détaillées, à l’aide des données de la Phase II ainsi que des études comparatives chez d’autres primates, seront nécessaires pour évaluer de façon plus approfondie la signification de ces résultats. |
Sélection de SNP marqueurs L’objectif principal du projet HapMap était de permettre la détermination d’un ensemble de SNP marqueurs pour les études d’association. L’utilisation de blocs d’haplotypes à des fins pratiques pour la description de SNP marqueurs est limitée à cause de la redondance de l’information entre les blocs (le taux de recombinaison n’est pas de 100 % entre chaque bloc). La plupart des algorithmes couramment employés s’appuient plutôt sur une approche sans blocs, en utilisant une mesure du DL, r2
. Celle-ci varie, comme D’, avec la recombinaison mais aussi avec la fréquence allélique des polymorphismes, qui est en fait un reflet de leur ancienneté dans l’histoire d’une population. Deux SNP ayant un r2
de 1 sont totalement équivalents et l’un peut être utilisé comme substitut de l’autre (l’information contenue est équivalente). En pratique, un r2
de 0,8 est habituellement choisi dans les études d’association pour décrire deux SNP équivalents. Il n’y a pas de méthode universelle pour décrire le meilleur ensemble de SNP marqueurs, mais en utilisant un outil simple comme Tagger, on peut obtenir rapidement une liste de SNP marqueurs nécessaires pour couvrir une région chromosomique (http://www.broad.mit.edu/mpg/tagger/). Afin d’évaluer le taux de couverture du génome obtenu lors des deux Phases du projet, dix régions de 500 kb ont été re-séquencées chez 48 personnes de toutes origines puis tous les SNP identifiés ont été génotypés sur tous les échantillons HapMap [12]. À l’aide de ces régions, on peut extrapoler et considérer que si toutes les variations communes étaient connues, environ 600 000 SNP marqueurs seraient nécessaires pour couvrir le génome entier pour les populations non-africaines, ce qui correspond à 1 SNP marqueur par 5 kb. Pour la population YRI, 1 SNP marqueur par 2 kb serait nécessaire. En comparaison, une sélection aléatoire d’un nombre équivalent de SNP ne permettrait de couvrir qu’un peu plus de la moitié du génome. À l’aide de la même approche, on évalue que la Phase I de la Carte d’Haplotype permet d’englober environ 70 % de l’information contenue dans le génome tandis que la densité plus grande de la Phase II permet d’en ratisser plus de 90 %. En utilisant un seuil différent de DL (avec un r
2 < 0,8) ou à l’aide d’une méthode reposant sur des haplotypes plutôt que sur des SNP seuls, on peut augmenter l’efficacité des SNP marqueurs [
22]. Cependant, l’impact réel de ces paramètres dans des études d’association n’a pas encore été évalué. La diminution considérable des coûts de génotypage et l’arrivée de nouvelles technologies commerciales utilisant ces données permettent maintenant d’envisager d’étudier tout le génome à la fois. De telles études auront comme conséquence d’augmenter dramatiquement la complexité des analyses ainsi que le nombre de faux-positifs. Cependant, des stratégies d’analyse permettant d’enrichir les vrais positifs ont déjà été décrites [
23–
25]. Il reste à vérifier si ces méthodes seront adéquates sur des données réelles, mais déjà une étude d’association sur le génome entier a conduit à la découverte d’un gène de susceptibilité pour la dégénérescence maculaire liée à l’âge [
26]. Cependant, il faut noter que la composante génétique associée à cette maladie est assez élevée. |
Application de la Carte à d’autres populations Les résultats de la Carte d’Haplotype ont été obtenus à partir de 269 individus provenant de quatre populations distinctes. Naturellement se pose maintenant la grande question de sa capacité à décrire adéquatement les haplotypes d’individus provenant de populations différentes de celles ayant servi à créer la Carte. Plusieurs études ont démontré que l’on pourrait user des SNP marqueurs HapMap dans d’autres populations principalement à partir des populations caucasiennes ou asiatiques [
27–
31]. Par exemple les SNP marqueurs dérivés des CEU pouvaient décrire en moyenne plus de 90 % des allèles ayant une fréquence de plus de 5 % dans un échantillon d’Estonie [31]. En revanche, il a déjà été démontré que la taille de l’échantillon utilisée pour le projet HapMap n’est pas suffisante pour répertorier efficacement les allèles dont la fréquence est inférieure à ce seuil, et ce, peu importe la population [31,
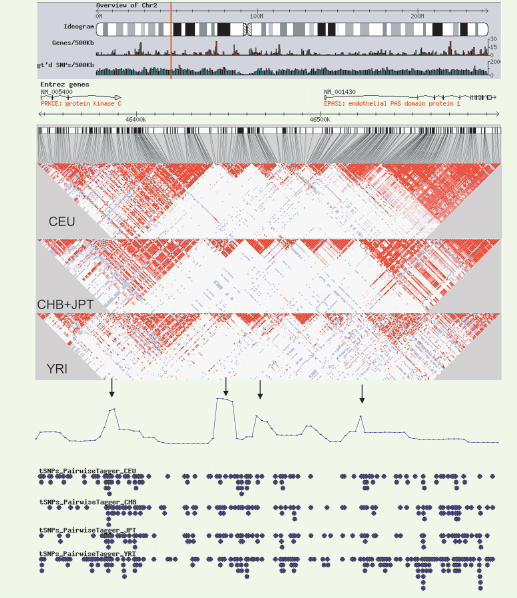
32]. Pour les populations africaines, plusieurs études seront sans doute nécessaires afin d’évaluer la validité des SNP marqueurs étant donné la grande diversité de ces populations.  | Figure 2.
DL observé sur une région du chromosome 2 dans les populations HapMap. Les données du site HapMap ( http://www.hapmap.org) ont été utilisées. Section 1 : Localisation des gènes et des SNP génotypés sur tout le chromosome 2. Section 2 : Localisation des gènes et des SNP génotypés entre les positions 46346800 et 46596799 de l’assemblage NCBI 34 du chromosome 2. Section 3 : Détail du DL entre les SNP de la région pour les populations HapMap (les populations JPT et CHB ont été combinées). Le programme Haploview ( http://www.broad.mit.edu/mpg/haploview/) avec la méthode des intervalles de confiance de Gabriel [ 10] a été utilisé. Différentes teintes de rouge : couleur en fonction du D’ (rouge foncé : D’=1) et aucune preuve de recombinaison (selon les intervalles de confiance). En blanc : D’ <1,0 et preuve de recombinaison. En bleu : D’ = 1,0, mais la présence de recombinaison est non-concluante. Les blocs d’haplotypes sont indiqués en noirs et contiennent au moins 95 % de paires de SNP sans preuve de recombinaison. Section 4 : Taux de recombinaison moyen pour les quatre populations mesuré avec la méthode de McVean [ 16]. Les points chauds de recombinaison sont indiqués avec une flèche. Section 5 : SNP marqueurs sélectionnés selon l’algorithme Tagger pairwise ( http://www.broad.mit.edu/mpg/tagger/) pour les quatre populations. |
|
La Carte d’Haplotype du Génome Humain contribuera sans aucun doute à l’avancement des connaissances sur les maladies complexes et sur l’organisation du génome humain. En plus de ces contributions, cette Carte s’avère extrêmement utile pour découvrir des gènes ayant subi une pression de sélection naturelle ou même pour mesurer indirectement l’effet de variations non-SNP comme les délétions et inversions [12]. Cependant, la Carte ne permettra pas dans un avenir proche de résoudre tous les mystères concernant la génétique des maladies complexes. Il est hautement improbable, par exemple, que grâce à la Carte on puisse révéler des gènes au sein desquels plusieurs variations rares contribuent au phénotype. Si les interactions gènes-gènes concourent potentiellement de façon significative à l’apparition d’un phénotype, des analyses fondées sur ces interactions à l’échelle du génome ne sont pas envisageables, du moins pour l’instant, à cause de la quantité exponentielle du nombre de tests à effectuer et du problème de faux-positifs qui s’y rattache. La réplication d’une étude d’association dans une ou plusieurs autres cohortes permet en principe d’éliminer la plupart des faux positifs, mais des différences dans les protocoles d’études ou au niveau génétique peut aisément confondre les analyses. L’établissement de standards dans les protocoles d’études et dans les méthodes d’analyses est donc essentiel afin d’assurer un succès complet à la Carte d’Haplotype. |
Nous aimerions remercier Albert V. Smith pour avoir fourni généreusement toutes les statistiques du projet HapMap. Nous remercions également Thomas J. Hudson pour son appui et sa confiance, ainsi que toute l’équipe du Centre d’Innovation Génome Québec et Université McGill pour leurs efforts qui ont permis de mener ce projet à terme. Nous aimerions également remercier Génome Canada et Génome Québec pour leur soutien financier qui a rendu possible la participation du Canada et du Québec à ce projet d’envergure internationale.
|
Footnotes |
1. Campion D. Dissection génétique des maladies à hérédité complexe. Med Sci (Paris) 2001; 17 : 1139–48. 2. Lander ES. The new genomics: global views of biology. Science 1996; 274 : 536–9. 3. Lohmueller KE, Pearce CL, Pike M, Lander ES, Hirschhorn JN. Meta-analysis of genetic association studies supports a contribution of common variants to susceptibility to common disease. Nat Genet 2003; 33 : 177–82. 4. Kruglyak L, Nickerson DA. Variation is the spice of life. Nat Genet 2001; 27 : 234–6. 5. Nordborg M, Tavare S. Linkage disequilibrium: what history has to tell us. Trends Genet 2002; 18 : 83–90. 6. Kruglyak L. Prospects for whole-genome linkage disequilibrium mapping of common disease genes. Nat Genet 1999; 22 : 139–44. 7. Daly MJ, Rioux JD, Schaffner SF, et al. High-resolution haplotype structure in the human genome. Nat Genet 2001; 29 : 229–32. 8. Johnson GC, Esposito L, Barratt BJ, et al. Haplotype tagging for the identification of common disease genes. Nat Genet 2001; 29 : 233–7. 9. Patil N, Berno AJ, Hinds DA, et al. Blocks of limited haplotype diversity revealed by high-resolution scanning of human chromosome 21. Science 2001; 294 : 1719–23. 10. Gabriel SB, Schaffner SF, Nguyen H, et al. The structure of haplotype blocks in the human genome. Science 2002; 296 : 2225–9. 11. Integrating ethics and science in the International HapMap Project. Nat Rev Genet 2004; 5 : 467–75. 12. Altshuler D, Brooks LD, Chakravarti A, et al. A haplotype map of the human genome. Nature 2005; 437 : 1299–320. 13. Hinds DA, Stuve LL, Nilsen GB, et al. Whole-genome patterns of common DNA variation in three human populations. Science 2005; 307 : 1072–9. 14. Ke X, Hunt S, Tapper W, et al. The impact of SNP density on fine-scale patterns of linkage disequilibrium. Hum Mol Genet 2004; 13 : 577–88. 15. Tapper W, Collins A, Gibson J, et al. A map of the human genome in linkage disequilibrium units. Proc Natl Acad Sci USA 2005; 102 : 11835–9. 16. McVean GA, Myers SR, Hunt S, et al. The fine-scale structure of recombination rate variation in the human genome. Science 2004; 304 : 581–4. 17. Myers S, Bottolo L, Freeman C, et al. A fine-scale map of recombination rates and hotspots across the human genome. Science 2005; 310 : 321–4. 18. Winckler W, Myers SR, Richter DJ, et al. Comparison of fine-scale recombination rates in humans and chimpanzees. Science 2005; 308 : 107–11 19. Jeffreys AJ, Neumann R, Panayi M, et al. Human recombination hot spots hidden in regions of strong marker association. Nat Genet 2005; 37 : 601–6. 20. Ardlie K, Liu-Cordero SN, Eberle MA, et al. Lower-than-expected linkage disequilibrium between tightly linked markers in humans suggests a role for gene conversion. Am J Hum Genet 2001; 69 : 582–9. 21. Jeffreys AJ, May CA. Intense and highly localized gene conversion activity in human meiotic crossover hot spots. Nat Genet 2004; 36 : 151–6. 22. Wiltshire S, De Bakker PI, Daly MJ. The value of gene-based selection of tag SNPs in genome-wide association studies. Eur J Hum Genet 2006; 14 : 1209–14. 23. Skol AD, Scott LJ, Abecasis GR, Boehnke M. Joint analysis is more efficient than replication-based analysis for two-stage genome-wide association studies. Nat Genet 2006; 38 : 209–13. 24. Hirschhorn JN, Daly MJ. Genome-wide association studies for common diseases and complex traits. Nat Rev Genet 2005; 6 : 95–108. 25. Wang WY, Barratt BJ, Clayton DG, Todd JA. Genome-wide association studies: theoretical and practical concerns. Nat Rev Genet 2005; 6 : 109–18. 26. Klein RJ, Zeiss C, Chew EY, et al. Complement factor H polymorphism in age-related macular degeneration. Science 2005; 308 : 385–9. 27. Mueller JC, Lohmussaar E, Magi R, et al. Linkage disequilibrium patterns and tagSNP transferability among European populations. Am J Hum Genet 2005; 76 : 387–98. 28. Evans DM, Cardon LR. A comparison of linkage disequilibrium patterns and estimated population recombination rates across multiple populations. Am J Hum Genet 2005; 76 : 681–7. 29. Willer CJ, Scott LJ, Bonnycastle LL, et al. Tag SNP selection for Finnish individuals based on the CEPH Utah HapMap database. Genet Epidemiol 2006; 30 : 180–90. 30. Ke X, Durrant C, Morris AP, et al. Efficiency and consistency of haplotype tagging of dense SNP maps in multiple samples. Hum Mol Genet 2004; 13 : 2557–65. 31. Montpetit A, Nelis M, Laflamme P, et al. An evaluation of the performance of Tag SNPs derived from HapMap in a Caucasian population. PLoS Genet 2006; 2 : e27. 32. Zeggini E, Rayner W, Morris AP, et al. An evaluation of HapMap sample size and tagging SNP performance in large-scale empirical and simulated data sets. Nat Genet 2005; 37 : 1320–2. 33. Syvanen AC. Toward genome-wide SNP genotyping. Nat Genet 2005; 37 (suppl) : S5–10. 34. Rioux JD, Daly MJ, Silverberg MS, et al. Genetic variation in the 5q31 cytokine gene cluster confers susceptibility to Crohn disease. Nat Genet 2001; 29 : 223–8. 35. Begovich AB, Carlton VE, Honigberg LA, et al. A missense single-nucleotide polymorphism in a gene encoding a protein tyrosine phosphatase (PTPN22) is associated with rheumatoid arthritis. Am J Hum Genet 2004; 75 : 330–7. 36. Laitinen T, Polvi A, Rydman P, et al. Characterization of a common susceptibility locus for asthma-related traits. Science 2004; 304 : 300–4. 37. Van Eerdewegh P, Little RD, Dupuis J, et al. Association of the ADAM33 gene with asthma and bronchial hyperresponsiveness. Nature 2002; 418 : 426–30. 38. Deeb SS, Fajas L, Nemoto M, et al. A Pro12Ala substitution in PPARgamma2 associated with decreased receptor activity, lower body mass index and improved insulin sensitivity. Nat Genet 1998; 20 : 284–7. 39. Strittmatter WJ, Saunders AM, Schmechel D, et al. Apolipoprotein E: high-avidity binding to beta-amyloid and increased frequency of type 4 allele in late-onset familial Alzheimer disease. Proc Natl Acad Sci USA 1993; 90 : 1977–81. 40. Stefansson H, Sigurdsson E, Steinthorsdottir V, et al. Neuregulin 1 and susceptibility to schizophrenia. Am J Hum Genet 2002; 71 : 877–92. 41. Mira MT, Alcais A, Nguyen VT, et al. Susceptibility to leprosy is associated with PARK2 and PACRG. Nature 2004; 427 : 636–40. 42. Helgadottir A, Manolescu A, Thorleifsson G, et al. The gene encoding 5-lipoxygenase activating protein confers risk of myocardial infarction and stroke. Nat Genet 2004; 36 : 233–9. 43. Paez JG, Janne PA, Lee JC, et al. EGFR mutations in lung cancer: correlation with clinical response to gefitinib therapy. Science 2004; 304 : 1497–500. |



 La Phase I contient un peu plus de un million de SNP polymorphiques dans au moins une population et ayant passé tous les contrôles de qualité (les SNP doivent avoir un taux de succès d’au moins 80 %, être en équilibre Hardy-Weinberg à p > 0,001 et avoir au plus une erreur de transmission mendélienne sur 30 familles ou une erreur de reproductibilité sur 5 réplicats) [
La Phase I contient un peu plus de un million de SNP polymorphiques dans au moins une population et ayant passé tous les contrôles de qualité (les SNP doivent avoir un taux de succès d’au moins 80 %, être en équilibre Hardy-Weinberg à p > 0,001 et avoir au plus une erreur de transmission mendélienne sur 30 familles ou une erreur de reproductibilité sur 5 réplicats) [