Tel le phénix renaissant de ses cendres, Deinococcus radiodurans est capable de ressusciter après avoir été exposée à des doses de rayonnements mortelles. Découverte en 1956 un peu par hasard, pour avoir survécu dans des boîtes de viande en conserve ayant reçu 4 000 grays de rayons γ, cette bactérie a aussitôt suscité un extrême intérêt de la part des chercheurs. Ils ont pu constater qu’elle était capable de résister aussi à la dessiccation, aux UV, et au peroxyde d’hydrogène. Ce microbe non pathogène se rencontre un peu partout sur la planète, non seulement dans des milieux riches en éléments organiques, mais aussi dans des sols arides : désert de Tataouine au sud tunisien [ 1], de Sonoran (Amérique du Nord) [ 2], roches de l’Antarctique où d’autres espèces du genre Deinococcus ont été découvertes [ 3]. Si certaines peuvent se développer au froid, beaucoup sont thermophiles. Du reste, les études phylogénétiques montrent que les Deinococci sont proches du genre Thermus, et il se pourrait qu’elles partagent le même ancêtre commun que les bactéries du genre Thermus comme T. thermophilus.
Son génome fut séquencé par plusieurs équipes étatsuniennes dès que cela fut possible tant la curiosité était grande de connaître l’arsenal dont elle disposait pour réparer son ADN [ 4].
Car, contrairement à ce que pourrait laisser supposer son nom, les irradiations provoquent la même fragmentation de l’ADN chez cette bactérie radiodurans que chez les autres. Mais, malgré ses chromosomes pulvérisés et son ADN réduit en segments de 20 à 30 kb, Deinococcus radiodurans est capable de se régénérer.
Pourtant, le séquençage de son génome n’a pas vraiment fourni la clef de l’énigme. Il est constitué de deux chromosomes circulaires (de 2 648.638 pb et 412 348 pb), d’un mégaplasmide (1 777 466 pb) et d’un petit plasmide (45 704pb). Certes, il contient des séquences répétées réparties sur 13 sites, et des gènes codant de nombreuses enzymes appartenant aux grandes voies de reconnaissance et de réparation des lésions de l’ADN. Mais bien d’autres espèces en sont munies sans détenir ce pouvoir de reconstituer en quelques heures leur patrimoine génétique : E. coli ne peut survivre à une fragmentation de son ADN en segments de 500 kb. Certes, il existe chez cette bactérie une polyploïdie importante (avec des cellules contenant de 4 à 10 génomes) qui peut contribuer à cette capacité de régénération, mais qui n’est pas suffisante pour l’expliquer. Il fallait donc analyser point par point les différentes étapes de réparation s’élaborant après irradiation.
C’est une équipe de chercheurs de l’Inserm (U571) à la Faculté de Médecine de Necker-Enfants Malades (Paris, France), dirigée par Miroslav Radman, en collaboration avec deux autres laboratoires français et une équipe croate, qui vient enfin d’élucider en détail ce mécanisme de restitutio ad integrum unique en biologie [ 5].
Les techniques utilisées ont comporté, entre autres, l’électrophorèse en champ pulsé pour mesurer la taille des fragments au fur et à mesure de leur fusion, l’adjonction de thymidine tritiée et de BrdU pour repérer les brins synthétisés par densité et par photolyse au niveau moléculaire, et l’utilisation de la microscopie en fluorescence pour les observer à l’échelon cellulaire. Photolyse et immunofluorescence d’ADN monocatenaire néosynthétisé sont les méthodes mises au point par l’équipe.
Le système de réparation comporte deux étapes.
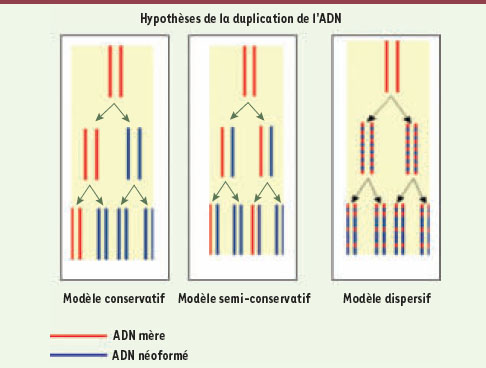
- En premier lieu, au niveau des points de cassure de l’ADN (double brin), les exonucléases produisent des segments simple-brin. À partir de ceux-ci, un brin complémentaire est synthétisé. Mais à la différence de la synthèse normale, semi-conservative, la synthèse de nouveaux brins se fait à partir des bouts libres des nombreux fragments (amorces), en utilisant l’ADN polymérase I. Celle-ci utilise comme matrice l’ADN complémentaire d’un fragment chevauchant appartenant à une autre copie génomique dans la même cellule. Puis, ces « bouts collants » néosynthétisés se réassocient (single strand annealing) reliant ainsi les fragments contigus (Figure 1). Outre la voie SSA (single strand annealing ou appariement des ADN simple-brin, D radiodurans utilise un autre mécanisme jusqu’alors inconnu : la voie de synthèse étendue par réassociation des brins (extended synthesis-dependant strand annealing ou ESDSA). Il en résulte une mosaïque de segments anciens fusionnées via les segments nouveaux, comme dans l’hypothèse du modèle « dispersif » de réplication exclu par Meselson et Stahl [ 6] (Figure 2).
- Une fois les longs fragments linéaires reconstitués à partir des petits fragments, ils se rassemblent et - par chevauchement et par un mécanisme de recombinaison homologue (grâce la protéine RecA) - ils opèrent des crossing over pour reformer le chromosome circulaire (Figure 3).
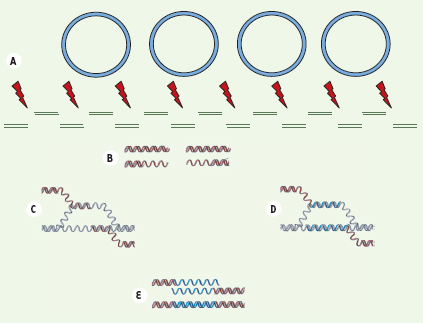
| Figure 1.
Mécanisme de réparation de la bactérie Deinococcus radiodurans . A. Après irradiation ou dessiccation, les chromosomes sont pulvérisés en petits segments double brin. B. Les exonucléases digèrent partiellement un des brins d’ADN fragmenté pour exposer le brin complémentaire.C. Ces simples brins s’associent à un ADN complémentaire provenant d’un fragment chevauchant lui-même issu d’une autre copie chromosomique présente dans cette bactérie procaryote polyploïde. D.
À partir de cet ADN qui sert d’amorce, un nouveau brin est synthétisé sur une matrice contiguë dans le chromosome. Sont utilisées la voie SSA (single strand annealing ou appariement des ADN simple brin, mais aussi une voie de synthèse étendue par réassociation des brins (extended synthesis-dependant strand annealing ou ESDSA). E. De longs brins sont ainsi synthétisés, les brins en excès étant excisés par les endonucléases (d’après [
7]). |
| Figure 2.
Schéma classique des trois hypothèses de duplication de l’ADN proposées dans l’article de Meselson et Stahl [6]. |
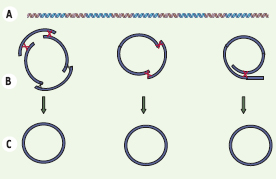
| Figure 3.
Les longs fragments chevauchants (A) se superposent et (B) opèrent des recombinaisons homologues (dépendantes de RecA) pour reconstituer les chromosomes circulaires (C) (d’après [5]). |
Il subsiste encore bien des interrogations. Comment D. radiodurans assure-t-elle sans se tromper la reconstruction de ses chromosomes ? Certaines étapes sont périlleuses : l’hybridation d’un simple brin à un fragment intact ; l’hybridation des brins nouvellement synthétisés…
Dès à présent on se prend à rêver. De telles bactéries pourraient-elles épurer les centres de déchets, nucléaires et autres ? Pourraient-elles ensemencer la vie sur des planètes stériles ? Ou encore, pourraient-elles aider à la thérapie des maladies dues aux troubles de réparation de l’ADN et jeter les bases d’une nouvelle médecine régénérative ?
En attendant, on peut dire que D. radiodurans a pleinement fait siens les premiers vers du fameux poème de Rudyard Kipling :
… Si tu peux voir détruit l’ouvrage de ta vie
Et sans dire un seul mot te mettre à rebâtir…