| |
| Med Sci (Paris). 2006 November; 22(11): 941–946. Published online 2006 November 15. doi: 10.1051/medsci/20062211941.H2A.Z : un variant d’histone qui orne les promoteurs des gènes Benoît Guillemette and Luc Gaudreau* Département de Biologie, Faculté des sciences, Université de Sherbrooke, 2500, boulevard de l’Université, Sherbrooke (Québec) J1K 2R1, Canada |
Chaque cellule humaine possède un total d’environ deux mètres d’ADN, condensé dans le noyau en une structure appelée chromatine. L’unité de base de la chromatine, le nucléosome, a été identifiée il y a environ 30 ans [
1,
2], mais ce n’est que depuis une dizaine d’années seulement que le domaine de l’étude de la chromatine a particulièrement attiré l’attention de la communauté scientifique. Le nucléosome est composé d’un octamère d’histones autour duquel l’ADN effectue 1,7 tour [
3]. Il existe quatre histones différentes présentes chacune en deux exemplaires au cœur du nucléosome : H3 et H4 forment un tétramère, H2A et H2B forment deux dimères. Ces histones s’assemblent grâce à un domaine protéique nommé histone fold. Cet assemblage entraîne la formation d’une surface d’interaction avec le phosphosquelette de l’ADN. La compaction ainsi créée rend l’accès à l’ADN difficile pour les énormes complexes protéiques en jeu dans le métabolisme de l’ADN. |
Modifications de la chromatine Au cours de processus cellulaires comme la différenciation, la cellule doit programmer finement l’expression de ses gènes. La modulation de la structure de la chromatine d’un gène est un mécanisme primordial par lequel la cellule régule son expression. On connaît au moins trois mécanismes permettant aux cellules de modifier la structure de la chromatine. Le premier passe par l’action de complexes enzymatiques qui utilisent l’énergie libérée par l’hydrolyse de l’ATP pour déstabiliser la structure nucléosomale et rendre l’ADN plus accessible. Le deuxième repose sur la modification post-traductionnelle des extrémités amino et carboxyterminales des histones qui se projettent à l’extérieur de la structure du nucléosome. Les modifications les plus étudiées sont la méthylation, l’acétylation, la phosphorylation et l’ubiquitinylation. Certaines d’entre elles peuvent moduler la charge des résidus et permettraient de déstabiliser l’interaction des extrémités des histones avec le phosphosquelette de l’ADN. D’un autre côté, certaines modifications pourraient constituer un code qui serait lu par des protéines spécifiques [
4,
5]. Par exemple, la méthylation de la lysine 9 de H3 permet la liaison de HP1 (heterochromatin protein 1), une protéine de l’hétérochromatine, structure compacte et transcriptionnellement silencieuse dans le noyau. Le troisième mécanisme par lequel la structure de la chromatine peut être modifiée est l’incorporation de variants d’histones. Ceux-ci remplacent les histones principales dans le nucléosome et lui confèrent des propriétés particulières. Des variants ont été identifiés pour H2A, H2B et H3 et jouent des rôles multiples [
6]. Tandis que les histones principales sont majoritairement exprimées lors de la phase S du cycle cellulaire, en même temps que la synthèse de l’ADN, la plupart des variants sont exprimés indépendamment du cycle cellulaire [
7]. Récemment, des complexes protéiques capables d’introduire certains variants dans la chromatine ont été répertoriés. Ceux-ci permettent d’intégrer les variants d’histones dans la chromatine à des endroits précis où ils peuvent jouer différents rôles. |
Le variant d’histone H2A.Z H2A.Z est un variant de l’histone H2A. Les deux protéines possèdent environ 60 % d’homologie entre elles dans leur séquence en acides aminés, les différences se trouvant principalement localisées aux extrémités amino et carboxyterminales [
8]. Son importance a été démontrée à l’examen de plusieurs processus biologiques, en particulier la transcription des gènes [
9–
12], la ségrégation des chromosomes [
13,
14], la condensation de l’hétérochromatine [
15,
16] et la réplication de l’ADN [
17]. Ce variant est essentiel à la survie de nombreux organismes, notamment la drosophile [
18], le xénope [
19] et la souris [
20]. Il a été hautement conservé au cours de l’évolution [
21], encore plus que l’histone principale H2A, ce qui suggère que ce variant joue un rôle important dans le fonctionnement de la cellule. Deux études récentes ont d’ailleurs démontré que H2A.Z est fortement exprimé dans des types de cancers non différenciés et métastatiques [
22,
23]. |
Rôle positif et global en transcription génique Plusieurs indices laissent croire que H2A.Z régule positivement la transcription des gènes auxquels il est associé. Allis et al., en 1980, ont découvert que H2A.Z se trouve exclusivement dans le macronoyau transcriptionnellement actif du protozoaire Tetrahymena thermophila [
24], suggérant déjà à l’époque un rôle positif global pour ce variant d’histone. Toutefois, une analyse génomique de l’effet de la délétion de HTZ1 (le gène codant pour H2A.Z) sur la transcription des gènes de la levure Saccharomyces cerevisiae a récemment révélé que seulement 214 gènes sont négativement affectés par cette délétion (tandis que 109 sont affectés positivement) [12]. De manière surprenante, environ la moitié de ces gènes sont situés à moins de 35 000 pb des télomères et se trouvent en petits groupes que Meneghini et al. ont appelés les HZAD (H2A.Z/Htz1 activated domains). Sir2 (silent information regulator 2) est une composante principale de l’hétérochromatine des télomères (avec Sir3 et 4) de la levure qui est délocalisée dans la souche délétée pour HTZ1. Ces résultats suggèrent que H2A.Z agit comme un agent qui limite l’étalement de l’hétérochromatine aux frontières télomériques et prévient ainsi l’inhibition de la transcription (« silençage ») des gènes situés à proximité. Il est à noter que la liaison de H2A.Z est plus étendue dans ces régions et demeure distincte du patron ponctuel dans le reste du génome [
25] (voir ci-dessous). L’analyse de Meneghini et al. [12] a toutefois été effectuée en milieu riche et en croissance exponentielle, ce qui pourrait mener à sous-estimer l’importance de H2A.Z pour l’induction de plusieurs autres gènes selon les circonstances. En effet, la délétion de HTZ1 cause des défauts dans la transcription de plusieurs gènes inductibles comme GAL1, PHO5 et PUR5 [9–
11]. De plus, lors d’une analyse globale de la liaison de H2A.Z dans la levure, notre équipe, à l’instar d’autres groupes de recherche, a trouvé que H2A.Z est présent dans une grande proportion (63 %) des promoteurs des gènes dans de la levure ; ces gènes semblent disséminés de façon stochastique dans le génome et sont transcriptionellement inactifs [25–
28], ce qui conforte l’hypothèse d’un rôle global de H2A.Z dans la transcription génique. H2A.Z est également localisé dans les régions promotrices de plusieurs gènes inductibles chez les eucaryotes supérieurs [
29,
30] (N. Gévry et L.Gaudreau, données non publiées), ce qui suggère que son rôle global de transcription aurait été conservé au cours de l’évolution. |
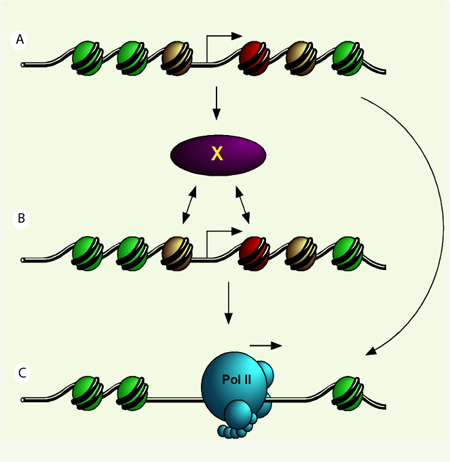
H2A.Z contribue à la structure des promoteurs La présence de H2A.Z au sein d’une grande proportion de promoteurs géniques de la levure et son importance dans le développement de plusieurs espèces suggèrent que ce variant d’histone pourrait jouer un rôle primordial dans la transcription de plusieurs gènes. La compréhension de son rôle au sein du nucléosome pourrait donc être essentielle pour comprendre les rouages de l’expression des gènes. La liaison de H2A.Z aux promoteurs des gènes dans la levure est généralement très ponctuelle ; elle s’effectue en moyenne soit dans un, soit dans deux nucléosomes situés à proximité du site d’initiation de la transcription [25, 28] (Figure 1a). La liaison de H2A.Z aux promoteurs de gènes inactifs suggère que ce variant d’histone pourrait préparer l’activation des gènes en causant des changements dans la structure de la chromatine des promoteurs. En effet, les nucléosomes des promoteurs qui contiennent H2A.Z sont mieux organisés et possèdent une région libre de nucléosome mieux définie que les promoteurs sans H2A.Z [25] (Figure 1A). De plus, la délétion de HTZ1 entraîne le déplacement d’un nucléosome dans le promoteur du gène GAL1 [25]. H2A.Z pourrait donc faciliter le positionnement de nucléosomes dans les promoteurs de gènes inactifs afin de rendre cette région de chromatine propice au recrutement de la machinerie transcriptionnelle (Figure 1C).  | Figure 1.
Rôle du variant d’histone H2A.Z dans l’expression des gènes. A. Schéma récapitulatif de la structure des promoteurs de gènes inactifs dans la levure S. cerevisiae qui contiennent H2A.Z (63 % des gènes). Les nucléosomes sont schématisés par des cercles autour desquels s’enroule l’ADN (noir). Le nucléosome (rouge) contient H2A.Z dans la plupart des promoteurs et est situé immédiatement en aval d’une région libre de nucléosome dans le promoteur. Les nucléosomes (jaune) de part et d’autre du nucléosome (rouge) peuvent aussi contenir H2A.Z dans quelques promoteurs. Les nucléosomes (vert) contiennent généralement l’histone H2A. Il est à noter, qu’en théorie, un nucléosome ne contient que deux molécules H2A.Z ou H2A [
32]. Le site d’initiation de la transcription est illustré par une flèche ; elle est située ici dans la région libre de nucléosome, mais elle peut varier d’emplacement selon les gènes. B et C. Deux mécanismes (non exclusifs) par lesquels H2A.Z pourrait contribuer à l’activation des gènes. B. H2A.Z pourrait interagir directement avec un facteur (X) encore inconnu qui modifie l’architecture du promoteur et prépare le gène à l’activation de la transcription. C. Lors de l’activation du gène, les nucléosomes contenant H2A.Z sont rapidement enlevés pour laisser place à l’ARN polymérase II (Pol II) et ses facteurs associés. |
En ce qui concerne leurs propriétés physiques, les nucléosomes reconstitués in vitro contenant H2A.Z sont différents de ceux contenant H2A (pour revue, voir [
31]). De plus, les nucléosomes reconstitués in vitro contenant H2A.Z sont réfractaires à certaines modifications post-traductionnelles [
26]. Ces résultats suggèrent que H2A.Z possèderait la capacité intrinsèque de modifier la structure de la chromatine. Il se pourrait également que H2A.Z interagisse avec une ou des protéines qui pourraient influencer la structure de la chromatine (Figure 1B). La région carboxyterminale de H2A.Z possède une région riche en acides aminés acidiques1,. Au sein de la structure nucléosomique, cette région acide rejoint H2B, créant ainsi une surface d’interaction potentielle [32]. Cette surface acide étendue est absente dans le nucléosome contenant l’histone principale H2A. Au moyen d’un second mécanisme (il n’exclut pas le premier), H2A.Z pourrait contribuer à l’activation des gènes en facilitant l’éjection des nucléosomes dans lequel il est contenu (Figure 1C). En effet, l’activation de gènes entraîne la perte de contact avec l’ADN des nucléosomes qui couvrent le promoteur. De plus, il a été démontré que H2A.Z est déplacé, lors de l’activation de plusieurs gènes dans la levure [9–11,
27], chez le poulet [30] et chez l’homme [29]. On ne sait pas dans ces cas si H2A.Z seul est déplacé, s’il y a perte de dimères H2A.Z/H2B ou bien perte de tout le nucléosome qui le contient. Le mécanisme par lequel H2A.Z est déplacé et les facteurs qui y contribuent sont également inconnus. |
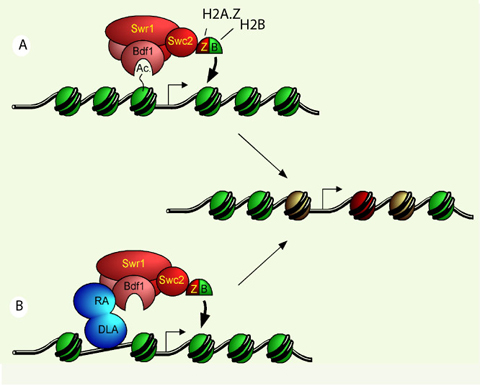
Chargement de H2A.Z par le complexe SWR1 Comment la cellule peut-elle diriger la localisation de H2A.Z aux sites des promoteurs des deux tiers des gènes à l’état inactif ? Dans le cas de la levure, le complexe protéique SWR1 (SWI/SNF-related 1) a été identifié comme un complexe de remodelage de la chromatine apte à charger un dimère H2A.Z/H2B à la place d’un dimère H2A/H2B dans le nucléosome [
33–
35]. La sous-unité catalytique de ce complexe, Swr1, possède un domaine ATPase homologue à Swi2/Snf2 ; elle hydrolyse l’ATP pour permettre l’échange de H2A.Z [35]. Bdf1 (bromo-domain factor 1) est une protéine qui fait partie du complexe SWR1 [33–35] et qui possède deux bromodomaines. Ces domaines protéiques peuvent lier les lysines acétylées sur les extrémités aminoterminales des histones. De cette manière, Bdf1 pourrait « lire » un patron d’acétylation propre aux nucléosomes au sein des promoteurs des gènes inactifs et permettre le chargement de H2A.Z spécifiquement dans ces régions (Figure 2a). BDF1 est partiellement requis pour le chargement de H2A.Z, mais la délétion double de BDF1 et de BDF2, ce dernier codant pour un homologue de Bdf1, réduit substantiellement la présence de H2A.Z dans la chromatine [27, 28]. 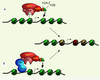 | Figure 2.
Deux mécanismes par lesquels le complexe SWR1 pourrait être recruté dans les promoteurs de gènes inactifs et incorporer H2A.Z. Le complexe SWR1 est composé d’une douzaine de protéines dont Swr1, la sous-unité catalytique, Swc2, qui interagit directement avec un dimère H2A.Z-H2B [
39] et Bdf1, une protéine à deux bromodomaines. A. SWR1 pourrait être recruté via l’interaction des bromodomaines de Bdf1 avec les extrémités des histones acétylées sur les lysines. B. SWR1 pourrait être recruté par suite de l’interaction avec la région d’activation (RA) de facteurs de transcription qui peuvent lier l’ADN par leur domaine de liaison à l’ADN (DLA), à la condition que le facteur de transcription puisse lier l’ADN pendant que le gène est inactif. |
Il a été aussi suggéré que le complexe SWR1 serait la cible de facteurs de transcription qui pourraient le recruter aux promoteurs (Figure 2B). En appui à cette hypothèse, Raisner et al. ont utilisé comme exemple le promoteur du gène SNT1 dans la levure [28]. Ils ont démontré qu’une séquence consensus pour la protéine Reb1 pouvait mener à la localisation de H2A.Z et à la formation d’une région libre de nucléosome dans ce promoteur. En combinaison avec une série de dA : dT, cette séquence peut forcer l’insertion de H2A.Z dans une région codante (dans laquelle il n’y a normalement pas de H2A.Z) et causer la formation d’une région libre de nucléosome [28]. L’idée que le complexe SWR1 peut être ciblé par un (ou plusieurs) facteur(s) de transcription implique que ce facteur doit lier le promoteur en état répressif. D’ailleurs, H2A.Z est enrichi dans certains sous-groupes de gènes corégulés de la levure [27], ce qui soutient l’idée que le complexe SWR1 soit ciblé par des facteurs de transcription. |
Assemblage de l’hétérochromatine En contraste avec les résultats énumérés plus haut où il régule positivement la transcription, H2A.Z serait aussi important pour le « silençage » transcriptionnel. En effet, H2A.Z est présent dans l’hétérochromatine péricentromérique (correspondant à l’hétérochromatine qui flanque les centromères) durant les étapes initiales du développement de la souris. De plus, il colocalise avec HP1α et INCENP, deux facteurs hétérochromatiques importants pour la ségrégation des chromosomes ; il entre également en interaction avec ces facteurs [15]. La déplétion de H2A.Z par interférence par l’ARN cause des défauts dans la ségrégation des chromosomes et entraîne une relocalisation de HP1α [13]. L’homologue de H2A.Z chez la drosophile, H2Av, est également essentiel au recrutement de HP1 lors de la formation de l’hétérochromatine péricentrique [16]. Enfin, la délétion de H2A.Z observée dans la levure entraîne la perte du « silençage » d’un gène intégré aux télomères [
36]. Il semble donc que H2A.Z ait acquis, au cours de l’évolution, une fonction dans l’assemblage de l’hétérochromatine. |
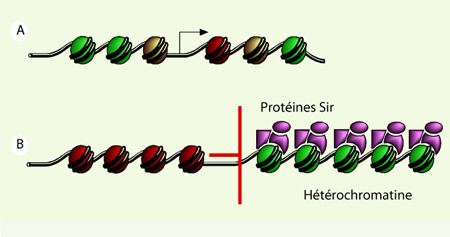
Mécanisme dépendant du patron de liaison La synthèse des résultats présentés précédemment suggère que H2A.Z joue plusieurs rôles dans différents processus cellulaires. Nous proposons que H2A.Z pourrait affecter différemment la structure de la chromatine selon la manière dont il est localisé : ponctuellement (dans un ou deux nucléosomes) ou de manière étendue (dans plusieurs nucléosomes adjacents). Le patron de liaison de H2A.Z avec les promoteurs des gènes est ponctuel mais diverge près des télomères où les régions contenant H2A.Z sont plus larges [25]. Cela suggère que dans ces régions, plusieurs nucléosomes adjacents contiennent H2A.Z (Figure 3B). Cette localisation différente s’accorde bien avec la position des gènes qui sont affectés négativement par le « silençage » hétérochromatique dans les cellules htz1Δ, soit jusqu’à 35 000 pb des télomères [12]. Il semble donc que la présence de plusieurs nucléosomes H2A.Z adjacents pourrait contribuer à former une « barrière » contre l’étendue de l’hétérochromatine dans la levure (Figure 3B). Cette propriété de H2A.Z pourrait également contribuer à l’élaboration de la structure de l’hétérochromatine chez les eucaryotes supérieurs. En effet, in vitro, une matrice d’ADN pouvant accueillir 12 nucléosomes contenant H2A.Z se replie plus facilement que la même matrice contenant H2A [
37,
38].  | Figure 3.
H2A.Z pourrait posséder différents mécanismes d’action selon la manière dont il est localisé. A. Dans les promoteurs de gènes disséminés dans le génome, H2A.Z est localisé ponctuellement dans un ou deux nucléosomes (rouge et jaune) à proximité du site de démarrage de la transcription (flèche). B. Dans les régions à moins de 35 000 pb des télomères, H2A.Z pourrait être présent dans plusieurs nucléosomes adjacents (rouge). Cette propriété pourrait contribuer au rôle anti- silençage de H2A.Z proposé par Meneghini et al. [ 12] et empêcher l’étalement des protéines Sir, constituants de l’hétérochromatine télomérique dans la levure. |
|
Plusieurs études récentes ont révélé d’importantes caractéristiques du variant d’histone H2A.Z, lui accordant un rôle central dans la modulation de l’expression des gènes. Les découvertes concernant ce variant pourront contribuer à comprendre les mécanismes par lesquels les gènes sont sélectivement exprimés lors des processus cellulaires physiologiques comme la différenciation et dérégulés lors de processus pathologiques comme les cancers. La localisation génomique globale de H2A.Z a révélé plusieurs aspects inattendus de la biologie de ce variant d’histone. L’élucidation des mécanismes par lesquels H2A.Z peut moduler la structure de la chromatine devient dès lors encore plus stimulante. |
Nous remercions Joëlle Brodeur pour la relecture du manuscrit ainsi que Benoit Leblanc pour la conception des figures. Les travaux provenant de notre laboratoire ont été financièrement soutenus par le Conseil de recherche en sciences naturelles et génie du Canada (CRSNG) et par les Instituts de recherche en santé du Canada (IRSC). Luc Gaudreau est titulaire de la chaire de recherche du Canada sur les mécanismes de transcription génique.
|
Footnotes |
1. Kornberg RD. Chromatin structure : a repeating unit of histones and DNA. Science 1974; 184 : 868–71. 2. Oudet P, Gross-Bellard M, Chambon P. Electron microscopic and biochemical evidence that chromatin structure is a repeating unit. Cell 1975; 4 : 281–300. 3. Luger K, Mader AW, Richmond RK, et al. Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature 1997; 389 : 251–60. 4. Strahl BD, Allis CD. The language of covalent histone modifications. Nature 2000; 403 : 41–5. 5. Turner BM. Cellular memory and the histone code. Cell 2002; 111 : 285–91. 6. Henikoff S, Furuyama T, Ahmad K. Histone variants, nucleosome assembly and epigenetic inheritance. Trends Genet 2004; 20 : 320–6. 7. Henikoff S, Ahmad K. Assembly of variant histones into chromatin. Annu Rev Cell Dev Biol 2005; 21 : 33–53. 8. Jin J, Cai Y, Li B,et al. In and out : histone variant exchange in chromatin. Trends Biochem Sci 2005; 30 : 680–7. 9. Adam M, Robert F, Larochelle M, Gaudreau L. H2A.Z is required for global chromatin integrity and for recruitment of RNA polymerase II under specific conditions. Mol Cell Biol 2001; 21 : 6270–9. 10. Larochelle M, Gaudreau L. H2A.Z has a function reminiscent of an activator required for preferential binding to intergenic DNA. EMBO J 2003; 22 : 4512–22. 11. Santisteban MS, Kalashnikova T, Smith MM. Histone H2A.Z regulates transcription and is partially redundant with nucleosome remodeling complexes. Cell 2000; 103 : 411–22. 12. Meneghini MD, Wu M, Madhani HD. Conserved histone variant H2A.Z protects euchromatin from the ectopic spread of silent heterochromatin. Cell 2003; 112 : 725–36. 13. Rangasamy D, Greaves I, Tremethick DJ. RNA interference demonstrates a novel role for H2A.Z in chromosome segregation. Nat Struct Mol Biol 2004; 11 : 650–5. 14. Krogan NJ, Baetz K, Keogh MC,et al. Regulation of chromosome stability by the histone H2A variant Htz1, the Swr1 chromatin remodeling complex, and the histone acetyltransferase NuA4. Proc Natl Acad Sci USA 2004; 101 : 13513–8. 15. Rangasamy D, Berven L, Ridgway P, Tremethick DJ. Pericentric heterochromatin becomes enriched with H2A.Z during early mammalian development. EMBO J 2003; 22 : 1599–607. 16. Swaminathan J, Baxter EM, Corces VG. The role of histone H2Av variant replacement and histone H4 acetylation in the establishment of Drosophila heterochromatin. Genes Dev 2005; 19 : 65–76. 17. Dhillon N, Oki M, Szyjka SJ, et al. Functions to regulate progression through the cell cycle. Mol Cell Biol 2006; 26 : 489–501. 18. Van Daal A, Elgin SC. A histone variant, H2AvD, is essential in Drosophila melanogaster. Mol Biol Cell 1992; 3 : 593–602. 19. Ridgway P, Brown KD, Rangasamy D, et al. Unique residues on the H2A.Z containing nucleosome surface are important for Xenopus laevis development. J Biol Chem 2004; 279 : 43815–20. 20. Faast R, Thonglairoam V, Schulz TC,et al. Histone variant H2A.Z is required for early mammalian development. Curr Biol 2001; 11 : 1183–7. 21. Thatcher TH, Gorovsky MA. Phylogenetic analysis of the core histones H2A, H2B, H3, and H4. Nucleic Acids Res 1994; 22 : 174–9. 22. Zucchi I, Mento E, Kuznetsov VA,et al. Gene expression profiles of epithelial cells microscopically isolated from a breast-invasive ductal carcinoma and a nodal metastasis. Proc Natl Acad Sci USA 2004; 101 : 18147–52. 23. Rhodes DR, Yu J, Shanker K,et al. Large-scale meta-analysis of cancer microarray data identifies common transcriptional profiles of neoplastic transformation and progression. Proc Natl Acad Sci USA 2004; 101 : 9309–14. 24. Allis CD, Glover CV, Bowen JK, Gorovsky MA. Histone variants specific to the transcriptionally active, amitotically dividing macronucleus of the unicellular eucaryote, Tetrahymena thermophila. Cell 1980; 20 : 609–17. 25. Guillemette B, Bataille AR, Gevry N,et al. Variant histone H2A.Z is globally localized to the promoters of inactive yeast genes and regulates nucleosome positioning. PLoS Biol 2005; 3 : e384. 26. Li B, Pattenden SG, Lee D,et al. Preferential occupancy of histone variant H2AZ at inactive promoters influences local histone modifications and chromatin remodeling. Proc Natl Acad Sci USA 2005; 102 : 18385–90. 27. Zhang H, Roberts DN, Cairns BR. Genome-wide dynamics of Htz1, a histone H2A variant that poises repressed/basal promoters for activation through histone loss. Cell 2005; 123 : 219–31. 28. Raisner RM, Hartley PD, Meneghini MD,et al. Histone variant H2A.Z marks the 5’ ends of both active and inactive genes in euchromatin. Cell 2005; 123 : 233–48. 29. Farris SD, Rubio ED, Moon JJ, et al. Transcription-induced chromatin remodeling at the c-myc gene involves the local exchange of histone H2A.Z. J Biol Chem 2005; 280 : 25298–303. 30. Bruce K, Myers FA, Mantouvalou E,et al. The replacement histone H2A.Z in a hyperacetylated form is a feature of active genes in the chicken. Nucleic Acids Res 2005; 33 : 5633–9. 31. Dryhurst D, Thambirajah AA, Ausio J. New twists on H2A.Z: a histone variant with a controversial structural and functional past. Biochem Cell Biol 2004; 82 : 490–7. 32. Suto RK, Clarkson MJ, Tremethick DJ, Luger K. Crystal structure of a nucleosome core particle containing the variant histone H2A.Z. Nat Struct Biol 2000; 7 : 1121–4. 33. Krogan NJ, Keogh MC, Datta N,et al. A Snf2 family ATPase complex required for recruitment of the histone H2A variant Htz1. Mol Cell 2003; 12 : 1565–76. 34. Kobor MS, Venkatasubrahmanyam S, Meneghini MD,et al. A protein complex containing the conserved Swi2/Snf2-related ATPase Swr1p deposits histone variant H2A.Z into euchromatin. PLoS Biol 2004; 2 : E131. 35. Mizuguchi G, Shen X, Landry J, et al. ATP-driven exchange of histone H2AZ variant catalyzed by SWR1 chromatin remodeling complex. Science 2004; 303 : 343–8. 36. Dhillon N, Kamakaka RT. A histone variant, Htz1p, and a Sir1p-like protein, Esc2p, mediate silencing at HMR. Mol Cell 2000; 6 : 769–80. 37. Fan JY, Gordon F, Luger K, et al. The essential histone variant H2A.Z regulates the equilibrium between different chromatin conformational states. Nat Struct Biol 2002; 9 : 172–6. 38. Fan JY, Rangasamy D, Luger K, Tremethick DJ. H2A.Z alters the nucleosome surface to promote HP1alpha-mediated chromatin fiber folding. Mol Cell 2004; 16 : 655–61. 39. Wu WH, Alami S, Luk E,et al. Swc2 is a widely conserved H2AZ-binding module essential for ATP-dependent histone exchange. Nat Struct Mol Biol 2005; 12 : 1064–71. |