Toutes les cellules eucaryotes contiennent des générateurs d’énergie extrêmement efficaces : les mitochondries. Elles proviennent d’un unique événement d’endosymbiose [ 1]. Et pourtant, même si leurs rôles biologiques sont conservés, elles ont évolué de manière très divergente entre les plantes et les animaux. Cette divergence est particulièrement remarquable dans l’organisation du génome et dans la régulation de son expression.
La différence de taille est frappante : alors que celle des génomes mitochondriaux des vertébrés va de 15 à 20 kb et que celle du génome de la levure S. cerevisiae est de 76 kb, la taille des génomes mitochondriaux des plantes varie de 180 kb à 2 400 kb. Cette augmentation de taille n’est pas corrélée à un accroissement de la capacité codante, mais s’explique par la présence de larges régions intergéniques, absentes chez les animaux [ 2].
En effet, le génome mitochondrial humain est une molécule circulaire double brin, très compacte, avec une seule petite région non codante et sans introns. Au contraire, chez les plantes, le « cercle maître » est plutôt un modèle théorique. De longues séquences répétées permettent des recombinaisons donnant lieu à de nombreuses molécules sub-génomiques circulaires, branchées, ou linéaires. Les gènes sont éparpillés sur ces molécules, avec de grandes régions intergéniques. De plus, les génomes mitochondriaux de plante contiennent des insertions d’ADN étranger viral, nucléaire, chloroplastique ou d’origine inconnue.
En conséquence de cette diversité dans l’organisation des génomes, les mécanismes contrôlant l’expression du génome diffèrent remarquablement. Chez l’homme, la transcription est très simple : chacun des deux brins d’ADN est transcrit à partir d’une seule région promotrice, donnant lieu à deux transcrits polycistroniques, qui sont ensuite clivés de part et d’autre des ARN de transfert pour libérer les ARN ribosomiques et messagers. Chez la plante, l’analyse des unités transcriptionnelles est complexe du fait de l’existence de promoteurs multiples ou alternatifs, de nombreux sites de maturation des transcrits précurseurs, de la présence d’introns, de cas de trans-épissage et de l’absence de sites connus de terminaison de la transcription [ 3]. L’image que l’on peut avoir de la transcription mitochondriale chez la plante est celle d’un processus pour le moins permissif, sans réel contrôle ou régulation. Ce n’est qu’ensuite, par des événements post-transcriptionnels tels que maturation et contrôle de la stabilité des ARN, que s’opère la régulation de l’expression des gènes.
Le contrôle de l’abondance de chaque ARN est essentiel pour le fonctionnement correct de la cellule. Il est le résultat de l’équilibre entre la synthèse et la dégradation des ARN. La polyadénylation (ajout d’une série d’adénosines à l’extrémité 3’ d’un ARN) joue ici un rôle crucial. Il est bien connu que les ARN messagers dans le cytoplasme sont protégés et donc stabilisés par une longue queue polyA. Cependant, cela est loin d’être une généralité. Dans la mitochondrie de plante, comme dans la bactérie et dans le chloroplaste, la polyadénylation est un signal qui dirige l’ARN vers la dégradation [ 4, 5].
La polynucléotide phosphorylase (PNPase) est une 3’-5’ exoribonucléase qui dégrade les ARN polyadénylés. La PNPase mitochondriale d’Arabidopsis thaliana a été caractérisée récemment dans notre laboratoire [ 6, 7]. Sa sous-expression conduit à l’accumulation de grands précurseurs d’ARN messagers et ribosomiques, qui n’ont pas été maturés. Ces premières études ont révélé que la PNPase était essentielle pour la maturation en 3’ et la dégradation de certains ARN de la mitochondrie [6, 7].
Afin de mieux définir les rôles biologiques de la PNPase, nous avons entrepris une recherche exhaustive des substrats de la PNPase. Près de 300 ARN polyadénylés ont été clonés, s’accumulant dans les plantes qui sous-expriment la PNPase. Ces ARN sont considérés comme des marqueurs de dégradation puisqu’ils permettent l’identification de transcrits nécessitant la PNPase pour leur dégradation. Ainsi, plusieurs sortes de substrats se dégagent de cette étude. Tout d’abord les sous-produits de la maturation des ARN ribosomiques et de transfert (les séquences transcrites qui ne sont plus dans l’ARN mature). Ensuite, de façon surprenante, des ARN provenant de régions qui, bien qu’étant exemptes de gènes connus, sont parfois très fortement transcrites. Certains de ces transcrits présentent un danger potentiel pour les fonctions de la mitochondrie. C’est le cas de ceux qui contiennent des cadres de lectures chimériques créés par recombinaison (dont certains peuvent mener à un phénotype connu de stérilité mâle cytoplasmique), ou des ARN antisens transcrits à partir du brin opposé à celui d’un gène connu. Des transcrits aberrants et non fonctionnels sont donc produits en permanence, mais rapidement dégradés par la PNPase [ 8].
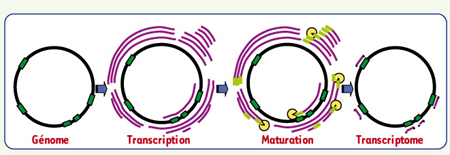
Ainsi, le transcriptome des mitochondries d’Arabidopsis thaliana ne dépend pas directement de la transcription, mais du contrôle de la stabilité des ARN qui s’appuie sur la polyadénylation et la dégradation par la PNPase (Figure 1).
| Figure 1.
Modèle des événements transcriptionnels et post-transcriptionnels menant à un transcriptome mature dans les mitochondries de plante. Le génome est représenté par un cercle, les gènes par des rectangles verts. La transcription donne lieu à une population de transcrits primaires, représentés par des traits violets. Après la polyadénylation, représentée par des ajouts verts, la PNPase, en jaune, dégrade les ARN aberrants et mature les précurseurs. Il en résulte un transcriptome constitué de transcrits matures, dont les extrémités sont ici marquées par des points. |
Pourquoi y a-t-il, apparemment, un tel gaspillage ? On ne peut pas encore répondre à cette question, mais, de fait, un mécanisme similaire de contrôle de qualité des ARN impliquant la polyadénylation a été identifié dans le noyau de la levure. Des ARN transcrits à partir de régions intergéniques du génome nucléaire de Saccharomyces cerevisiae sont rapidement dégradés par l’exosome nucléaire après la polyadénylation de la cible par le complexe TRAMP (Trf4p/Air2p/Mtr4p polyadenylation) [ 9, 10]. Ces données, corrélées à nos découvertes, suggèrent un phénomène ubiquitaire, malgré des organisations génomiques différentes.
Une transcription permissive contrebalancée par un système de surveillance dépendant de la polyadénylation semble ainsi être un mécanisme conservé parmi différents systèmes génétiques.