L’architecture du noyau eucaryote est extrêmement variable selon le type cellulaire. La structure intime de la chromatine est en revanche remarquablement conservée à l’échelle de son unité fonctionnelle, le nucléosome, particule résultant de l’assemblage des quatre types d’histones (H2A, H2B, H3 et H4) et de la molécule d’ADN. Chez de nombreuses espèces, le noyau du spermatozoïde déroge à cette règle fondamentale en adoptant une organisation de sa chromatine radicalement différente. Selon les espèces, les histones sont totalement ou partiellement remplacées dans le noyau du gamète mâle par des petites protéines chromosomiques très basiques appelées protamines. Les protamines permettent à l’ADN d’atteindre un niveau de compaction très élevé. À la fécondation, lorsque le noyau du spermatozoïde est libéré dans le cytoplasme de l’œuf, la chromatine paternelle doit rapidement éliminer ses protamines pour réacquérir une organisation en nucléosomes essentielle pour le développement de l’embryon. Nous avons identifié une mutation chez la drosophile, appelée sésame, qui affecte très spécifiquement ce processus [ 1]. Dans les œufs pondus par des femelles mutantes, le noyau du spermatozoïde provenant d’un mâle sauvage est incapable de décondenser sa chromatine. Le pronucleus mâle, anormalement condensé, est alors exclu de la première mitose zygotique et les embryons ne se développent qu’avec le seul jeu de chromosomes maternels. Nous avons récemment découvert que la mutation sésame affecte le gène Hira qui code un facteur d’assemblage de la chromatine présent chez tous les eucaryotes [ 2]. La mutation induit le remplacement d’un seul résidu très conservé, dans un domaine de HIRA prédit pour interagir avec d’autres protéines. Les propriétés d’assemblage de la chromatine du facteur HIRA ont été récemment caractérisées in vitro par le groupe de Geneviève Almouzni et ses collaborateurs [ 3]. HIRA fait ainsi partie d’un complexe protéique capable d’assembler des nucléosomes indépendamment de la synthèse d’ADN, par opposition au complexe CAF-1, dont la fonction est d’assembler les nucléosomes sur l’ADN en cours de réplication ou de réparation.
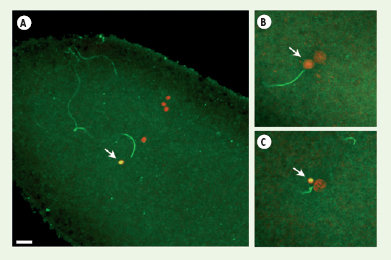
Dans un œuf sauvage de drosophile, le noyau du spermatozoïde doit assembler sa chromatine en remplaçant ses protamines par des histones qui sont fournies par l’œuf. Ce processus, qui s’accompagne de la décondensation de la chromatine paternelle, intervient bien avant que le pronucleus mâle ne réplique son ADN, ce qu’il fait au moment où il rejoint son partenaire femelle. La formation du pronucleus mâle relève donc d’un mode d’assemblage indépendant de la réplication. Ainsi, la découverte de la fonction de HIRA dans le noyau mâle s’accorde avec les propriétés biochimiques de ce facteur telles qu’elles ont été décrites in vitro. Pour confirmer l’implication de HIRA dans l’assemblage de la chromatine paternelle, nous avons étudié la distribution de cette protéine dans les œufs de drosophile [2]. De façon remarquable, HIRA s’accumule très spécifiquement dans le noyau mâle dès le début du processus de décondensation (Figure 1).
| Figure 1.
HIRA permet la décondensation du noyau mâle à la fécondation. A. Dans un œuf sauvage ou mutant pour ssm, la protéine HIRA maternelle s’accumule dans le noyau mâle en cours de décondensation qui apparaît ainsi en jaune (flèche). Le pronucleus femelle en cours de migration est visible ainsi que les trois globules polaires, en position plus périphérique. Le flagelle du spermatozoïde (vert) est également visible sur cette section confocale. B. Dans un œuf pondu par une femelle sauvage, lorsque les pronucleus sont apposés, le pronucleus mâle est décondensé et la protéine HIRA n’y est plus que faiblement détectée (flèche). C. Dans un œuf pondu par une femelle mutante sésame au même stade, le noyau mâle (flèche) apparaît anormalement condensé et HIRA y est encore détectée (barre d’échelle : 10 μm). |
Chez la drosophile, le gène Hira est transcrit dans tous les tissus et à tous les stades de développement. Vraisemblablement, la mutation ponctuelle sésame, qui est viable à l’état homozygote, n’affecte qu’une sous-fonction du complexe HIRA, spécifiquement dévolue à la formation du pronucleus mâle. On sait par ailleurs que l’invalidation de HIRA chez la souris est létale au stade embryonnaire [ 4]. L’obtention de nouveaux allèles mutants de Hira chez la drosophile devrait donc nous permettre d’élucider les autres fonctions de cette protéine.
Chez les vertébrés, HIRA fait partie d’un complexe protéique contenant le variant H3.3 de l’histone H3 [ 5]. Contrairement aux variants de phase S (H3.1 et H3.2 chez les vertébrés, H3 chez la drosophile) qui sont spécifiquement déposés pendant la synthèse d’ADN, les nucléosomes contenant H3.3 peuvent être formés tout au long du cycle cellulaire. Par ailleurs, il est maintenant clairement établi que le variant H3.3 constitue une marque de la chromatine transcriptionnellement active [ 6, 7]. Des expériences de reconstitution de chromatine ont permis de démontrer que HIRA dépose spécifiquement H3.3 indépendamment de la synthèse d’ADN [5]. Afin de vérifier si HIRA se comporte bien comme le chaperon de H3.3 in vivo, nous avons analysé la distribution des deux variants H3 et H3.3 lors de la formation du pronucleus mâle, en contexte sauvage et mutant pour sésame. Nous avons pu ainsi clairement établir que pendant la phase de décondensation du noyau du spermatozoïde dans l’œuf, seul H3.3 est déposé sur la chromatine paternelle et de façon dépendante de HIRA [2].
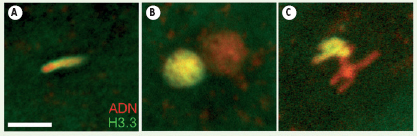
Ainsi, dans le zygote diploïde nouvellement formé, le variant H3.3 distingue clairement les chromosomes paternels des chromosomes maternels qui contiennent essentiellement l’autre variant, H3 (Figure 2). Cette conséquence inattendue de l’implication de HIRA dans la formation du pronucleus mâle soulève plusieurs questions. Tout d’abord, cette marque paternelle existe-t-elle chez d’autres animaux ? Une étude récente suggère que c’est le cas chez la souris, espèce dont le noyau du spermatozoïde est essentiellement organisé avec des protamines, comme chez la drosophile [ 8]. Il est donc très vraisemblable que, d’une manière générale, le remplacement protamine/histones à la fécondation requiert la voie d’assemblage HIRA/H3.3.
| Figure 2.
Le variant d’histone H3.3 est spécifiquement déposé sur la chromatine paternelle à la fécondation. L’histone H3.3 d’origine maternelle est détectée sur le noyau du spermatozoïde au début du processus de décondensation (A), sur le pronucleus mâle (B) et enfin sur les chromosomes paternels lors de la première mitose du zygote (C) (barre d’échelle : 10 μm) |
En corollaire de la découverte de cette marque paternelle se pose le problème de son rôle éventuel pour le développement du futur embryon. Chez la drosophile, nous observons que le variant H3.3 est progressivement remplacé par H3 au cours des phases S successives et il devient indétectable sur les chromosomes paternels après quelques cycles de divisions syncytiales. Si ce marquage joue un rôle chez cette espèce, il reste donc à définir. Chez la souris, en revanche, il vient d’être suggéré que le remplacement des protamines par les histones à la fécondation pourrait participer à l’inactivation du chromosome X paternel dans les embryons femelles [ 9]. Enfin, et de façon plus générale, une meilleure compréhension des mécanismes de l’assemblage de la chromatine paternelle à la fécondation devrait contribuer à améliorer et sécuriser les techniques d’assistance à la reproduction chez l’homme, actuellement en plein essor [ 10].