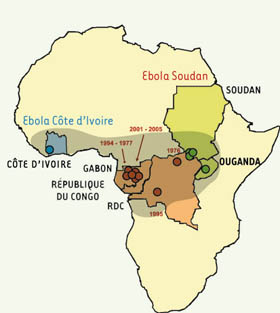
Plusieurs pays des régions forestières équatoriales de l’Afrique sont touchés depuis près de trois décennies par des épidémies de fièvre hémorragique à virus Ebola. Ce virus, à la forme caractéristique de filament, possède pour matériel génétique un brin d’ARN de polarité négative, et est subdivisé en quatre sous-types dont trois sévissent en Afrique, Ebola Soudan, Ebola Zaïre, Ebola Côte d’Ivoire [ 1] (Figure 1). L’infection de l’homme par le virus Ebola s’exprime globalement par un syndrome fiévreux aigu accompagné de troubles digestifs et respiratoires graves et de signes hémorragiques multiples. La mort survient généralement chez 8 malades sur 10 pour Ebola Zaïre et 1 malade sur 2 pour Ebola Soudan. Cette maladie est une zoonose transmise à l’homme de manière accidentelle lors d’un contact direct avec un animal infecté, et se manifeste le plus souvent sous la forme de flambées épidémiques. Depuis la découverte du virus Ebola en 1976, 14 flambées épidémiques (10 dues à Ebola Zaïre et 4 à Ebola Soudan) ont affecté le continent africain, dont 8 la seule région frontalière du Gabon et de la République du Congo entre 1995 et 2005. Les épidémies survenues entre 2001 et 2005 dans cette région ont également touché certaines espèces animales dont les gorilles, les chimpanzés et les céphalophes (petites antilopes de forêt). Plusieurs centaines, voire des milliers d’animaux auraient péri pendant cette période du fait d’Ebola, à tel point que l’on assiste à un déclin dramatique des populations naturelles de grands singes peuplant cette région [ 2, 3]. Des travaux publiés dans Science en janvier 2004 ont montré que les nombreuses carcasses animales qui en ont résulté ont constitué les sources de contamination des épidémies humaines survenues entre 2001 et 2005 dans cette région [3].
| Figure 1.
Historique des épidémies de fièvre hémorragique Ebola en Afrique. Le cas humain isolé dû au sous-type Côte d’Ivoire est représenté en bleu, les épidémies dues au sous-type Soudan en vert et les épidémies dues au sous-type Zaïre en rouge. Les captures mentionnées dans le texte se sont déroulées pendant les épidémies de 2001-2005. L’aire en gris représente l’aire de répartition des trois espèces de chauves-souris incriminées : Hypsignathus monstrosus, Epomops franqueti et Myonycteris torquata. |
Malgré les avancées déterminantes réalisées ces dernières années par notre équipe dans la compréhension du cycle du virus Ebola, la source initiale du virus, c’est-à-dire son réservoir, était resté jusqu’à ce jour inconnu bien que de nombreuses études aient été réalisées dans ce sens depuis 1976 [ 4]. Dans cette optique, notre équipe a effectué, au cours des années 2002 et 2003, trois missions de captures d’animaux dans deux zones forestières touchées par les différentes épidémies survenues entre 2001 et 2004. Les captures d’animaux se sont effectuées dans un rayon d’une dizaine de kilomètres autour d’une carcasse de gorille infectée par le virus Ebola, se sont étendues sur 3 semaines, et ont débuté quelques jours après la découverte de l’animal. Les conditions étaient donc réunies pour que les captures se déroulent au plus fort de la circulation du virus dans son milieu naturel. Au total, 1 030 animaux ont été capturés, autopsiés et analysés. Les captures et les analyses de laboratoire se sont échelonnées sur 4 années [ 5].
Ces analyses ont montré que trois espèces de chauves-souris frugivores sont asymptomatiquement infectées par le virus Ebola : Hypsignathus monstrosus, Epomops franqueti et Myonycteris torquata. Ainsi, des IgG anti-Ebola on été détectées dans le sérum de 16 chauves-souris dont 4 Hypsignathus, 8 Epomops et 4 Myonycteris, alors qu’elles n’ont été retrouvées dans aucune autre espèce de chauve-souris ni aucune autre espèce animale. De même, des séquences nucléotidiques virales ont été détectées dans les organes de 13 chauves-souris dont 3 Hypsignathus, 5 Epomops et 5 Myonycteris. Le séquençage des fragments amplifiés a confirmé la spécificité des séquences. L’analyse phylogénique de ces séquences par les méthodes bayésienne et maximum de parcimonie a montré leur appartenance au « sous-type Zaïre » du virus Ebola [5].
Même si ces travaux, publiés dans Nature en décembre 2005, n’ont pu aboutir à l’isolement du virus, ils constituent les premières preuves biologiques identifiant certaines espèces de chauves-souris frugivores comme réservoir du virus Ebola. Ils complètent certains indices épidémiologiques recueillis lors des épidémies antérieures [ 6], concordent avec les aires de répartition de ces espèces recouvrant les régions épidémiques (Figure 1) et confirment les études ayant mis en évidence une virémie transitoire chez certaines espèces de chauves-souris après infection expérimentale par le virus Ebola [ 7]. Enfin, il est intéressant de constater que des chauves-souris frugivores sont également réservoir des virus Hendra et Nipah de la famille des Paramyxoviridae [ 8, 9], et du virus de la rage de la famille des Rhabdoviridae, deux familles virales génétiquement proches des Filoviridae [ 10].
Les enquêtes épidémiologiques menées sur le terrain ont montré que les mortalités de grands singes dues au virus Ebola apparaissent souvent à la fin des saisons sèches, période où les ressources alimentaires se raréfient. Un tel appauvrissement alimentaire favoriserait la consommation des mêmes fruits, aux mêmes moments, par différentes espèces animales au régime frugivore, telles que les chauves-souris et les grands singes. Ces rassemblements augmenteraient alors la probabilité de contact entre ces deux espèces. À cet appauvrissement alimentaire se surajoutent plusieurs événements comportementaux et physiologiques chez les espèces de chauves-souris incriminées, telles que les compétitions intraspécifiques entre mâles et les mises bas groupées des femelles. L’ensemble de ces facteurs modifierait la nature et le niveau des réponses immunitaires, propices à la reprise de la réplication virale dans les organes cibles, voire à l’apparition de virus dans la circulation sanguine. La contamination des grands singes interviendrait alors à la faveur de contacts directs avec le sang et/ou les tissus placentaires des femelles chauves-souris au moment de la parturition.
L’identification des chauves-souris frugivores comme réservoirs du virus Ebola permettra de concevoir puis de mettre en place des stratégies adaptatives de prévention des épidémies auprès de populations habituées à consommer ces animaux, ainsi que des stratégies visant à diminuer le passage du virus aux grands singes dont les populations sauvages se trouvent menacées dans les régions endémiques.