| |
| Med Sci (Paris). 2005 October; 21(10): 860–865. Published online 2005 October 15. doi: 10.1051/medsci/20052110860.Luca : À la recherche du plus proche ancêtre commun universel Patrick Forterre,1* Simonetta Gribaldo,1,2 and Céline Brochier3 1Biologie Moléculaire du gène chez les extrêmophiles (BMGE), Département de Microbiologie fondamentale et médicale, Institut Pasteur, 28, rue du Docteur Roux, 75724 Paris Cedex 15, France 2Atelier de Bio-informatique, Université Paris 6, 12, rue Cuvier, 75005 Paris, France 3EA EGEE (évolution, génome, environnement), Université de Provence Aix- Marseille I, Centre Saint-Charles, Marseille, France |
L’une des principales avancées de la biologie au XXe siècle a été de mettre en évidence l’unité du monde vivant : tous les êtres vivant actuellement sont formés des mêmes macromolécules, utilisent la même machinerie de synthèse protéique et le même code génétique, ce qui suggère qu’ils descendent tous d’une même lignée d’ancêtres communs. Cela n’implique pas, cependant, que la vie soit apparue une seule fois sur notre planète : en effet, la question de l’origine unique ou multiple de la vie, de même que celle des mécanismes de son apparition, sont toujours très discutées. Toutefois, si d’autres formes de vie sont apparues sur Terre, elles n’ont pas laissé de descendants, et ont toutes été éliminées par celle que nous connaissons. La lignées des ancêtres communs à tous les êtres vivants actuels s’est terminée avec un dernier ancêtre, que l’on appelle généralement Luca, the last universal common ancestor. Ce terme, proposé lors d’un colloque organisé en 1996 à la Fondation des Treilles [
1,
2], est de plus en plus utilisé aujourd’hui, pour son côté médiatique (de Luca à Lucy) comme pour sa précision scientifique. Les autres termes utilisés dans le passé avaient l’inconvénient de préjuger de la nature de notre dernier ancêtre (c’est le cas du terme progénote, utilisé par Carl Woese [
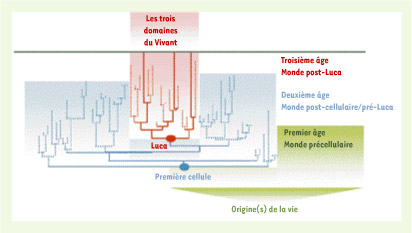
3], qui renvoie à un ancêtre très primitif), ou d’être imprécis (c’est le cas du terme cenancestor, « ancêtre commun », qui ne précise pas de quel ancêtre il s’agit). L’adjectif « dernier » est particulièrement important, car il permet d’éviter la confusion, fréquente, entre Luca et la première cellule apparue sur notre planète (Figure 1) ; « dernier » signifie « le plus proche de nous dans le temps » : c’est après Luca que les trois grands domaines du vivant reconnus actuellement, Archaea, Bacteria et Eucarya, se sont différenciés.  | Figure 1.
É
volution de la vie sur terre depuis son (ou ses) origine(s). Trois stades évolutifs (c’est-à-dire trois grandes étapes) peuvent être distingués : le premier âge, incluant toutes les premières formes de vie non cellulaires (Monde précellulaire), le second âge, qui regroupe des descendants de la première cellule (Monde post-cellulaire/pré-Luca) et qui s’achève avec Luca, et, enfin, le troisième âge, qui commence avec Luca et se poursuit jusqu’à nos jours (Monde post-Luca). Il est important de noter que les représentants de différents grades ont pu coexister à certaines périodes : ainsi, les premières cellules (représentants du second âge) ont très probablement cohabité avec leurs cousins acellulaires, descendants du premier âge ; de même, les descendants de Luca ont du coexister un temps avec leurs cousins du second âge. La question de la durée de ces cohabitations reste totalement ouverte. |
Certains auteurs ont parfois eu tendance à rejeter l’idée d’un Luca unique, pour lui substituer celle d’une communauté de cellules primitives ayant toutes contribué à l’émergence des trois domaines. Il est bien évident que nous ne devons pas avoir une vision simpliste de Luca : tout comme l’Ève mitochondriale ne vivait pas seule dans son village en Afrique, Luca n’était pas le seul organisme présent sur Terre à son époque. De même que les descendants d’Ève ont intégré pendant encore longtemps des gènes nucléaires provenant d’autres lignées féminines aujourd’hui disparues, les descendants de Luca ont dû intégrer pendant un certain temps les gènes provenant de différentes lignées cellulaires, éliminées ensuite par des descendants plus tardifs de Luca. Quoi qu’il en soit, de même que la génétique des populations et notre mode de reproduction implique l’existence d’une Ève mitochondriale unique pour tous les humains actuels, le mécanisme fondamental de la vie cellulaire (toute cellule provient d’une autre cellule) implique l’existence d’un Luca unique. C’est Carl Woese qui a le premier, dans les années 70, souligné l’importance des recherches visant à déterminer la nature de Luca [3]. Ce dernier représente en effet une étape historique intermédiaire entre l’apparition de la vie sur Terre et le temps présent : reconstruire Luca revient donc à poser un jalon essentiel dans l’histoire de la vie. À partir de là, il serait a priori plus facile de remonter encore plus loin dans le temps, vers nos origines, et de déterminer comment les mécanismes moléculaires modernes ont évolué. Si l’on admet, avec Dobzhansky, que « rien n’a de sens en biologie en dehors de l’évolution » [
4], on doit admettre également l’importance de l’enjeu : il est en effet impossible de comprendre complètement un objet biologique si l’on ne connaît pas son origine et son évolution, car tout objet biologique est un produit historique. |
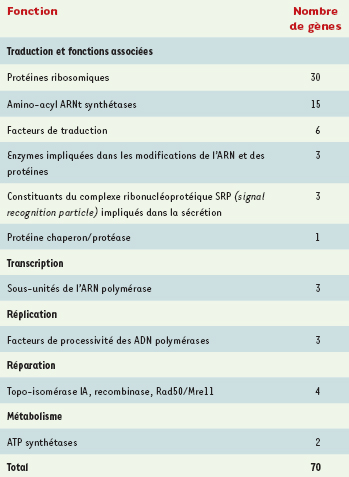
Les protéines universelles Le développement récent de la génomique comparative à grande échelle a, pour la première fois, permis d’aborder le problème de Luca en disposant de données objectives. Ces travaux ont notamment abouti à la détermination du nombre et de la nature des protéines universelles, c’est-à-dire dont les homologues sont présents chez tous les êtres vivants, et qui donc étaient très certainement déjà présentes chez Luca. L’estimation du nombre de ces protéines universelles peut varier selon les critères utilisés pour déterminer si deux protéines sont homologues, et selon les critères requis pour conclure à l’universalité d’une protéine : doit-elle être présente dans tous les génomes séquencés sans exception (plusieurs centaines aujourd’hui), ou peut-on tolérer son absence, par perte supposée ou remplacement non homologue, dans un certain nombre de génomes ? Quels que soient les critères retenus, le nombre de protéines universelles reconnues est toujours rarement supérieur à une centaine, et se situe plutôt autour de 60 à 80 [
5]. Ces chiffres, qui correspondent au nombre minimum de protéines héritées de Luca et conservées au cours de l’évolution au sein des trois domaines, sont très probablement fortement sous-estimés : il est évident que Luca possédait d’autres gènes qui ont pu être perdus ou remplacés par des analogues fonctionnels dans l’un ou l’autre des trois domaines (et même dans les trois, voir plus loin la discussion sur les protéines 3R). Ces gènes, qui devaient notamment lui permettre d’interagir avec son environnement ou être liés à son métabolisme, ne sont pas précisément identifiables aujourd’hui. Les 60 à 80 protéines universelles (Tableau I) sont à plus de 80 % des protéines impliquées dans la traduction : nombreuses protéines ribosomiques, facteurs d’élongation de la chaîne polypeptidique, amino-acyl ARNt synthétases et quelques enzymes de modification des ARN de transfert. Luca possédait donc un mécanisme de synthèse des protéines proche des mécanismes contemporains, capable de produire des protéines relativement sophistiquées. On retrouve également, parmi les protéines universelles, la protéine SRP54, appartenant au complexe ribonucléoprotéique SRP (signal recognition particle), et Sra, son récepteur membranaire, facteurs permettant la translocation membranaire des protéines sécrétées en cours de synthèse : cette observation suggère fortement que Luca était déjà un organisme cellulaire, entouré par une membrane cytoplasmique [
6]. Cette hypopthèse est d’ailleurs corroborée par la présence, dans la liste des protéines universelle, des ATP synthétases membranaires, et par la complexité du système de synthèse protéique : il semble en effet très peu probable qu’un organite tel que le ribosome ait pu apparaître et évoluer dans un monde acellulaire.  | Tableau I.
Protéines ubiquitaires dans les trois domaines du vivant (d’après [ 5]). |
Bien que tous les êtres vivants actuels soient capables d’un métabolisme propre, la liste des protéines universelles ne comprend pratiquement pas d’enzymes du métabolisme. Cela n’est pas si surprenant, car les études phylogénétiques de ces protéines montrent qu’elles ont été fréquemment perdues ou redistribuées entre lignées cellulaires par transfert de gène, ce qui ne permet pas d’inférer le métabolisme de Luca à partir de la seule génomique comparée. |
Un point important encore à résoudre pour mieux comprendre Luca concerne la nature des caractères ou des mécanismes moléculaires communs aux archées et aux eucaryotes (comme, par exemple, l’utilisation d’ARN guides pour la méthylation des ARN ribosomiques) : s’agit-il de caractères nouveaux partagés (synapomorphies), apparus après Luca dans une branche commune aux eucaryotes et aux archées, ou de caractères anciens (synplésiomorphies) hérités de Luca, et perdus ou modifiés secondairement chez les bactéries ? Dans le premier cas, on doit conclure à un Luca relativement simple, dont les ribosomes ne comportaient que les protéines présentes dans la liste des protéines universelles (entre 30 et 40). Dans le second cas, en revanche, on peut imaginer un Luca plus complexe, dont le ribosome comprenait déjà toutes les protéines communes aujourd’hui aux archées et aux eucaryotes (une soixantaine). Il est aujourd’hui difficile de trancher en l’absence d’un groupe extérieur permettant de polariser les caractères communs aux archées et eux eucaryotes. On peut toutefois espérer que le ribosome a conservé la mémoire de notre plus ancienne histoire, et que la comparaison de sa structure fine (i.e. interactions au niveau atomique entre ses composants protéiques et l’ARN) dans les trois domaines (elle n’est connue pour le moment que chez les bactéries et les archées) permettra de choisir entre les scénarios évolutifs possibles. Enfin, certains imaginent que nous sommes les descendants d’une chimère issue de la fusion d’une bactérie et d’une archée [
7,
8], et que seuls les procaryotes dérivent directement de Luca. Il reste alors à expliquer l’origine des nombreuses protéines spécifiques des eucaryotes et la formation des structures complexes qui caractérisent ces derniers (membrane et pore nucléaires, spliceosome…) à partir de systèmes procaryotes plus simples. D’autres auteurs, dont nous faisons partie, imaginent plutôt l’apparition de ces structures dans une lignée spécifique aux eucaryotes, certaines d’entre elles ayant pu apparaître dans le monde à ARN [
9]. |
Luca possédait-il déjà un génome à ADN ? Le point le plus inattendu, lorsqu’on examine la liste des protéines universelles, est le petit nombre de protéines impliquées dans le métabolisme de l’ADN. Bien que tous les aspects de ce métabolisme soient présents dans les trois domaines, les acteurs (enzymes) impliqués sont rarement homologues dans les trois domaines à la fois (c’est-à-dire que ce sont des analogues fonctionnels ayant des origines évolutives distinctes), ce qui explique qu’ils n’apparaissent pas dans la liste. C’est le cas des ribonucléotides réductases (synthèse des ribonucléotides), des thymydylate synthétases (synthèse du dTMP) ou des enzymes impliquées dans la réplication, la réparation ou la recombinaison de l’ADN (protéines 3R). Les trois protéines essentielles agissant au niveau des fourches de réplication (ADN polymérases, primases et hélicases), notamment, sont homologues entre archées et eucaryotes, mais ne le sont pas entre archées/eucaryotes et bactéries [
10]. Cette observation a conduit Mushegian et Koonin à suggérer, en 1996, que Luca possédait encore un génome à ARN, et que l’ADN aurait été inventé deux fois, dans la branche des bactéries et dans une branche commune aux archées et aux eucaryotes [
11]. Pour expliquer la présence, dans la liste des protéines universelles, des ARN polymérases impliquées dans la transcription de l’ADN et de quelques protéines 3R, Koonin et ses collaborateurs ont proposé un Luca dont le génome était composé d’ARN, mais qui contenait déjà de l’ADN répliqué par rétrotranscription [10] : ce ne serait donc que le mécanisme actuel de réplication direct ADN-ADN qui aurait été inventé deux fois. Pendant longtemps, l’idée d’un Luca à ARN a été accueillie avec scepticisme, en raison de l’infidélité supposée de la réplication de l’ARN et de l’absence de mécanismes de réparation de l’ARN. Il était notamment admis que les ARN polymérases, contrairement aux ADN polymérases, ne pouvaient pas corriger leurs erreurs, et qu’une cellule à ARN n’était alors pas compatible avec la complexité supposée d’un organisme tel que Luca. Toutefois, des mécanismes spécifiques de réparation de l’ARN ont été mis en évidence ces dernières années, et il est apparu que les ARN polymérases possédaient également des mécanismes de correction d’erreur (pour revue, voir [
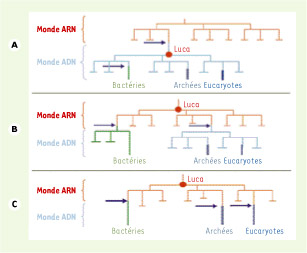
12]) : on peut donc aujourd’hui envisager l’existence d’un Luca à ARN complexe sans être confronté au problème insurmontable de l’infidélité des ARN polymérases. L’hypothèse d’un Luca possédant un génome à ADN reste toutefois possible, à condition que les ADN polymérases, primases et hélicases ancestrales présentes chez Luca aient ensuite été remplacées par des analogues fonctionnels non homologues, soit chez les bactéries, soit dans une lignée commune aux archées et aux eucaryotes (Figure 2A). Ces protéines « remplaçantes » auraient pu provenir de virus à ADN [
13] : en effet, il est frappant de constater que les virus à ADN codent généralement pour leurs propres protéines 3R, qui sont parfois non homologues à leurs analogues fonctionnels cellulaires (ainsi la primase du virus de l’herpès n’est pas homologue aux primases bactériennes, ni aux primases de type archée/eucaryote). Certes, le remplacement de protéines impliquées dans des mécanismes fondamentaux par des protéines d’origine virale peut paraître surprenant au premier abord ; néanmoins, il a été démontré qu’au cours de l’évolution des mitochondries, l’ARN polymérase, l’ADN polymérase et la primase de l’a-protéobactérie ancestrale ont été remplacées par une ADN polymérase, une primase et une hélicase provenant toutes trois d’un virus apparenté aux bactériophages T3/T7 [
14]. Et l’idée selon laquelle les virus seraient intervenus à une étape précoce de l’évolution du monde vivant est en accord avec l’hypothèse selon laquelle les virus existaient avant l’apparition de Luca [
15,
16], une hypothèse étayée par la découverte d’homologies entre des virus infectant des cellules appartenant à différents domaines. Ainsi, d’indéniables similarités de structure au niveau des protéines de capside ont pu être mises en évidence entre certains bactériophages, un virus d’archée hyperthermophile et des virus eucaryotes [
17,
18]. 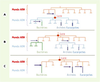 | Figure 2.
Hypothèses de l’origine des génomes à ADN dans les trois domaines par transfert à partir de virus à ADN. Les transferts à partir de virus à génome ADN sont matérialisés par les flèches violettes. Trois hypothèses peuvent être envisagées. A. L’acquisition de l’ADN à partir d’un seul virus s’est produite avant Luca (qui avait donc un génome à ADN). La non-homologie des protéines 3R bactériennes avec les analogues fonctionnels archébactériens et eucaryotes serait due à un replacement non homologue par des protéines d’origine virale qui se serait produit secondairement dans la lignée bactérienne. B. L’acquisition de l’ADN s’est produite deux fois indépendamment à partir de deux virus différents (une fois dans la lignée bactérienne et une fois dans la lignée commune aux eucaryotes et aux archées). C. L’acquisition de l’ADN s’est produite trois fois indépendamment à partir de trois virus différents (dans chacun des trois domaines). |
Pour expliquer la présence d’une grande variété de protéines 3R chez les virus, l’un d’entre nous a récemment proposé que l’ADN lui-même aurait pu être inventé par un virus à ARN, comme forme de résistance aux mécanismes dirigés par l’hôte (une cellule ARN) contre le génome viral, et que les mécanismes de réplication et les protéines 3R seraient apparus dans un monde à ADN viral qui aurait précédé le monde à ADN cellulaire [
19]. Dans cette hypothèse, le transfert de l’ADN des virus aux cellules se serait produit au moins deux fois, ce qui permet d’expliquer les différences observées entre les protéines de réplication des bactéries et des archées/eucaryotes. Le moment où ces transferts se seraient produits reste toutefois indéterminé. On peut imaginer qu’un premier transfert ait eu lieu avant Luca (ce qui implique que Luca avait un génome à ADN), et que les protéines de réplication présentes chez Luca aient ensuite été remplacées par de nouvelles protéines de réplication (second transfert), soit dans la lignée bactérienne, soit dans celle des archées/eucaryotes (Figure 2A). On peut également imaginer que les deux transferts aient pu se produire après Luca, en accord avec les idées de Woese et Koonin sur un Luca dont le génome était encore constitué d’ARN (Figure 2B). Enfin, un dernier scénario a récemment été proposé, celui de trois transferts indépendants [
20] : dans ce modèle, le remplacement de l’ARN par l’ADN se serait produit dans trois lignées différentes de cellules à ARN et aurait impliqué trois virus différents (Figure 2C). Ces transferts auraient été ainsi à l’origine des trois domaines actuels. L’un des modèles proposés correspond-il à la réalité ? Luca était-il toujours membre du monde à ARN ou possédait-il déjà de l’ADN ? La question reste ouverte. |
Conclusions : l’exploration continue… La génomique comparée a donné une impulsion nouvelle aux travaux sur Luca. Ces dernières années, l’exploration du monde vivant a également apporté des informations inattendues. Ainsi, des structures similaires au noyau eucaryote ont été découvertes chez des bactéries du groupe des Planctomycetales [
21,
22], ouvrant un débat sur l’existence possible d’un Luca à noyau. De même, des gènes codant pour la tubuline a et b (considérée comme spécifique des eucaryotes) ont été identifiés chez des bactéries du groupe des Verrucomicrobiales (pour une revue récente sur les éléments du cytosquelette chez les bactéries, voir [
23]). Ces découvertes ont rendu la frontière entre le monde eucaryote et le monde procaryote plus floue. De même, la découverte récente d’un virus géant, le Mimivirus, dont le génome fait 1,2 Mb (ce qui est trois fois plus grand que les plus petits génomes décrits chez des organismes cellulaires), soulève à nouveau la question de la nature des virus et de leur rôle dans l’évolution [
24], le génome du Mimivirus contenant des gènes codant pour des protéines présentes dans les trois domaines cellulaires, dont quelques protéines impliquées dans la traduction ; un arbre universel fondé sur ces protéines positionne le Mimivirus entre les archées et les eucaryotes [24]. Ce résultat est à rapprocher de l’hypothèse récente selon laquelle le noyau des cellules eucaryotes pourrait être d’origine virale [
25–
27]. Le Mimivirus, qui infecte aujourd’hui une amibe, est-il une relique d’un quatrième domaine cellulaire aujourd’hui disparu ? Ou encore un proche parent du virus ayant donné naissance aux cellules eucaryotes ? Plus prosaïquement, est-il le descendant d’un très vieux virus qui a capturé des gènes d’anciennes lignées eucaryotes ? (voir [
28] et [
29] pour une discussion intéressante autour de cette controverse). Le séquençage d’un plus grand nombre de génomes (en particulier chez les protistes et les virus), ainsi que l’obtention de données expérimentales, devraient permettre de répondre à ces questions. |
3. Woese CR, Fox GE. The concept of cellular evolution. J Mol Evol 1977; 10 : 1–6. 4. Dobzhansky T. Nothing in biology makes sense, except in the light of evolution. Amm Biol Teachers 1973; 35 : 125–9. 5. Koonin EV. Comparative genomics, minimal gene-sets and the last universal common ancestor. Nat Rev Microbiol 2003; 1 : 127–36. 6. Pereto J, Lopez-Garcia P, Moreira D. Ancestral lipid biosynthesis and early membrane evolution. Trends Biochem Sci 2004; 29 : 469–77. 7. Lopez-Garcia P, Moreira D. On hydrogen transfer and a chimeric origin of eukaryotes. Trends Biochem Sci 1999; 24 : 424. 8. Rivera MC, Lake JA. The ring of life provides evidence for a genome fusion origin of eukaryotes. Nature 2004; 431 : 152–5. 9. Penny D, Poole A. The nature of the last universal common ancestor. Curr Opin Genet Dev 1999; 9 : 672–7. 10. Leipe DD, Aravind L, Koonin EV. Did DNA replication evolve twice independently ? Nucleic Acids Res 1999; 27 : 3389–401. 11. Mushegian AR, Koonin EV. A minimal gene set for cellular life derived by comparison of complete bacterial genomes. Proc Natl Acad Sci USA 1996; 93 : 10268–73. 12. Poole AM, Logan DT. Modern mRNA proofreading and repair: clues that the last universal common ancestor possessed an RNA genome ? Mol Biol Evol 2005; 22 : 1444–55. 13. Forterre P. Displacement of cellular proteins by functional analogues from plasmids or viruses could explain puzzling phylogenies of many DNA informational proteins. Mol Microbiol 1999; 33 : 457–65. 14. Filee J, Forterre P, Laurent J. The role played by viruses in the evolution of their hosts: a view based on informational protein phylogenies. Res Microbiol 2003; 154 : 237–43. 15. Bamford DH. Do viruses form lineages across different domains of life ? Res Microbiol 2003; 154 : 231–6. 16. Forterre P. The great virus comeback: from an evolutionary perspective. Res Microbiol 2003; 154 : 223–5. 17. Merckel MC, Huiskonen JT, Bamford DH, et al. The structure of the bacteriophage PRD1 spike sheds light on the evolution of viral capsid architecture. Mol Cell 2005; 18 : 161–70. 18. Rice G, Tang L, Stedman K, et al. The structure of a thermophilic archaeal virus shows a double-stranded DNA viral capsid type that spans all domains of life. Proc Natl Acad Sci USA 2004; 101 : 7716–20. 19. Forterre P. The origin of DNA genomes and DNA replication proteins. Curr Opin Microbiol 2002; 5 : 525–32. 20. Forterre P. The two ages of the RNA world, and the transition to the DNA world: a story of viruses and cells. Biochimie 2005 (sous presse). 21. Fuerst JA, Webb R. Membrane-bounded nucleoid in eubacterium Gemmata obscuriglobus. Proc Natl Acad Sci USA 1991; 88 : 8184–8. 22. Lindsay MR, Webb RI, Strous M, et al. Cell compartmentalisation in planctomycetes: novel types of structural organisation for the bacterial cell. Arch Microbiol 2001; 175 : 413–29. 23. Lowe J, van den Ent F, Amos LA. Molecules of the bacterial cytoskeleton. Annu Rev Biophys Biomol Struct 2004; 33 : 177–98. 24. Raoult D, Audic S, Robert C, et al. The 1.2-megabase genome sequence of Mimivirus. Science 2004; 306 : 1344–50. 25. Bell PJ. Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus ? J Mol Evol 2001; 53 : 251–6. 26. Claverie JM. Un virus encore plus géant que les autres. Med Sci (Paris) 2005; 21 : 15–6. 27. Takemura M. Poxviruses and the origin of the eukaryotic nucleus. J Mol Evol 2001; 52 : 419–25. 28. Moreira D, Lopez-Garcia P. Comment on The 1.2-megabase genome sequence of Mimivirus. Science 2005; 308 : 1114. 29. Raoult D, Audic S, Robert C, et al. Response to comment on the 1.2 megabase genome sequence of Mimivirus. Science 2005; 308 : 1114b. |