| |
| Med Sci (Paris). 2005 June; 21(6-7): 613–617. Published online 2005 June 15. doi: 10.1051/medsci/2005216-7613.Repliement et production de protéines recombinantes Jean-Michel Betton* and Alain Chaffotte Unité de repliement et modélisation des protéines, Institut Pasteur, 28, rue du Docteur Roux, 75724 Paris Cedex 15, France |
Avec l’essor du génie génétique au début des années 70 est apparue la possibilité de programmer une cellule pour synthétiser des protéines d’intérêt. Ainsi, au début des années 80, l’insuline fut la première protéine recombinante humaine produite dans une bactérie [
1]. Aujourd’hui, l’ère du post-génome a profondément modifié ce domaine, qui ne se limite plus à quelques protéines d’intérêt pharmaceutique, mais implique toutes les sciences du vivant où l’étude simultanée de plusieurs centaines de protéines est nécessaire [
2]. Cependant, la conversion de l’information génétique en une protéine biologiquement active, tant pour la découverte de fonctions inconnues que pour l’identification de nouvelles cibles thérapeutiques, reste un défi d’actualité [
3]. |
Jusqu’au milieu des années 90, la bactérie était l’hôte de choix pour produire une protéine recombinante. Cette position dominante découlait essentiellement de ses propriétés de croissance rapide, de son faible coût de production, de sa génétique très étudiée, et des règles de sécurité imposées pour la manipulation de l’ADN recombinant. Actuellement, avec le développement des technologies de cultures cellulaires à grande échelle, environ 60 % à 70 % des protéines d’intérêt pharmaceutique sont produites dans les cellules animales. L’autre raison de ce changement est l’accroissement de la demande pour les anticorps recombinants humains ou humanisés [
4], qui sont fréquemment peu produits dans la bactérie. Le choix du système d’expression est principalement guidé par les modifications que la protéine doit subir pour être biologiquement active. Bien qu’il existe des techniques permettant le transfert rapide des gènes clonés entre différents systèmes d’expression, le vecteur génétique, utilisé pour exprimer le gène recombinant, doit être adapté à l’hôte choisi. De même, il est important de connaître le compartiment cellulaire dans lequel la protéine d’intérêt se replie ou exerce son activité biologique naturelle. Selon que cette protéine est cytoplasmique, membranaire ou se replie dans un compartiment extracytoplasmique (Figure 1), le vecteur devra contenir des séquences spécifiques permettant l’adressage correct de la protéine. À ce stade, l’utilisation d’outils informatiques prédictifs facilite l’analyse de la structure primaire des protéines en apportant des indications quant à sa localisation cellulaire. Ainsi, la présence d’une hélice hydrophobe indique que la protéine est insérée dans une membrane, ou doit éventuellement la traverser au cours de sa biosynthèse pour atteindre un compartiment cellulaire différent du cytoplasme dans lequel elle est synthétisée. La construction du vecteur génétique devra donc tenir compte de tous ces éléments indispensables à l’adressage correct de la protéine dans l’hôte cellulaire, surtout en cas d’expression hétérologue. La glycosylation des protéines oriente, évidemment, vers une production extracytoplasmique dans les systèmes d’expression eucaryote bien documentés, comme les levures [
5], les cellules de mammifères [
6] ou encore les cellules végétales [
7]. Cependant, une modification post-traductionnelle souvent négligée au sein des structures protéiques est la formation des ponts disulfures. Indépendamment du système utilisé, procaryote ou eucaryote, il n’est pas rare de trouver dans la littérature des essais de production infructueux, dans lesquels la protéine est produite dans un compartiment cellulaire inapproprié ne permettant pas cette modification. La formation des ponts disulfures est catalysée par un complexe enzymatique d’oxydoréductases présent dans un compartiment métaboliquement oxydant, qui est soit le périplasme [
8] pour les bactéries Gram-négative, soit le réticulum endoplasmique [
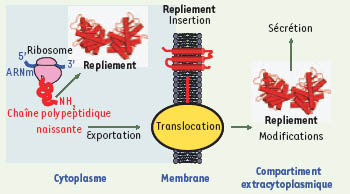
9] pour les cellules eucaryotes. Les protéines possédant des ponts disulfures sont généralement actives dans un milieu extracytoplasmique, et cette liaison covalente est le plus souvent nécessaire à leur stabilité. Par conséquent, la production des protéines à ponts disulfures dans l’environnement réducteur du cytoplasme conduit le plus souvent à des problèmes de repliement.  | Figure 1.
Voies de repliement cellulaire des protéines. Dans son contexte cellulaire, le repliement des protéines est plus complexe que dans un tube à essais. La compartimentation cellulaire, qui est une source de complexité rarement mimée dans les études de repliement in vitro, impose un contrôle précis du repliement des protéines qui doivent s’insérer ou franchir une membrane lipidique au cours de leur biogenèse. |
À côté du choix eucaryote versus procaryote, une troisième possibilité a été apportée récemment par l’amélioration des systèmes acellulaires qui, fondés sur le couplage des réactions de transcription et traduction in vitro, ont aujourd’hui des rendements de synthèse élevés [
10]. Bien qu’ils soient onéreux, ces systèmes permettent la production de protéines toxiques et offrent une flexibilité incomparable, par rapport aux systèmes cellulaires, pour manipuler les paramètres d’expression [
11]. Enfin, le choix d’un système d’expression repose sur d’autres critères comme sa simplicité d’utilisation, le coût de production et de purification de la protéine, la capacité de contrôler le produit final et les modifications introduites, le temps requis du clonage du gène à la protéine purifiée, et les contraintes réglementaires. |
Formation des corps d’inclusion Pour remplir leur fonction, les protéines doivent adopter une conformation tridimensionnelle précise, l’état natif, acquise au cours du repliement cellulaire. La synthèse des protéines au niveau des ribosomes est un processus universellement conservé, ce qui permet à une bactérie de produire des protéines humaines [
12], par exemple. Cependant, de nombreuses protéines ne sont pas produites dans leur état natif, mais s’agrègent dans un état le plus souvent biologiquement inactif, que l’on appelle corps d’inclusion. Ce phénomène d’agrégation est le principal obstacle à la production de protéines et, contrairement à ce qui est couramment admis, la formation des corps d’inclusion n’est pas l’apanage des systèmes bactériens, mais s’observe également dans des cellules eucaryotes. Le mécanisme moléculaire de l’agrégation des protéines recombinantes suggère que les chaînes polypeptidiques naissantes, adoptant des conformations partiellement repliées et instables (les intermédiaires de repliement) (→), peuvent emprunter deux voies alternatives et compétitives, selon une partition cinétique [
13] : le repliement correct, qui conduit à la forme native, ou le repliement incorrect, qui conduit à l’agrégation. Les corps d’inclusion sont des agrégats protéiques non ordonnés [
14], relativement homogènes et denses ; ils sont de plus en plus considérés comme des systèmes en équilibre dynamique [
15] entre association et dissociation d’espèces partiellement ou incorrectement repliées (Figure 2). La concentration très élevée des chaînes polypeptidiques naissantes dans une cellule recombinante favorise les réactions d’agrégation aux dépens de la réaction productive vers l’état natif. (→) m/s 2005, n° 6-7, p.601)
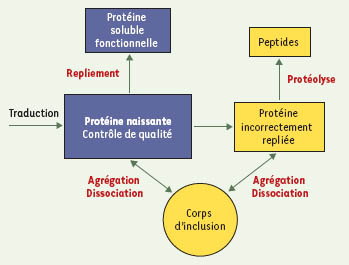
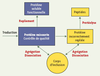 | Figure 2.
Formation des corps d’inclusion. Illustration, à partir d’une chaîne polypeptidique naissante, des différentes réactions conduisant à la distribution cellulaire des protéines entre les états repliés, agrégés ou dégradés. |
Dans le contexte cellulaire, le repliement est facilité par un ensemble d’acteurs : les chaperons, qui assistent les protéines en cours de repliement (→) des catalyseurs, qui interviennent sur les étapes lentes de ce processus, les oxydoréductases, qui catalysent la formation des ponts disulfures, et les peptidyl-prolyl isomérases, qui catalysent l’isomérisation cis-trans des liaisons peptidiques [
16]. En agissant sur les conformations intermédiaires, les protéines chaperons contrôlent directement le processus de repliement, en collaboration avec les protéases cellulaires. En effet, les protéines incorrectement repliées sont également les substrats cellulaires de systèmes de dégradation en assurant l’élimination rapide [
17]. Dans certains cas, l’agrégation résulte de la disponibilité limitée des chaperons et induit une réponse au stress dans la cellule qui produit des quantités non physiologiques d’une protéine inutile. Cependant, la formation des corps d’inclusion peut avoir un effet bénéfique en protégeant la protéine recombinante de la dégradation par les protéases cellulaires. (→) m/s 2005, n° 6-7, p.619) |
Renaturation des protéines in vitro
Bien qu’il n’existe pas d’approche universelle favorisant le repliement correct des protéines qui s’agrègent, plusieurs techniques permettant l’obtention d’une forme native sont couramment utilisées, avec des taux de succès très variés. Parmi elles, on distingue celles qui visent à renaturer la protéine in vitro à partir des corps d’inclusion purifiés [
18] et celles qui interviennent directement sur l’expression cellulaire afin de minimiser les réactions d’agrégation [
19]. La purification des corps d’inclusion est rarement une étape critique, dans la mesure où ces agrégats sont facilement isolés par centrifugation après lyse cellulaire et contiennent la protéine recombinante à un degré de pureté très élevé. L’étape suivante de solubilisation est en revanche souvent plus délicate, car elle nécessite la dénaturation chimique complète de la protéine agrégée par des dénaturants forts comme l’urée 8 M ou la guanidine 6 M, éventuellement en présence d’un agent réducteur. Enfin, intervient l’étape critique d’élimination du dénaturant qui, réalisée par dialyse ou forte dilution, favorise la renaturation de la protéine. À cette étape, certaines petites molécules peuvent être additionnées à la solution de renaturation, comme des détergents, des alcools, des amides, ou de l’arginine [
20]. En général, ces additifs, dont le mécanisme d’action est souvent peu documenté, diminuent l’agrégation des protéines en inhibant leurs interactions intermoléculaires. De même, les paramètres de température et de concentration de protéine sont contrôlés pour favoriser le repliement correct aux dépens de l’agrégation. Pour les protéines possédant une séquence polyhistidine, la chromatographie de chélation métallique constitue un troisième moyen permettant l’élimination du dénaturant tout en maintenant la protéine fixée [18]. Cette méthode permet une séparation physique des protéines lors de la renaturation sur la colonne, et limite donc les réactions d’agrégation. Les inconvénients de ces diverses techniques de renaturation in vitro sont souvent les faibles taux de renaturation obtenus, les difficultés d’optimisation des conditions expérimentales spécifiques de chaque protéine et la précipitation de la protéine renaturée. Pour ces raisons, il est souvent souhaitable d’intervenir en amont, sur le système d’expression ou sur le gène codant pour la protéine, afin de promouvoir le repliement cellulaire. |
Minimiser l’agrégation in vivo
Les méthodes d’optimisation du repliement productif des protéines recombinantes peuvent être schématiquement présentées en deux catégories. Dans la première, les facteurs cellulaires et les paramètres de culture qui influencent l’expression et le repliement des protéines sont systématiquement testés. Parmi ceux-ci, citons le contexte génétique de la cellule recombinante, la composition du milieu culture, la température de croissance des cellules, le nombre de copies du gène recombinant par cellule et son niveau de transcription ou de traduction, la surexpression des chaperons et l’inactivation des gènes codant pour les protéases. En règle générale, l’élévation de température augmente la vitesse d’agrégation, puisque cette réaction multimoléculaire est dominée par les interactions hydrophobes. En diminuant la température de croissance et le niveau de production, le repliement correct de la protéine sera donc favorisé. La variation de ces deux paramètres donne souvent des résultats encourageants. Cependant, la manipulation de ces facteurs extrinsèques n’est pas toujours suffisante, et l’on peut recourir à une stratégie qui modifie génétiquement la séquence de la protéine recombinante, par l’ingénierie des protéines. La première approche, utilisée le plus souvent pour faciliter leur purification, est la construction d’une protéine de fusion. Il est fréquemment observé qu’une protéine fusionnée à une protéine d’intérêt par son extrémité aminoterminale a une influence positive sur le repliement de cette dernière. Pour cette application, les systèmes de protéines de fusion les plus utilisées sont la protéine affine du maltose (MBP), la glutathion S-transférase ou encore NusA pour l’expression chez E. coli. Cette stratégie augmente la stabilité intracellulaire de la protéine recombinante et simplifie sa purification, qui est réalisée en une simple étape chromatographique sur une colonne d’affinité contenant le ligand immobilisé de la protéine de fusion. Cependant, la conservation de ces propriétés bénéfiques n’est pas toujours assurée après purification et clivage du partenaire de fusion. Une seconde approche est de rechercher, à l’aide des techniques d’évolution moléculaire dirigée1, des variants de la protéine d’intérêt qui ne s’agrègent plus. Les grands programmes internationaux de protéomique structurale, dans lesquels le nombre de protéines cibles étudié est important, ont considérablement contribué aux développements de cette approche qui requiert des débits de criblage élevés. Bien sûr, les sytèmes d’expression bactérien se prêtent plus facilement à ce type d’approche expérimentale. Le principe est relativement simple et repose sur une propriété fonctionnelle spécifique de la protéine de fusion utilisée, indépendante de la protéine cible. Dans ces approches, la protéine de fusion la plus documentée est la protéine à fluorescence verte (GFP), dont les propriétés de fluorescence sont acquises par sa structure native et peuvent être directement reliées à la solubilité intracellulaire des protéines [
21] (Figure 3). En effet, si une protéine d’intérêt fusionnée à la GFP s’agrège, elle entraînera dans les corps d’inclusion la GFP qui sera incapable de se replier : la cellule exprimant la fusion ne présentera pas cette fluorescence verte caractéristique. À l’inverse, si la protéine d’intérêt se replie correctement, la fluorescence spécifique de la GFP native sera émise par la cellule qui produit cette fusion. À partir d’une banque de mutants du gène cible, contenant des mutations ponctuelles, les variants fluorescents, donc solubles, seront ainsi isolés. Généralement, les substitutions d’acides aminés qui accélèrent le repliement vers la forme native de la protéine sont localisées en surface, et contribuent à une plus forte solubilité de la protéine sans toutefois modifier sa fonction. Très récemment, cette technique a été améliorée par l’introduction d’un système de deux fragments complémentaires dans la GFP qui élimine un grand nombre d’artéfacts inhérents à la technique originale [
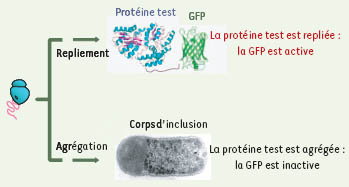
22].  | Figure 3.
Principe d’une fusion par la GFP (green fluorescent protein) pour tester le repliement cellulaire des protéines recombinantes. La fluorescence spécifique de la GFP est acquise uniquement à partir de sa structure tertiaire correctement repliée. Si le repliement d’une protéine test, fusionnée à la GFP, conduit à sa structure native, les bactéries produisant la fusion seront fluorescentes. Au contraire, si la protéine test s’agrège au cours du repliement, la formation des corps d’inclusion (visualisés par microscopie électronique) bloquera le repliement de la GFP, et les bactéries ne seront pas fluorescentes. |
|
Contrôle conformationnel des protéines La production d’une protéine ayant pour objectif d’obtenir celle-ci dans une conformation proche de son état natif, il est important de pouvoir contrôler le produit final. Ce contrôle final revêt un caractère essentiel dans les cas où la protéine recombinante est purifiée après renaturation des corps d’inclusion, ou dans le cas d’une application thérapeutique [
23], l’obtention d’une protéine soluble ne signifiant pas toujours qu’elle est dans un état natif. Un ensemble de techniques biochimiques comme l’électrophorèse et la chromatographie par gel filtration, ou biophysiques comme la spectrométrie de masse et le dichroïsme circulaire, sont conduites pour tester l’intégrité, l’homogénéité et la qualité des structures protéiques. Si la protéine est produite afin de déterminer sa structure tridimensionnelle, par cristallographie aux rayons X ou par résonance magnétique nucléaire, l’obtention de cette structure est une excellente validation du processus complet. Mais les tests d’activités biologiques qui dépendent de la protéine d’intérêt, et qui sont par nature très variés, représentent la meilleure sonde de la structure native. |
L’accroissement des connaissances aussi bien sur les principes gouvernant le repliement correct et incorrect des protéines que sur le développement d’outils sophistiqués pour modifier le patrimoine génétique des cellules ou l’automatisation des méthodes d’évolution moléculaire sont autant de voies suivies ouvrant les perspectives les plus encourageantes dans ce domaine. De même, la possibilité récente de modifier génétiquement la bactérie pour qu’elle réalise la glycosylation des protéines recombinantes [
24], qui était hier encore une utopie, est devenue un pari technologique accessible. |
Footnotes |
1. Pavlou AK, Reichert JM. Recombinant protein therapeutics: success rates, market trends and values to 2010. Nat Biotechnol 2004; 22 : 1513–9. 2. Braun P, LaBaer J. High throughput protein production for functional proteomics. Trends Biotechnol 2003; 21 : 383–8. 3. Gustafsson C, Govindarajan S, Minshull J. Codon bias and heterologous protein expression. Trends Biotechnol 2004; 22 : 346–53. 4. Brekke OH, Sandlie I. Therapeutic antibodies for human diseases at the dawn of the twenty-first century. Nat Rev Drug Discov 2003; 2 : 52–62. 5. Gerngross TU. Advances in the production of human therapeutic proteins in yeast and filamentous fungi. Nat Biotechnol 2004; 22 : 1409–14. 6. Wurm FM. Production of recombinant protein therapeutics in cultivated mammalian cells. Nat Biotechnol 2004; 22 : 1393–8. 7. Hellwig S, Drossard J, Twyman RM, Fischer R. Plant cell cultures for the production of recombinant proteins. Nat Biotechnol 2004; 22 : 1415–22. 8. Ritz D, Beckwith J. Roles of thiol-redox pathways in bacteria. Annu Rev Microbiol 2001; 55 : 21–48. 9. Wilkinson B, Gilbert HF. Protein disulfide isomerase. Biochem Biophys Acta 2004; 1699 : 35–44. 10. Spirin AS. High-throughput cell-free systems for synthesis of functionally active proteins. Trends Biotechnol 2004; 22. 11. Betton JM. Rapid translation system (RTS): a promising alternative for recombinant protein production. Curr Protein Pept Sci 2003; 4 : 73–80. 12. Braun P, Hu Y, Shen B, et al. Proteome-scale purification of human proteins from bacteria. Proc Natl Acad Sci USA 2002; 99 : 2654–9. 13. Kiefhaber T, Rudolph R, Kohler H-H, Buchner J. Protein aggregation in vitro and in vivo: a quantitative model of the kinetic competition between folding and aggregation. Biotechnology 1991; 9 : 825–9. 14. Fink AL. Protein aggregation: folding aggregates, inclusion bodies and amyloid. Fold Design 1998; 3 : R9–23. 15. Carrio MM, Villaverde A. Construction and deconstruction of bacterial inclusion bodies. J Biotechnol 2002; 96 : 3–12. 16. Fischer G, Tradler T, Zarnt T. The mode of action of peptidyl prolyl cis/trans isomerases in vivo: binding versus catalysis. FEBS Lett 1998; 426 : 17–20. 17. Wickner S, Maurizi MR, Gottesman S. Posttranslational quality control: folding, refolding, and degrading proteins. Science 1999; 286 : 1888–93. 18. Middelberg APJ. Preparative protein refolding. Trends Biotechnol 2002; 20 : 437–43. 19. Baneyx F, Mujacic M. Recombinant protein folding and misfolding in Escherichia coli. Nat Biotechnol 2004; 22 : 1399–408. 20. Clark ED, Schwartz E, Rudolph R. Inhibition of aggregation side reactions during in
vitro protein folding. Meth Enzymol 1999; 309 : 217–36. 21. Waldo GS, Standish BM, Berendzen J, Terwilliger TC. Rapid protein-folding assay using green fluorescent protein. Nat Biotechnol 1999; 17 : 691–5. 22. Cabantous S, Terwilliger TC, Waldo GS. Protein tagging and detection with engineered self-assembling fragments of green fluorescent protein. Nat Biotechnol 2005; 23 : 102–7. 23. Chirino AJ, Mire-Sluis A. Characterizing biological products and assessing comparability following manufacturing changes. Nat Biotechnol 2004; 22 : 1383–91. 24. Zhang Z, Gildersleeve J, Yang YY, et al. A new strategy for the synthesis of glycoproteins. Science 2004; 303 : 371–3. |