| |
| Med Sci (Paris). 2005 April; 21(4): 371–376. Published online 2005 April 15. doi: 10.1051/medsci/2005214371.L’épigénétique comme aspect de la postgénomique François Képès* Programme d’Épigénomique, Genopole ; ATelier de Génomique Cognitive (ATGC), CNRS UMR 8071/Genopole, 93, rue Henri-Rochefort, 91000 Évry, France |
Une cellule transmet à sa descendance, outre la séquence de ses gènes, les niveaux d’activité de beaucoup d’entre eux. De même que la séquence peut varier par mutation, le niveau d’activité génique peut changer par « épimutation », ou modification épigénétique, c’est-à-dire héritable et sans altération génétique concomitante. Une épimutation peut avoir des conséquences phénotypiques considérables, qui résistent éventuellement à la disparition des conditions externes qui l’ont provoquée. Par exemple, des épimutations sont responsables de la divergence entre un neurone et une cellule épithéliale provenant du même œuf et contenant le même génome. Cette différence phénotypique est considérable au regard de celle qui sépare les neurones de deux espèces animales aux génotypes dissemblables, soulignant en cela l’importance des épimutations. |
Une vision graduelle, du génome à l’épigénome La tradition oppose les visions génétique et épigénétique. Cette opposition ne cède pas dans l’histoire récente, où l’épigénétique s’identifie surtout en tant qu’opposition à l’approche réductionniste de la génétique [
1]. Dans sa dimension factuelle, cette opposition (Figure 1A) cesserait pourtant d’être absolue si l’on adaptait notre référentiel aux connaissances actuelles. Ce référentiel pourrait comprendre, selon une échelle chimique qui part de la séquence génétique stricto sensu (Figure 1B) :  | Figure 1. Génome et épigénome. A. Vision traditionnelle opposant le génétique à l’épigénétique. Les mutations génétiques affectent la séquence nucléotidique de l’ADN. Les autres modifications, ou épimutations, sont dites épigénétiques. B. Vision nouvelle et graduelle allant du génétique aux différentes facettes de l’épigénétique. Les découvertes récentes concernant la dynamique des phénomènes épigénétiques suggèrent un référentiel commun aux mutations et aux épimutations : ce référentiel met en œuvre des échelles corrélées d’ordres chimique, spatial et temporel. À l’extrême gauche se trouve les mutations génétiques. À l’extrême droite se situent les états régulatoires de la cellule, par exemple un état de différenciation, implémentés par des composants distribués dans tout ou partie de la cellule. Entre ces deux extrêmes se trouvent les méthylations de l’ADN, le recrutement au niveau de l’ADN des histones ou de protéines histone-like, ainsi que leurs éventuelles modifications post-traductionnelles, et les réarrangements structuraux à longue portée du chromosome. Du génome à l’épigénome, les excursions à partir du chromosome sont de plus en plus lointaines (échelle spatiale), les temps caractéristiques pour qu’une (épi-)mutation se produise de plus en plus longs et, à l’inverse, le temps moyen pour que, à la suite d’(épi-)mutations, un changement phénotypique soit fixé est de plus en plus court. |
-
des nucléotides A, T, G, C ;
-
des segments d’ADN où les cytosines sont méthylées ou non ;
-
des segments d’ADN associés ou non à des histones,
-
celles-ci étant méthylées, acétylées, phosphorylées, ADP-ribosylées, ubiquitinylées, ou non ;
-
des portions de chromosome subissant des réarrangements à longue portée de leur structure tridimensionnelle ;
-
des états régulatoires stables et héritables. Dans ce dernier cas, les acteurs moléculaires peuvent se trouver spatialement éloignés du chromosome. Par exemple, la régulation peut s’appuyer sur un « interrupteur » bistable fondé sur des interactions entre protéines cytoplasmiques, avec pour conséquence l’activation ou l’inactivation d’une protéine régulatrice.
En partant de la séquence stricto sensu, on s’élève donc, de (a) vers (f), au-dessus du génome, dans les niveaux d’organisation du vivant : du génome, on est graduellement passés à l’« épigénome » en investissant des composants répartis dans l’ensemble de la cellule. Cela signifie que l’échelle chimique décrite ci-dessus a une contrepartie spatiale :
-
les modifications affectent un nucléotide ;
-
les cytosines d’une courte région chromosomique sont concernées ;
-
une région chromosomique s’associe aux histones ;
-
qui pourraient se trouver modifiées dans une sous-région ;
-
les réarrangements concernent un chromosome entier, peut-être l’ensemble des chromosomes ;
-
les états cytoplasmiques entrent en jeu.
Cette échelle chimique et spatiale est corrélée à l’échelle du temps que requiert la modification. Mais, de manière plus intéressante, elle est approximativement anticorrélée à l’échelle des temps nécessaires pour que les modifications accumulées engendrent un changement observable (Figure 1B). Ainsi, l’échelle de temps la plus longue est associée au niveau le plus stable, celui des nucléotides et de leurs mutations (a) : la vaste majorité de ces mutations reste en effet sans conséquence phénotypique, et l’échelle de temps de fixation de mutations engendrant une conséquence phénotypique, évolutionnaire, se chiffre en milliers de générations. À l’autre extrémité, les états différenciés, obtenus structuralement (e) ou non (f), peuvent faire l’objet de bascules liées à un stimulus (réponse à un stress par exemple), à une échelle assez proche ou en dessous du temps de génération cellulaire : la différenciation tissulaire provient d’événements étalés sur un petit nombre de divisions cellulaires successives, les états différenciés occupant ainsi des échelles de l’ordre du temps de génération cellulaire. Entre ces deux extrêmes s’étagent les échelles de temps ontogéniques des modifications chimiques des cytosines de l’ADN et des histones formant le cœur des nucléosomes. Par exemple, la teneur globale en résidus cytosine méthylés diminue avec l’âge des cellules mammifères, en même temps qu’augmente la méthylation localisée au niveau des promoteurs géniques [
2,
3]. La lignée germinale peut également être altérée par ce type de modifications, entraînant des conséquences sur plusieurs générations organismiques. En somme, nos connaissances suggèrent désormais de remplacer l’opposition traditionnelle génétique/épigénétique (Figure 1A) par une vision graduelle reposant sur quatre échelles corrélées de changements, chimique, spatiale et temporelles (Figure 1B). Cette vision graduelle est alimentée par les liens multiples qu’entretiennent les effets épigénétiques d’une extrémité à l’autre de l’échelle. Les cytosines méthylées entraînent des mutations lors de la réplication, au point de façonner certaines séquences d’ADN ; réciproquement, une mutation impliquant l’arrivée ou le départ d’une cytosine change évidemment la capacité du site à être méthylé. Une méthylation de l’histone H3 sur sa lysine 9 (H3-K9) déclenche ou cible la méthylation de l’ADN [
4–
6], et réciproquement [
7,
8]. Chez l’arabette et la levure de fission, un lien a été établi entre les effets épigénétiques de certains petits ARN sur les états régulatoires de la cellule et les méthylations sur H3-K9 et l’ADN [
9,
10]. Enfin, durant la carcinogenèse, se produisent de manière corrélée des mutations génétiques, des changements de méthylation ou de structure de l’ADN et des dérèglements transcriptionnels [
11]. Après avoir plaidé pour une vision nuancée allant du génétique aux différentes facettes de l’épigénétique, l’objectif principal de cet article est de détailler plus avant les points (e) et (f), les autres modes épigénétiques étant largement traités dans d’autres articles de ce numéro. Pour (e) et (f), nos connaissances résident principalement dans le monde des micro-organismes, qui fournira donc la plupart des illustrations. |
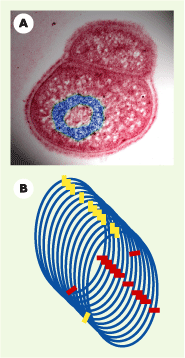
Réarrangements structuraux tridimensionnels du chromosome (e) Dans cette section sont traités les réarrangements à longue portée de la structure tridimensionnelle du chromosome, susceptibles d’influencer l’expression génique de manière héritable : il ne s’agit donc pas des remaniements monodimensionnels de la séquence apportés par les recombinaisons. Deux types de structures seront considérés, en quasi-équilibre et en non-équilibre. Cette dernière catégorie est définie par le fait que les structures chromosomiques dépendent totalement de la continuité des activités ayant l’ADN pour substrat, telles que la transcription. Les structures en quasi-équilibre ne dépendent en revanche pas du fonctionnement continu d’une activité liée à l’ADN et de la consommation continue d’énergie. Voici un exemple pour chacun de ces types de structure. L’archétype d’une structure chromosomique en quasi-équilibre est l’anneau extrêmement compact et latéralement ordonné. Les morphologies annulaires sont en général l’apanage de chromosomes situés dans des formes dormantes de vie telles que les spores, où elles représentent des états de quasi-équilibre du chromosome et peuvent subsister longtemps sans consommer d’énergie [
12]. Cependant, la bactérie Deinococcus radiodurans montre ce chromosome annulaire également durant sa croissance (Figure 2A), et cet arrangement particulier est responsable de son exceptionnelle radiorésistance [
13].  | Figure 2. Structures chromosomiques stables. A. Micrographie électronique par transmission d’une cellule de bactérie D. radiodurans, mettant en évidence l’arrangement annulaire de son chromosome (en bleu). Cette organisation extrêmement compacte permet aux fragments d’ADN produits par un traitement comme l’irradiation de maintenir leur ordre séquentiel et de rester proches l’un de l’autre, ce qui favorise la ligature de leurs extrémités libres. Cela représente le cas d’une structure chromosomique en quasi-équilibre (don généreux du Dr A. Minsky, The Weizmann Institute of Science, Israël). B. Arrangement solénoïdal de la fibre d’ADN. Les rectangles rouges représentent les gènes cibles d’un facteur de transcription, les rectangles jaunes ceux d’un autre facteur de transcription. Un tour de solénoïde représente un intervalle entre cibles du même facteur. Ainsi, chaque jeu de cibles est majoritairement regroupé dans l’espace cellulaire, mais séparé des autres jeux. Cet arrangement ne perdure que tant que la transcription est active : il s’agit d’une structure chromosomique en non-équilibre. |
L’héritabilité d’une structure chromosomique en non-équilibre est une hypothèse récente étayée par diverses observations. Pour satisfaire le critère d’héritabilité, il est nécessaire que l’activité structurante se poursuive durant la mitose ou la méïose eucaryote, ou la division bactérienne. C’est le cas notamment de l’activité transcriptionnelle, qui se poursuit durant tout le cycle cellulaire chez de nombreux micro-organismes. Cependant, cette héritabilité concerne aussi les cellules mammifères, où l’on a démontré que les chromosomes préservent leur position nucléaire globale de génération en génération, même si les mécanismes de cette préservation sont mal compris [
14]. L’importance structurante de la transcription a été mise en évidence par un faisceau d’observations morphologiques et de données d’analyse des transcriptomes. D’une part, la morphologie indique que, chez la bactérie comme chez les mammifères, l’activité transcriptionnelle n’opère pas de manière diffuse, mais se regroupe en points focaux [
15,
16]. Chez la bactérie, l’activité redevient diffuse lorsque l’intensité transcriptionnelle baisse, suggérant l’origine dynamique du phénomène [15]. D’autre part, des données récemment obtenues par l’étude des transcriptomes suggèrent qu’un tel point focal correspondrait à des gènes régulés par le même facteur de transcription : en effet, ces gènes cibles « co-régulés » tendent à être régulièrement espacés le long des chromosomes chez l’entérobactérie Escherichia coli comme chez la levure de bière Saccharomyces cerevisiae [
17–
19]. Si l’on observe les cibles de tout autre facteur de transcription, la même période est observée pour un chromosome donné. L’origine dynamique de ces surprenantes régularités transparaît dans le fait que cette période, constante pour un chromosome donné, diffère d’un chromosome à l’autre, fût-ce au sein d’un même organisme. Ce fait et quelques autres suggèrent donc que ces régularités reflètent la dynamique transcriptionnelle collective. À défaut, il faudrait admettre l’existence de molécules structurant le chromosome de manière périodique et figée mais, bizarrement, selon une période qui dépendrait du chromosome considéré. En somme, les périodicités observées sont compatibles avec une épi-organisation solénoïdale du chromosome, qui regrouperait dynamiquement en des points focaux les deux partenaires transcriptionnels, les facteurs de transcription et leurs sites de liaison sur l’ADN (Figure 2B). Ce regroupement optimiserait les régulations transcriptionnelles en augmentant les concentrations locales des deux partenaires, selon un processus bien démontré dans le cas intragénique (où les sites se trouvent dans la région régulatrice du même gène) [
20,
21], qui serait maintenant extrapolé à la situation intergénique. Ici, la dynamique transcriptionnelle est à la fois structurante et fonction de la structure. |
États régulatoires nucléo-cytoplasmiques stables (f) Il est ici question d’effets régulatoires qui verrouillent les cellules dans un schéma transcriptionnel relativement stable, héritable dans les cellules filles après division cellulaire. Les effecteurs sont des facteurs de transcription au sens large, soit des protéines, comme il a été discuté ci-dessus, soit de petits ARN régulatoires dits antisens, non codants, ou interférants ; en effet, certains petits ARN peuvent par exemple entraîner la formation d’hétérochromatine silencieuse sur le plan transcriptionnel. Les propriétés variées de ces états régulatoires peuvent être illustrés par trois exemples, avec comme fil directeur l’analyse logique généralisée [
22]. La première démonstration de l’existence de verrouillages épigénétiques date des années 1950 [
23,
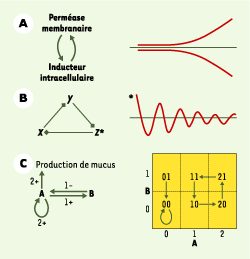
24]. L’opéron lactose d’E. coli est exprimé en présence d’un inducteur apparenté au lactose : on dit alors que les cellules sont induites. L’un des cistrons de l’opéron code pour la perméase à lactose, protéine membranaire qui pompe l’inducteur externe dans la cellule. Les expériences consistent, à partir d’une culture bactérienne non induite, à lui ajouter une forte dose d’inducteur, puis la fractionner en deux aliquotes, NI et I. Les deux cultures sont ensuite diluées du même facteur, de sorte que la concentration externe en inducteur suffirait à maintenir un état pré-induit, mais ne suffirait plus à induire des cellules non préalablement induites. NI est diluée immédiatement, tandis que I est diluée après 10 minutes : NI n’étant pas induite au moment de la dilution, elle reste non induite, tandis que I, ayant eu le temps d’être induite, le reste. Alors que NI et I partagent une histoire identique à un « détail » près, NI restera non induite et I induite pour au moins 150 générations. Ce phénomène s’explique aisément (Figure 3A, à gauche). L’inducteur est requis dans la cellule pour synthétiser la perméase, et l’inducteur, à cette concentration finale, nécessite la perméase pour entrer dans la cellule. Une cellule sans perméase ne pourra pomper d’inducteur et, en conséquence, ne produira pas de perméase (NI). Mais une cellule contenant au moins une perméase pompera de l’inducteur et produira donc plus de perméase (I). L’expérience a ainsi mis en évidence un interrupteur bistable fondé sur un circuit positif au sens de R. Thomas [22] - dans lequel tout composant s’autoactive via le circuit (Figure 3A, à droite). Cet exemple montre que l’information transmissible sur un mode épigénétique met en jeu des composants qui peuvent être situés aux confins de la cellule.
 | Figure 3. États régulatoires cellulaires. A. Le système bistable du lactose. L’opéron lactose d’E. coli code pour une perméase membranaire capable de pomper l’inducteur dans la cellule. Cet inducteur, apparenté au lactose, induit la production de perméase. À gauche, le graphe d’interactions est formalisé comme un circuit positif : la perméase accroît l’inducteur intracellulaire, qui augmente la perméase. À droite, cette stimulation réciproque résulte en une dynamique bistable, avec des trajectoires divergentes : si l’inducteur est en basse concentration, il va tendre à diminuer, et s’il est élevé, il va augmenter. B. Le répressilateur. À gauche, le graphe formalisé indique que trois gènes X, Y et Z s’inhibent l’un l’autre selon une permutation circulaire. C’est un circuit négatif susceptible de stabilisation, ou d’oscillation plus ou moins amortie. À droite, si l’un des gènes, par exemple Z, code pour un rapporteur fluorescent, il sera possible d’observer la trajectoire, ici une oscillation amortie. C. Réseau réglant la production de mucus chez P. aeruginosa. À gauche, le graphe formalisé comprend un activateur A et un inhibiteur B de la production de mucus, agissant dans deux circuits imbriqués, positif (autoactivation de A) et négatif (entre A et B). Ce graphe est l’un de ceux qui sont compatibles avec les connaissances disponibles sur la production de mucus. L’activateur A, ayant deux cibles (lui-même et B), connaît deux seuils et peut adopter au maximum trois valeurs significativement différentes parmi {0, 1, 2} : à valeur 0, A n’active aucune de ses cibles ; à 1, il en active une seule ; à 2, il active les deux. De son côté, l’inhibiteur B, ayant une seule cible, connaît un seuil unique et peut adopter au maximum deux valeurs parmi {0, 1}. Chaque flèche dans le graphe est marquée du numéro d’ordre de son seuil (1 ou 2) et du signe de son influence (-, inhibition ; +, activation). En connaissant les valeurs des paramètres (non indiqués ici), on peut en déduire une dynamique. Le graphe d’état asynchrone est représenté à droite. Les seuils entre les valeurs que prennent A et B sont indiqués par des pointillés, et les transitions par des flèches. Ce graphe montre deux états stables, soit A = 0, soit un cycle 11 ’ 10 ’ 20 ’ 21 ’ 11… |
Le cas du répressilateur [
25] est intéressant pour deux raisons. D’abord, c’est un circuit artificiel fonctionnant dans la cellule vivante, une forme de simulation in vivo. Ensuite, la stabilité transgénérationnelle réside ici dans la répétition quasi identique d’un cycle d’états, et non dans la perpétuation d’un état unique. Les auteurs ont implanté dans une souche bactérienne un circuit de régulation transcriptionnelle constitué de trois gènes s’inhibant l’un l’autre en permutation circulaire (Figure 3B, à gauche). Il s’agit donc d’un circuit négatif au sens de R. Thomas [22] - dans lequel tout composant s’auto-inhibe via le circuit - qui peut entraîner, selon le câblage et le paramétrage, soit une stabilisation, soit une oscillation (Figure 3B, à droite). Dans une grande proportion de cellules, les auteurs ont observé au microscope une oscillation stable, dont la période est supérieure au temps de génération, ce qui implique que les cellules filles héritent de l’état de l’oscillateur parental. La bactérie opportuniste Pseudomonas aeruginosa envahit les poumons des patients atteints de mucoviscidose. Plus tard, la bactérie devient capable de produire un mucus qui augmente sa résistance aux antibiotiques et la déficience respiratoire du patient. La question est de savoir si ce changement est le fruit d’une mutation ou d’une épimutation : la réponse à cette question conditionne en effet l’approche thérapeutique à privilégier. La Figure 3C (à gauche) illustre un des réseaux compatibles avec les données disponibles sur la production de mucus. L’activateur « A » s’autoactive (circuit positif) et active l’inhibiteur « B » qui inhibe « A » (circuit négatif) [
26] : les circuits positif et négatif sont donc imbriqués. Le graphe d’état associé à ce réseau (Figure 3C, à droite) indique les trajectoires définies en considérant tous les états possibles de A et B. Les méthodes formelles issues de l’informatique permettent de certifier les modèles (réseaux et graphes d’état associés) qui sont compatibles avec les interactions et les dynamiques observées, et de rejeter les autres [
27]. Cette approche facilite en outre la formulation des expériences à la paillasse ou en clinique qui seront les plus discriminatrices pour confirmer ou infirmer les modèles restant en lice, ici pour distinguer entre l’hypothèse de la mutation ou de l’épimutation [
28]. Ainsi s’institue une véritable boucle alliant les expérimentations par simulation et celles réalisées à la paillasse. |
Les riches modalités de l’épigénome, que cet article a rapidement parcourues, suggèrent de ne pas céder à la tentation d’attribuer a priori au génome une part majeure de l’information cellulaire, tentation visiblement alimentée par la relative facilité d’accès à sa séquence. Elles nous incitent à abandonner la naïveté du déterminisme purement génétique sans sombrer dans un indéterminisme démenti par les faits. Elles suggèrent de relever au contraire le défi d’un déterminisme épigénétique, plus difficile à appréhender, qui connecte en un réseau complexe les constituants de l’objet biologique étudié [
29]. Il est important que cette approche scientifique intègre les connaissances issues de l’analyse des niveaux plus élevés du vivant. C’est pourquoi ce projet pourrait être qualifié d’« épigénomique », un terme qui, par analogie avec l’« épigénétique », renvoie à la construction d’objets biologiques par niveaux croissants d’intégration, tout en se référant au génome et à l’épigénome plutôt qu’au gène et à l’épigène [
30,
31]. |
L’auteur remercie les membres de l’atelier génopolitain « Épigenèse » pour de passionnantes discussions, Avi Minsky pour la micrographie de la Figure 2A, Genopole® et le CNRS pour leur soutien.
|
1. Morange M. The relations between genetics and epigenetics : a historical point of view. Ann NY Acad Sci 2002; 981 : 50–60. 2. Wilson VL, Jones PA. DNA methylation decreases in aging but not in immortal cells. Science 1983; 220 : 1055–7. 3. Issa JP. CpG-island methylation in aging and cancer. Curr Top Microbiol Immunol 2000; 249 : 101–18. 4. Tamaru H, Selker EU. A histone H3 methyltransferase controls DNA methylation in Neurospora crassa. Nature 2001; 414 : 277–83. 5. Jackson JP, Lindroth AM, Cao X, Jacobsen SE. Control of CpNpG methylation by the KRYPTONITE histone H3 methyltransferase. Nature 2002; 416 : 556–60. 6. Lehnertz B, Ueda Y, Derijck AA, et al. Suv39h-mediated histone H3 lysine 9 methylation directs DNA methylation to major satellite repeats at pericentric heterochromatin. Curr Biol 2003; 13 : 1192–200. 7. Johnson L, Cao X, Jacobsen S. Interplay between two epigenetic marks. DNA methylation and histone H3 lysine 9 methylation. Curr Biol 2002; 12 : 1360–7. 8. Soppe WJ, Jasencakova Z, Houben A, et al. DNA methylation controls histone H3 lysine 9 methylation and heterochromatin assembly in Arabidopsis. EMBO J 2002; 21 : 6549–59. 9. Volpe TA, Kidner C, Hall IM, et al. Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi. Science 2002; 297 : 1833–7. 10. Zilberman D, Cao X, Jacobsen SE. ARGONAUTE4 control of locus-specific siRNA accumulation and DNA and histone methylation. Science 2003; 299 : 716–9. 11. Baylin SB, Esteller M, Rountree MR, et al. Aberrant patterns of DNA methylation, chromatin formation and gene expression in cancer. Hum Mol Genet 2001; 10 : 687–92. 12. Minsky A, Shimoni E, Frenkiel-Krispin D. Stress, order and survival. Nat Rev Mol Cell Biol 2002; 3 : 50–60. 13. Levin-Zaidman S, Englander J, Shimoni E, et al. Ringlike structure of the Deinococcus radiodurans genome : a key to radioresistance ? Science 2003; 299 : 254–6. 14. Gerlich D, Beaudouin J, Kalbfuss B, et al. Global chromosome positions are transmitted through mitosis in mammalian cells. Cell 2003; 112 : 751–64. 15. Lewis PJ, Thaker SD, Errington, J. Compartmentalization of transcription and translation in Bacillus subtilis. EMBO J 2000; 19 : 710–8. 16. Cook PR. Predicting three-dimensional genome structure from transcriptional activity. Nat Genet 2002; 32 : 347–52. 17. Képès F. Periodic epi-organization of the yeast genome revealed by the distribution of promoter sites. J Mol Biol 2003; 329 : 859–65. 18. Képès F. Periodic transcriptional organization of the E. coli genome. J Mol Biol 2004; 340 : 957–64. 19. Képès F, Vaillant C. Transcription-based solenoidal model of chromosomes. ComPlexUs 2003; 1 : 171–80. 20. Müller-Hill B. The function of auxiliary operators. Mol Microbiol 1998; 29 : 13–8. 21. Vilar JM, Leibler S. DNA looping and physical constraints on transcription regulation. J Mol Biol 2003; 331 : 981–9. 22. Thomas R, D’Ari R. Biological feedback. Boca Raton, FL : CRC Press, 1990. 23. Novick A, Wiener M. Enzyme induction is an all-or-none phenomenon. Proc Natl Acad Sci USA 1957; 43 : 553–6. 24. Cohn M, Horibata K. Analysis of the differentiation and of the heterogeneity within a population of Escherichia coli undergoing induced béta-galactosidase synthesis. J Bacteriol 1959; 78 : 613–23. 25. Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature 2000; 403 : 335–8. 26. Guespin-Michel J, Kaufman M. Positive feedback circuits and adaptive regulations in bacteria. Acta Biotheor 2001; 49 : 207–18. 27. Bernot G, Comet JP, Richard A, Guespin J. Application of formal methods to biological regulatory networks : extending Thomas’ asynchronous logical approach with temporal logic. J Theor Biol 2004; 229 : 339–47. 28. Guespin-Michel J, Bernot G, Comet JP, et al. Epigenesis and dynamic similarity in two regulatory networks in Pseudomonas aeruginosa. Acta Biotheor 2004; 52 : 379–90. 29. Atlan H. La fin du « tout génétique » ? Versailles : Éditions INRA, 1999. 30. Képès F. Le rêve du génomicien. Geninfo 1999; 2 : 3. 31. Dardel F, Képès F. Bioinformatique : génomique et post-génomique. Palaiseau : Presses de l’École Polytechnique, 2000, 2002 (2e ed) : 235. |