Parce que les globules rouges étaient facilement accessibles, les désordres génétiques de l’hémoglobine ont été souvent les modèles exemplaires de mutations, retrouvées ultérieurement dans d’autres gènes. On a ainsi décrit différents types de mutations au niveau des régions codantes ou de leur environnement immédiat, moins fréquemment des délétions impliquant des zones régulatrices majeures situées à distance des gènes qu’elles contrôlent. Quelques cas sont, cependant, toujours restés inexpliqués pour lesquels était formulée l’hypothèse d’un effet de position, l’expression d’un gène pouvant être profondément modifiée par son déplacement dans une configuration chromatinienne « défavorable » [ 1, 2]. L’existence de séquences modifiées en cis, potentiellement à l’origine de modifications épigénétiques (localisation nucléaire, temps de réplication, profil de méthylation de l’ADN…), et donc de l’inactivation d’un gène, n’avait jusqu’à présent pas été démontrée.
L’équipe d’hématologie moléculaire de D.R. Higgs (John Radcliffe Hospital, Oxford, Royaume-Uni) a présenté en 2000 un cas d’α-thalassémie qui ne s’expliquait pas complètement par les mécanismes connus [ 3]. Chez un sujet d’origine polonaise, les auteurs avaient identifié une délétion d’environ 18 kb, englobant, au niveau du locus α, le gène α1 (HBA1) et le gène θ (HBQ1), mais respectant le gène α2 (HBA2) et la zone régulatrice majeure située en amont, délétion qu’ils ont intitulée (α-ZF) d’après les initiales du patient (Figure 1). Le phénotype hématologique αα/αZF était cependant nettement plus sévère que ce qu’on attendait d’un sujet exprimant trois gènes α sur quatre. Le chromosome porteur de la délétion a été isolé et analysé en cellules lymphoblastoïdes après hybridation interspécifique avec un chromosome murin. On a alors constaté l’absence complète d’expression d’ARNm humain. Le gène HBA2 intact - dont les séquences régulatrices étaient présentes - n’était pas exprimé. La question posée était donc le mode par lequel la délétion inactivait ce deuxième gène α. On n’a pu mettre en évidence ni la délétion d’un élément de régulation positive, ni en aval un élément de régulation négative qui se trouverait juxtaposé au gène HBA2.
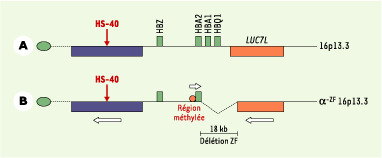
| Figure 1.
Région télomérique 16p où est localisé le locus alpha-globine. A. Chromosome normal. B. Chromosome ZF où a été observée la délétion. Le locus alpha, dont la transcription est orientée du télomère vers le centromère, comporte quatre gènes : HBZ, HBA2, HBA1, HBQ1. Il est situé dans une région dense, entre deux gènes dont la transcription est orientée du centromère vers le télomère. L’élément régulateur majeur du locus α, HS-40, est situé dans un intron d’un gène plus télomérique. Le gène LUC7L est situé du côté centromérique. Du fait de la délétion ZF, ce gène est tronqué, la transcription n’est pas interrompue par un signal polyA, et s’étend jusqu’au gène HBA2 et sa région promotrice. L’effet en est une méthylation des îlots CpG et une inactivation du gène. |
Formulant l’hypothèse d’un effet de position, les auteurs ont étudié différents aspects de la régulation chromatinienne. Ils ont constaté une méthylation des îlots CpG du gène éteint, méthylation qui ne diffusait pas au niveau des autres gènes du locus α. Dans cette même région promotrice du gène HBA2, on observait une sensibilité réduite aux endonucléases. L’exploration de la région centromérique située en aval de la délétion la montrait riche en séquences Alu, mais, surtout, identifiait la présence d’un gène fortement exprimé, le gène LUC7L. Ce gène, « hétérochromatique », pourrait coder pour une protéine de liaison à l’ARN [ 4]. Il est transcrit dans la direction opposée à celle du locusα. Les auteurs ont alors formulé l’hypothèse selon laquelle le transcrit de ce gène, anormalement étendu du fait de la délétion, pouvait être responsable de la méthylation de la région promotrice de HBA2 et de l’extinction subséquente du gène.
L’expression d’un transcrit antisens a été observée comme impliquée dans la méthylation des îlots CpG et l’extinction de l’expression génique au cours de l’empreinte parentale [ 5, 6]. C’est aussi un phénomène classique au cours de l’inactivation de l’X [ 7]. Il n’avait, cependant, jamais été décrit dans un locus autosomique non soumis à l’empreinte. La même équipe de D.R. Higgs a donc poussé l’exploration de sa première publication afin de confirmer le diagnostic [ 8]. En précisant les limites de la délétion, on constate qu’elle ampute l’extrémité 3’ du gène LUC7L, dont les trois derniers exons et la zone de polyadénylation sont délétés (Figure 1). Quand le chromosome α-ZF est exprimé en lymphocytes transformés par le virus EBV, l’analyse par RT-PCR montre que le transcrit de ce gène tronqué n’est pas normalement interrompu et s’étend jusqu’aux îlots CpG du gène HBA2. Les auteurs ont aussi reproduit chez la souris transgénique les données de la délétion humaine. Selon que le gène LUC7L tronqué s’exprime dans le sens opposé ou dans le même sens que le gène HBA2, on observe ou non, tôt dans le développement, une méthylation des îlots CpG et une disparition des sites HS. Le modèle a aussi été reproduit au cours de la différenciation de cellules embryonnaires ES. Deux observations ont finalement contribué à valider le rôle essentiel de l’ARNm antisens dans l’ensemble des phénomènes observés : (1) l’extinction de HBA2 obtenue soit par suppression de la zone régulatrice majeure HS-40, soit par mutation des séquences CCAAT ou TATA, ne s’accompagne pas de méthylation ; (2) le phénomène a été reproduit dans une construction remplaçant le fragment de LUC7L par un fragment équivalent d’ubiquitine (UBC) transcrit antisens. Il s’agit donc non d’un cas isolé, mais d’un mécanisme général qui a pu être reproduit dans des constructions relativement simples. Il démontre que l’inactivation d’un gène structurellement normal peut être le résultat d’une modification épigénétique dont le mécanisme est la transcription antisens d’un ARN. Le même mécanisme pourrait être retrouvé dans d’autres maladies génétiques, localisées surtout dans les régions où la densité des gènes est élevée, comme c’est le cas à l’extrémité télomérique 16p. Quel est également le rôle possible de ce mécanisme dans les dérèglements acquis ? Le transcrit d’un ARN aberrant (séquence LINE, SINE ou séquence rétrovirale) pourrait-il modifier l’expression d’un oncogène ou d’un gène suppresseur de tumeur ? Quels sont l’ordre et la hiérarchie des événements épigénétiques au cours du développement ?