| |
| Med Sci (Paris). 2004 October; 20(10): 894–898. Published online 2004 October 15. doi: 10.1051/medsci/20042010894.Les microARN Une nouvelle classe de régulateurs de l’expression génique Caroline Hartmann,1,2* Fabienne Corre-Menguy,1,2 Adnane Boualem,1,2 Mariana Jovanovic,1,2 and Christine Lelandais-Brière1,2 1Université Denis Diderot Paris VII, 2, place Jussieu, 75251 Paris Cedex 05, France 2Institut de Biotechnologie des Plantes, Bâtiment 630, Université Paris XI, 91405 Orsay, France |
Jusqu’à la découverte des ribozymes, les ARN (ARNr, ARNt, ARNm) étaient considérés uniquement comme des intermédiaires indispensables à la synthèse des protéines. Il est clair aujourd’hui que toutes les cellules possèdent des ARN qui assurent certaines fonctions directement sous la forme d’ARN. Ces dernières années, deux nouvelles classes de petits ARN non codants (ARNnc) de 21 à 25 nucléotides ont été caractérisées chez les métazoaires et chez les plantes : les ARN interférents (ARNsi, small interfering RNA) double brin et les microARN simple brin. Ces deux types d’ARNnc pourraient interagir avec leurs ARNm cibles selon deux modes différents pour provoquer leur dégradation, ou l’inhibition de leur traduction [
1,
2]. La biogenèse et l’activité biologique de ces ARNnc nécessitent la participation d’une ou de plusieurs ribonucléase(s) de type III, des protéines de la famille Argonaute et, pour certains ARNsi, des enzymes capables de synthétiser de l’ARN à partir d’une matrice ARN. Cependant, le rôle exact de chacune de ces familles de protéines dans le métabolisme et la fonction des ARNnc n’est pas encore complètement défini. Cet article fait le point sur l’état actuel des connaissances concernant les microARN identifiés à ce jour, aussi bien chez les animaux que chez les végétaux. |
La découverte des microARN Le microARN lin-4 : une particularité des nématodes Le premier microARN a été identifié en 1993, chez le nématode C. elegans, par une équipe qui tentait de décrypter les mécanismes moléculaires régissant le développement de cet organisme modèle [
3]. Le cycle de développement de ce petit ver comporte six stades successifs : l’embryogenèse, quatre stades larvaires (L1 à L4) et le stade adulte. Chez le mutant lin-4, qui réitère en permanence la phase larvaire L1, le phénotype sauvage peut être restauré par un ADN de 693 paires de bases dépourvu de phase ouverte de lecture. Ce fragment d’ADN comporte un gène codant pour un petit ARN de 22 nucléotides qui, à lui seul, est capable de restaurer le phénotype sauvage en se fixant, par hybridation moléculaire, sur plusieurs sites de la région 3’ non codante (3’UTR) des ARNm codant pour les protéines LIN-14 et LIN-28. Cette fixation provoque un arrêt de la synthèse de ces protéines [ 3]. La protéine LIN-14 est un répresseur de la transition du stade larvaire L1 vers le stade L2. Chez le nématode, à la fin du stade L1, le gène lin-4 est transcrit par une ARN polymérase de type II ou III [
4] en un pri-microARN d’environ 125 nucléotides qui est clivé par la protéine Drosha [
5] pour libérer un précurseur de 61 nucléotides qui forme une structure double brin en tige-boucle avec quelques mésappariements dans la région de la tige (Figure 1). Ce précurseur est ensuite activement exporté [
6] dans le cytoplasme où il est découpé par une protéine Dicer pour engendrer un microARN mature de 22 nucléotides [ 5]. La fixation du microARN lin-4 sur la région 3’UTR du messager codant pour la protéine LIN-14 inhibe la synthèse de cette protéine, ce qui permet la transition du stade larvaire L1 vers le stade L2.
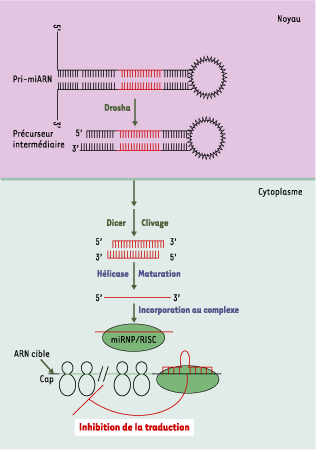
 | Figure 1.
Voie de biogenèse d’un microARN et mode d’action chez les métazoaires. Le pri-microARN double brin (Pri-miARN), transcrit à partir du gène, donne naissance à un ARN double brin (précurseur intermédiaire) par action d’une ribonucléase Drosha. Après une exportation active du précurseur dans le cytoplasme, une forme double brin du microARN est produite par l’action de la protéine Dicer. Une étape de maturation supplémentaire, à laquelle participe vraisemblablement une hélicase, est ensuite nécessaire avant que le microARN simple brin mature se fixe sur le complexe miRNP/RISC (microribonucleoprotein/RNA-induced silencing complex). Ce complexe guide ensuite le microARN vers la région 3’ non codante (3’UTR) de l’ARNm cible. Après hybridation imparfaite entre le microARN et la cible, la traduction de la protéine correspondante est inhibée. |
Après cette découverte surprenante, aucune homologie de séquence avec le gène lin-4 n’a été identifiée dans les bases de données disponibles à l’époque. Ce mode de régulation a été catalogué alors comme une bizarrerie spécifique du développement des nématodes. Le microARN let-7 : la découverte d’un nouveau mécanisme de régulation du développement En 2000, un nouveau microARN, let-7, a été identifié chez C. elegans ; il contrôle la transition entre le stade larvaire L4 et le stade adulte. Cette découverte a permis de généraliser le mode d’action de lin-4. En effet, le gène let-7 code pour un ARN de 21 nucléotides qui inhibe l’accumulation des protéines LIN-41 et LIN-42 en se fixant sur la région 3’UTR des ARNm correspondants [
7]. Des séquences homologues du gène let-7 ont été identifiées chez toutes les espèces d’invertébrés et de vertébrés à symétrie bilatérale examinées. De plus, quelle que soit l’espèce considérée, l’expression de let -7 est contrôlée au cours du développement [
8]. Cette conservation de séquence et de mode de régulation au sein d’espèces très éloignées suppose une forte pression de sélection sur ce gène. L’identification de gènes homologues de let-7 est à l’origine d’une recherche systématique de microARN chez les métazoaires et chez les plantes. |
Les microARN chez les métazoaires Les microARN : des molécules ubiquitaires En octobre 2001, la revue Science publiait les premiers résultats de cette recherche systématique. Chez la mouche, l’homme et deux espèces de nématodes, une centaine d’ARNnc ont été identifiés [
9–
11] par deux stratégies différentes : une approche expérimentale et une approche in silico. À cette époque, l’approche expérimentale s’était révélée la plus efficace [ 11]. Cependant, en 2003, deux logiciels spécifiquement conçus pour la recherche de gènes de microARN ont permis d’estimer le nombre de ces gènes chez les espèces modèles. Dans tous les cas, le nombre de gènes de microARN correspond à environ 1 % des gènes prédits. Chez une espèce donnée, il existe autant de gènes de microARN que de familles de gènes codant pour des facteurs de transcription qui sont également connus pour jouer un rôle primordial dans la régulation de l’expression génique [
12,
13]. Il faut noter que plus du tiers des gènes de microARN identifiés chez C. elegans possèdent des homologues chez l’homme [
14]. Les microARN identifiés présentent des caractéristiques communes. Tout d’abord, dans la grande majorité des cas, seul le microARN est retrouvé dans les banques d’ADNc, la partie complémentaire du précurseur (appelée miR*) étant sans doute rapidement dégradée après l’étape de maturation. Les gènes codant pour ces ARN sont soit dispersés, soit regroupés dans des régions intergéniques ou, plus rarement, présents dans des introns de gènes codant pour des protéines. Lorsqu’ils sont regroupés au même locus, ces gènes peuvent être transcrits sous la forme d’un polycistron [
15]. Par contre, les gènes dispersés correspondent à des unités de transcription indépendantes. Chez C. elegans, le taux d’accumulation de différents microARN a été déterminé : il peut varier de 1000 à 50 000 molécules par cellule [14]. À l’heure actuelle, aucun facteur intervenant dans la régulation de la transcription de cette catégorie de gènes ou dans la stabilité des molécules d’ARN elles-mêmes n’a été identifié. Les microARN : des acteurs du développement et de la différenciation cellulaire Dans la grande majorité des cas, les analyses transcriptionnelles ont révélé la présence simultanée du précurseur double brin et du microARN mature simple brin, comme précédemment décrit pour lin-4 et let-7. Plusieurs microARN présentent une modification de leur taux d’accumulation en fonction du stade de développement examiné et/ou de l’organe considéré, arguments en faveur de leur implication dans les mécanismes moléculaires régissant le développement et la différenciation cellulaire [ 9]. Au cours de l’année 2003, plusieurs démonstrations pertinentes de l’implication de ce type d’ARN dans la différenciation et la mort cellulaire ont été publiées [
16–
18]. Dans sa dernière revue [ 4], David Bartel propose, au vu des profils d’expression observés et du nombre de gènes de microARN caractérisés en 2004 chez les métazoaires, que chaque type tissulaire à un stade de développement particulier pourrait avoir un pool spécifique de microARN qui réglerait le transcriptome. Les cibles de ces microARN et les partenaires éventuellement engagés dans le contrôle de la traduction des protéines correspondantes restent cependant à identifier. |
Les microARN chez les plantes Ce mécanisme de régulation génique a-t-il été mis en place avant ou après la séparation entre la lignée verte et les métazoaires ? Chez la plante modèle Arabidopsis thaliana, 150 ARNnc ont été clonés en 2002 et plus de 2000 en 2004 [
19–
22] (voir également le site internet de J.C. Carrington : http://cgrb.orst.edu/smallRNA/db/). Quatre critères permettent de discriminer les ARNsi des microARN [
23]. Selon ces critères, seulement 20 microARN, correspondant à 15 familles différentes, ont été répertoriés chez A. thaliana. Chez cette espèce, certains gènes de microARN sont répétés dans le génome. Il existe souvent, pour un microARN donné, plusieurs gènes paralogues présentant une différence de un à deux nucléotides [
24]. Tout comme chez les animaux, plusieurs gènes de microARN sont conservés entre A. thaliana, une espèce dicotylédone, et des monocotylédones telles que le riz ou le maïs. De plus, les gènes codant pour ces microARN sont également présents soit dans des régions introniques soit dispersés ou regroupés dans des régions intergéniques où ils sont vraisemblablement transcrits à partir de leur propre promoteur. La présence de microARN chez les plantes suggère que le mode de régulation génique auquel ils participent préexistait à la séparation des deux règnes, bien qu’aucun microARN commun aux animaux et aux végétaux n’ait encore été identifié. Chez A. thaliana, certaines étapes du métabolisme des microARN sont différentes de celles décrites chez les animaux (Figure 2). Aucun pri-microARN n’a été identifié et les précurseurs doubles brins, qui sont généralement plus longs (60 à 300 nucléotides) et présentent des structures secondaires plus complexes, sont indétectables dans les expériences de type Northern [4]. La protéine DCL1, une des quatre protéines d’A. thaliana homologues de la protéine Dicer découverte chez les animaux, est localisée dans le noyau ; elle assure vraisemblablement le clivage des précurseurs des microARN [
25]. Trois autres protéines, HEN1, HYL1 et AGO1 sont également indispensables à l’accumulation des microARN [
21,
26–
28]. Comme DCL1, HEN1 et HYL1 comportent des signaux de rétention nucléaire ; ces deux protéines pourraient participer au contrôle de la taille des microARN. En revanche, la protéine AGO1 fait vraisemblablement partie du complexe RISC (RNA-induced silencing complex). Enfin, AGO1 et HEN1 interviennent à la fois dans les voies de synthèse des microARN et des ARN interférents [26], tandis que HYL1 semble spécifique des microARN [
27].
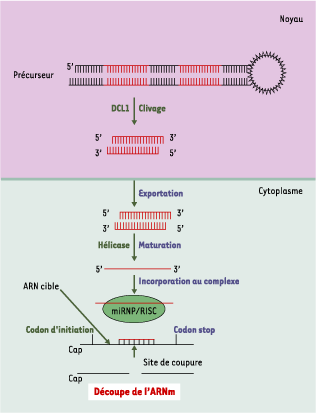
 | Figure 2.
Voie de biogenèse d’un microARN et mode d’action chez les plantes. Aucun pri-microARN n’a été jusqu’alors identifié. Un précurseur, généralement plus long que celui observé chez les animaux (60 à 300 nucléotides), est découpé dans le noyau par DCL1 (homologue de la protéine Dicer). La forme double brin du microARN est ensuite activement exportée dans le cytoplasme où elle subit une nouvelle étape de maturation qui fait également intervenir une hélicase. La forme simple brin mature est ensuite chargée sur le complexe miRNP/RISC. La fixation par hybridation presque parfaite du microARN sur la séquence codante de l’ARNm cible est responsable du clivage de l’ARNm cible et de l’arrêt de la synthèse de la protéine correspondante. |
|
Aussi bien chez les plantes que chez les animaux, un microARN ne pourra interagir avec sa cible qu’après son intégration dans un complexe nucléo-protéique appelé miRNP (micro-ribonucleoprotein) qui pourrait être identique au complexe RISC (RNA-induced silencing complex) qui intervient dans le mécanisme d’interférence de l’ARN [
29]. Chez les métazoaires Chez les animaux, dans la grande majorité des cas, l’hybridation imparfaite des microARN sur plusieurs sites de la région 3’UTR d’ARN messagers spécifiques inhibe la traduction des protéines correspondantes [ 3, 7]. Un seul microARN, miR 196 chez la souris, présente un appariement presque complet (21 sur 22 nucléotides) avec la région 3’UTR des ARNm HOXB8. Cette interaction provoque le clivage de l’ARNm cible, ce qui réprime la synthèse de la protéine correspondante [
30]. Aucune autre homologie complète entre microARN et ARNm n’a été identifiée chez les métazoaires. Avant la mise au point, très récente, de deux logiciels performants pour la recherche de cibles [
31,
32], les appariements imparfaits entre les séquences des microARN et des ARNm cibles rendaient l’identification de celles–ci très délicate. Chez les métazoaires, les analyses bio-informatiques ont révélé que les résidus deux à huit de la région 5’ des microARN étaient très conservés entre microARN homologues. Cette région jouerait donc un rôle prépondérant dans la reconnaissance de la cible. Plus de 400 ARNm cibles ont été identifiés chez les mammifères. La fiabilité de prédiction de l’un de ces logiciels a été testée expérimentalement chez des cellules HeLa exprimant de manière endogène les microARN ; dans ce travail, une inhibition de l’expression du gène rapporteur luciférase a été mise en évidence pour 11 des 15 cibles examinées [ 32]. Chez les plantes Chez les plantes, on observe une particularité jamais rapportée pour les animaux : l’existence d’un site unique de fixation dans la région codante de l’ARNm cible, sur lequel le microARN peut s’hybrider de façon presque parfaite (Figure 2). Le faible nombre de mésappariements entre le microARN et sa cible est responsable du mécanisme de répression mis en œuvre : la coupure de l’ARNm au milieu du site d’hybridation du microARN [
33] (Figure 2). Dans les faits, la grande majorité des microARN de plantes fonctionnent comme des ARN interférents [
34]. Toutefois, très récemment, il a été montré, chez A. thaliana, que le miR172 réprimait directement la traduction de la protéine APETALA2, comme cela est généralement le cas chez les animaux [
35,
36]. La grande majorité des microARN, chez les plantes, présentent une forte homologie de séquence avec leurs cibles. Cette particularité a permis, dès 2002, chez A. thaliana, d’identifier 49 cibles potentielles, soit au moins une cible pour chaque microARN [
37]. La plupart d’entre elles ont été validées expérimentalement par la suite (pour revue, voir [
38]). Les microARN identifiés chez les plantes ciblent préférentiellement des facteurs de transcription qui jouent un rôle prépondérant dans le développement ou la différenciation cellulaire, bien que les facteurs de transcription ne représentent que 6 % des séquences annotées chez A. thaliana [
39]. Les microARN pourraient donc constituer un mécanisme privilégié de contrôle de l’expression de cette catégorie particulière de protéines chez les plantes. Le rôle des microARN dans le développement et la différenciation est conforté par l’observation des mutants chez lesquels des protéines impliquées dans le métabolisme des microARN sont touchées (AGO1, DCL1, HEN1 et HYL1) ; ces mutants présentent en effet de fortes modifications développementales [ 26– 28]. De plus, l’expression de cibles (TCP2, TCP4, MYB33) portant des mutations silencieuses résistantes au clivage par des microARN se traduit par des anomalies importantes du développement [ 28, 36,
40]. |
Au cours des trois dernières années, des progrès immenses ont été faits dans la découverte des petits ARNnc dont les ARNsi et les microARN ne représentent vraisemblablement que la partie émergée de l’iceberg. Cette catégorie particulière d’ARN, les ribo-régulateurs, est capable d’éteindre l’activité d’un gène en agissant directement sur l’ARNm ou le gène. Ces ribo-régulateurs exercent systématiquement une régulation négative soit en découpant l’ARNm cible soit en se fixant sur la région 3’ UTR de l’ARNm et en bloquant la traduction. Très récemment, il a été montré que ces deux mécanismes d’action co-existent dans les règnes animal et végétal. |
Les auteurs tiennent à remercier Hervé Vaucheret et André Rode pour la relecture attentive du manuscrit.
|
1. Banerjee D, Slack F. Control of developmental timing by small tenporal RNAs : a paradigm for RNA-mediated regulation of gene expression. BioEssays 2002; 24 : 119–29. 2. Vaucheret H, Beclin C, Fagard M, et al. Post-transcriptional gene silencing in plants. J Cell Sc 2001; 114 : 3083–91. 3. Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochromic gene lin-4 encodes small RNA with antisens complementary to lin-14. Cell 1993; 75 : 843–54. 4. Bartel DP. MicroRNAs : genomics, biogenesis, mechanism, and function. Cell 2004; 116 : 281–97. 5. Lee Y, Anh C, Han J, et al. The nuclear RNase III Drosha initiates microRNA processing. Nature 2003; 425 : 415–9. 6. Lund E, Güttinger S, Calado A, et al. Nuclear export of microARN precursors. Science 2004; 303 : 95–8. 7. Reinhart BJ, Slack FJ, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature 2000; 403 : 901–6. 8. Pasquinelli AE, Reinhart BJ, Slack FJ, et al. Conservation of the sequence and temporal expression of let-7 heterochromic regulatory RNA. Nature 2000; 408 : 86–9. 9. Lagos-Quintina M, Rauhut R, Lendeckel W, Tuschl T. Identification of novel genes coding for small expressed RNAs. Science 2001; 294 : 853–8. 10. Lau NC, Lim LP, Weinstein G, Bartel DP. An abundant class of tiny RNA with probable regulatory roles in Caenorhabditis elegans. Science 2001; 294 : 858–62. 11. Lee RC, Ambros V. An extensive class of small RNA in Caenorhabditis elegans. Science 2001; 294 : 862–4. 12. Lai EC, Tomoncak P, Williams RW, Rubin GM. Computational identification of Drosophila microRNA genes. Genome Biol. 2003; 42 : 1–20. 13. Lim LP, Lau NC, Weinstein EG, et al. The microRNAs of Caenorhabditis elegans. Genes Dev 2003; 17 : 1–18. 14. Lim LP, Glasner ME, Yekta S, et al. Vertebrate microRNA genes. Science 2003; 299 : 1540. 15. Lee Y, Jeon K, Lee JT, et al. MicroRNA maturation : stepwise processing and subcellular localization. EMBO J 2002; 21 : 4663–70. 16. Brennecke J, Hipfner DR, Stack A, et al. Bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila. Cell 2003; 113 : 25–36. 17. Xu P, Vernooy SY, Guo M, Hay BA. The Drosophila microRNA Mir-14 supresses cell death and is required for normal fat metabolism. Curr Biol 2003; 13 : 790–5. 18. Kim J, Krichevsky A, Grad Y, et al. Identification of many microRNAs that copurify with polyribosomes in mammalian neurons. Proc Natl Acad Sci USA 2004; 101 : 360–5. 19. Reinhart BJ, Weinstein EG, Rhoades MW, et al. MicroRNAs in plants. Genes Dev 2002; 16 : 1616–26. 20. Llave C, Kasschau KD, Rector MA, Carrington JC. Endogenous and silencing-associated small RNAs in plants. Plant Cell 2002; 14 : 1605–19. 21. Park W, Li J, Song R, et al. CARPEL FACTORY, a dicer homolog, and HEN1, a novel protein, act in microRNA metabolism in Arabidopsis thaliana. Curr Biol 2002; 12 : 1484–95. 22. Xie Z, Johanen LK, Gustafson AM, et al. Genetic and functional diversification of small RNA pathways in plants. PloS Biol. 2004; 2 : 1–11. 23. Ambros V, Bartel B, Bartel DP, et al. A uniform system for microRNA annotation. RNA 2003; 9 : 277–9. 24. Wang JF, Zhou H, Chen YQ, et al. Identification of 20 microRNA from Oryza sativa. Nucleic Acids Res 2004; 32 : 1688–95. 25. Schauer SE, Jacobsen SE, Meinke DW, Ray A. DICER-LIKE1 : blind men and elephants in Arabidopsis development. Trends Plant Sci 2002; 7 : 487–91. 26. Boutet S, Vazquez F, Liu J, et al. Arabidopsis HEN1 : a genetic link between endogenous miRNA controlling development and siRNA controlling silencing and virus resistance. Curr Biol 2003; 13 : 843–8. 27. Vazquez F, Gasciolli V, Crété P, Vaucheret H. The nuclear dsRNA binding protein HYL1 is required for microRNA accumulation and plant development but not posttranscriptional transgene silencing. Curr Biol 2004; 14 : 346–51. 28. Vaucheret H, Vazquez F, Crete P, Bartel DP. The action of ARGONAUTE1 in the miRNA pathway and its regulation by the miRNA pathway are crucial for plant development. Genes Dev 2004; 18 : 1187–97. 29. Hutvagner G, Zamore PD. A microARN in a multiple turnover RNAi enzyme complex. Science 2002; 297 : 2056–60. 30. Yekta S, Shih IH, Bartel DP. MicroRNA-directed cleavage of HOXB8. Science 2004; 304 : 594–6. 31. Stark A, Brennecke J, Rusell RB, Cohen SM. Identification of Drosophila microRNA targets. PloS Biol 2003; 1 : 1–12. 32. Lewis BP, Shih IU, Jones-Rhoades MW, et al. Prediction of mammalian microRNA targets. Cell 2003; 115 : 787–98. 33. Llave C, Xie Z, Kasschau KD, Carrington JC. Cleavage of Scarecrow-like mRNA targets directed by a class of Arabidopsis miRNA. Science 2002; 297 : 2053–6. 34. Plasterk RHA. RNA silencing : Genome’s immune system. Science 2002; 296 : 1263–5. 35. Aukerman MJ, Sakai H. Regulation of flowering time and floral organ identity by a microRNA and its APETALA2-like target genes. Plant Cell 2003; 11 : 2730–41. 36. Chen X. A microRNA as a translational repressor of APETALA2 in Arabidopsis flower development. Science 2004; 303 : 2022–5. 37. Rhoades MW, Reinhart BJ, Lim LP, et al. Prediction of plant microRNA targets. Cell 2002; 110 : 513–20. 38. Mallory AC, Vaucheret H. MicroRNAs : something important between the genes. Curr Op in Plant Biol 2004; 7 : 1–6. 39. Riechmann JL, Heard J, Martin G, et al. Arabidopsis transcription factor ; genome-wide comparative analysis among eukaryotes. Science 2000; 290 : 2105–10. 40. Palatnik JF, Allen E, Wu X, et al. Control of leaf morphogenesis by microRNAs. Nature 2003; 425 : 257–63. |