| |
| Med Sci (Paris). 2004 October; 20(10): 876–881. Published online 2004 October 15. doi: 10.1051/medsci/20042010876.La capture d’enveloppe par les rétrovirus Felix J. Kim,1,2* Nicolas Manel,1 Jean-Luc Battini,1 and Marc Sitbon1* 1Institut de Génétique moléculaire de Montpellier (IGMM), CNRS-UMR5535, IFR122 et Université de Montpellier II, 1919, route de Mende, 34293 Montpellier Cedex 05, France 2Neuropharmacology, Sloan Kettering Institute, Memorial Sloan-Kettering Cancer Center, 1275 York Avenue, New York, NY 10021, États-Unis |
Rétrotransposons et rétrovirus Rétro-éléments et génomes La transposition des rétro-éléments [
1] nécessite la transcription inverse d’un ARN en ADN, qui s’intègre alors dans le génome cellulaire. Ces rétrotransposons sont abondants et participent directement au phénotype de l’hôte [
2] : ils ont ainsi accompagné la spéciation de l’homme, chez lequel les séquences rétrovirales endogènes (HERV) représentent 5 % à 8 % du génome [
3,
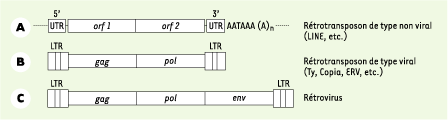
4]. Comme les rétrovirus, les rétro-éléments de type viral portent des séquences gag-pol encadrées de longues séquences répétées (LTR, long terminal repeat) (Figure 1). En revanche, ces rétro-éléments ne codent généralement pas pour des glycoprotéines virales d’enveloppe (Env) et ne sont habituellement pas capables de « dissémination » extracellulaire.
 | Figure 1.
Principaux rétrotransposons. A. Rétrotransposon de type non viral, tel que les LINE ( long interspersed nuclear element), dépourvu de LTR ( long terminal repeat) et contenant deux cadres de lecture ouverts ( ORF1 et ORF2, open reading frame 1 and 2). L’ORF 1 code pour des protéines de type Gag, tandis que l’ORF 2 porte les séquences codant pour les protéase, transcriptase inverse, RNAse H et intégrase caractéristiques des rétrovirus. B. Rétrotransposon de type viral délimité par des LTR, mais dépourvu du gène env, tel que les rétrotransposons Ty de levure, Copia de drosophile et certaines séquences endogènes rétrovirales (ERV). C. Rétrovirus infectieux, de type exogène ou endogène, arborant les trois cadres de lecture gag, pol et env [
5]. |
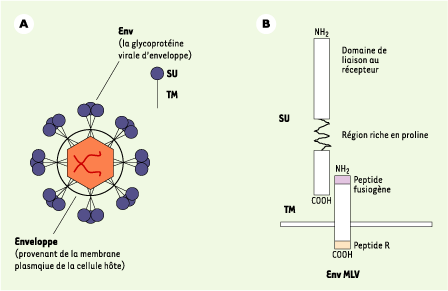
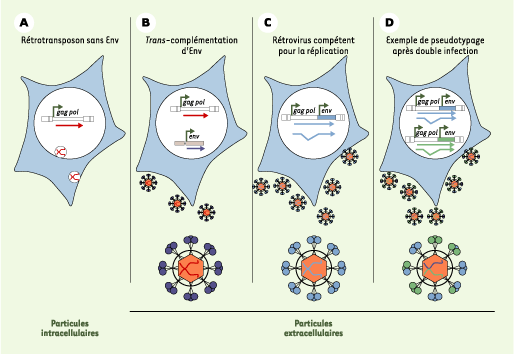
Les rétrovirus, des rétrotransposons bien enveloppés ? L’enveloppe rétrovirale arrachée aux membranes cellulaires lors du bourgeonnement de la particule porte la glycoprotéine virale Env. L’adjonction d’Env permet à la particule de disséminer à l’extérieur de la cellule en apportant deux fonctions principales : la liaison à un récepteur cellulaire par sa composante de surface (SU) et la fusion aux membranes cellulaires par sa composante transmembranaire TM (Figure 2). Si des particules rétrovirales extracellulaires peuvent être produites abondamment en l’absence de la protéine Env, par surexpression des protéines Gag et Pol, les particules endogènes naturelles dépourvues d’Env ne sont généralement pas relarguées à l’extérieur en l’absence de cette surexpression artificielle, mais s’accumulent à l’intérieur de la cellule (Figure 3A) (voir aussi [ 5], chapitre Assembly and maturation). En revanche, l’ajout d’Env favorise l’assemblage de virions à la membrane plasmique et polarise la production virale à l’extérieur de cellules épithéliales [
6,
7] (Figure 3B). Il a d’ailleurs été montré que la protéine Env déroute l’ARN et les virions de la voie lysosomiale vers la voie endosomique d’export extracellulaire [
8]. Ces propriétés suggèrent que l’émergence de rétrovirus à partir de rétrotransposons aurait bénéficié du routage extracellulaire des particules par Env (Figure 3A) et de l’effet protecteur d’une enveloppe dérivée de la membrane cellulaire.
 | Figure 2.
Organisation de l’enveloppe rétrovirale. A. Trimères de molécules d’Env à la surface d’un virion et monomère isolé, avec ses deux composantes de surface SU et transmembranaire TM. B. Organisation en modules de la protéine Env prototype des MLV de souris et de celle, apparentée, du rétrovirus humain HTLV. Une molécule d’Env comprend deux sous-unités issues du clivage d’une seule protéine précurseur : la sous-unité de surface (SU), entièrement extracellulaire, et la sous-unité d’ancrage transmembranaire (TM) ; sont représentés le domaine de liaison au récepteur et la région riche en prolines de la sous-unité SU, ainsi que le peptide fusogène aminoterminal et le peptide R carboxyterminal de la sous-unité TM impliqués dans la fusion membranaire [
14,
20,
21]. |
 | Figure 3.
Voies de production de particules rétrotransposables. A. Rétrotransposons dénués du gène Env, et donc de la glycoprotéine d’enveloppe Env : les particules bourgeonnent à l’intérieur de la cellule [ 5]. B. Pseudotypage d’Env par complémentation en trans : cette complémentation par une Env hétérologue permet aux particules en formation d’acquérir une protéine Env et, ainsi, de bourgeonner à l’extérieur de la cellule ; sont représentés les gènes gag et pol du rétrotransposon et un gène env hétérologue. C. Complémentation d’Env en cis dans le cas d’un rétrovirus entier : sont représentés le provirus entier et les ARN dérivés génomique entier et épissé env. D. Coproduction d’Env hétérologues : cas de deux rétrovirus produisant des pseudotypes d’Env et un génome rétroviral hétérodimère. Une variante de ce schéma est qu’une des deux Env provient d’un rétrovirus endogène défectueux ; ce pseudotypage étend le spectre d’infection de la particule. |
Sur la base de ces avantages sélectifs, la « capture d’enveloppe » par un rétrotransposon récapitulerait l’évolution d’un effet sporadique de trans-complémentation (Figure 3B) en une cis-complémentation constitutive (Figure 3C) donnant au rétrovirus ainsi constitué de nouvelles capacités infectieuses, tropiques et, parfois, pathogènes. L’Env capturée par un rétrovirus reste susceptible d’être utilisée en trans par d’autres rétrovirus ou par des rétro-éléments défectueux (pseudotypage1) (Figure 3D) [
9]. |
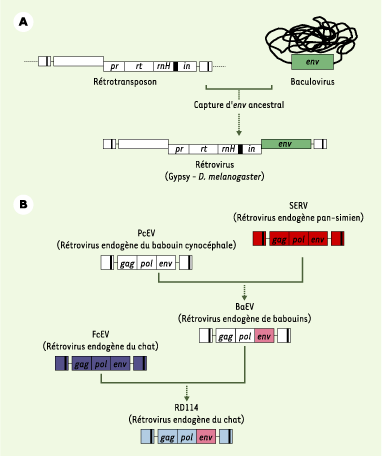
Rétrotransposons et capture de gènes env Rétrotransposons d’invertébrés aux env venues d’ailleurs De récents travaux ont fourni des indices précis sur la « capture d’enveloppe » par les rétro-éléments de type viral d’invertébrés. Ainsi, les rétrovirus du groupe Gypsy de Drosophila melanogaster, ainsi que d’autres rétrotransposons apparentés (errantivirus), sont infectieux [
10] du fait de la présence du gène ld130 codant pour une protéine Env analogue à la protéine F des baculovirus (virus d’insectes à grand génome ADN double brin) [
11,
12] ; de même, l’Env des rétrovirus Cer du nématode Caenorrhabditis elegans est apparentée à une protéine des phlébovirus (virus à ARN simple brin) ; enfin, le rétrovirus Tas du ver Ascaris lumricoides semblerait avoir capturé une Env apparentée à la glycoprotéine gB d’un herpès virus (virus à ADN double brin) [ 11]. Des indications supplémentaires sur un mécanisme éventuel de capture sont fournies par le rétrotransposon TED du lépidoptère Trichoplusiani, dont le gène env est également apparenté au gène ld130 du baculovirus. Des formes intégrées de TED ont été retrouvées dans le génome du baculovirus AcNV (Autographa californica nucleopolyhedrosis virus). La capture d’un gène env par le rétro-élément ancêtre de Gypsy et de TED a donc pu se produire dans une cellule par recombinaison d’un rétrotransposon (moins de 10 kb) intégré dans l’ADN d’un baculovirus (plus de 100 kb) [12]. L’infection par un virus exogène ADN à grand génome, baculovirus ou herpès virus, pourrait donc, sporadiquement, fournir les conditions favorables à la capture d’env par un rétro-élément et à l’émergence de nouveaux rétrovirus chez les invertébrés. Captures d’env chez les rétrotransposons de vertébrés Chez les vertébrés, l’acquisition de l’oncogène src par le virus aviaire du sarcome de Rous a été la première description de capture d’un gène hétérologue par un rétrovirus. L’origine des Env des rétrovirus de vertébrés reste cependant inconnue et l’acquisition de novo d’un gène env n’est pas documentée. Seule la « capture » de séquences env par recombinaison avec d’autres séquences env rétrovirales hétérologues a été décrite. Cependant, des rétrovirus aviaires ont été retrouvés intégrés dans un herpès virus [
13] : par analogie avec ce qui a été décrit pour les rétrovirus d’invertébrés, cette capacité d’un rétrovirus à s’intégrer dans le génome d’un virus à grand génome ADN pourrait constituer une étape propice à la capture d’env. Des chats, des souris et des rétrovirus Le modèle murin fournit de nombreux exemples du rôle des rétro-éléments dans l’évolution des génomes et dans la production de nouveaux rétrovirus. Ainsi, les virus leucémogènes murins MLV-MCF ( murine leukemia virus-mink cell focus-forming virus), également appelés MLV polytropes, et MLV 10A1 apparaissent à la suite de recombinaisons entre un MLV infectieux et des séquences rétrovirales endogènes de type env. Ces séquences élargissent le tropisme du MLV par l’acquisition de capacités de liaison à de nouveaux récepteurs (pour revue, voir [ 14]). Toutefois, la dissémination in vivo de ces virus recombinants dépend paradoxalement aussi de leur pseudotypage par l’Env du MLV infectieux [ 9] (Figure 3D). Un processus similaire de recombinaison en cours d’infection a été identifié chez les rétrovirus leucémogènes de chat (FeLV), où la capture d’env par un FeLV de type A produit un nouveau rétrovirus, FeLV B, de tropisme différent [
15]. Gènes env, chaînes de prédation et infections inter-espèces Un des exemples retraçant le mieux la capture d’Env par un rétrovirus de vertébrés est l’échange d’env entre des rétrovirus de babouin et de chat (Figure 4). BaEV ( baboon endogenous virus) est un virus endogène infectieux retrouvé dans plusieurs familles de singes (mandrills, babouins, mangabeys et singes verts africains). BaEV est composé des gènes gag et pol d’un rétrovirus endogène de type C du babouin Papio cynocephalus (PcEV) et d’un gène env du virus endogène simien (SERV) de type D [
16]. Ce même BaEV est impliqué dans l’émergence du virus infectieux endogène félin RD114. Ce dernier est un rétrovirus chimère composé du gène env du BaEV type D et des gènes gag-pol de type C provenant du virus endogène de chat FcEV ( felis catus endogenous virus) [ 16]. À l’origine du RD114 félin existent donc au moins deux événements de capture d’env par recombinaison : la capture du gène env de SERV par PcEV, qui aurait permis l’émergence du BaEV, et la capture du gène env de BaEV par FcEV, donnant naissance au rétrovirus RD114. On peut raisonnablement penser que ces infections inter-espèces prennent place lors de contaminations sanguines occasionnées par des rapports de prédation. 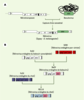 | Figure 4.
Captures d’env. A.
Capture de novo d’un gène env par un rétrotransposon d’invertébré, conduisant à la formation du rétrovirus réplicatif ancestral Gypsy de drosophile [ 11]. B. Acquisition séquentielle d’un gène env de rétrovirus de primates par le rétrovirus endogène félin RD114. Cette capture d’une Env de virus de type BaEV par le rétrovirus endogène de chat FcEV survient après des recombinaisons entre les virus endogènes simiens SERV et PcEV [ 16]. L’enveloppe du RD114 dérivée de cette capture permet l’infection efficace de cellules de primates, y compris humain [
45] (d’après [
46]). |
Une cascade de captures d’enveloppe est peut-être aussi à l’origine de l’émergence du GaLV (gibbon ape leukemia virus), rétrovirus de type C du singe gibbon asiatique. En effet, l’Env du GaLV est apparentée aux Env murine (du MLV) et féline (du FeLV). Sur la base de cette parenté étroite, il est tentant d’établir le schéma d’une transmission successive de ces séquences rétrovirales suivant la voie de prédation ascendante la plus probable, souris-chat-primate. Cette transmission horizontale mettrait en jeu des rétrovirus exogènes de souris (MLV écotropes) capturant des env rétrovirales endogènes de souris, de type « xénotropes », capables ainsi d’élargir leur tropisme à d’autres espèces [14,
17]. Cela leur permettrait d’infecter leurs prédateurs félins, entraînant ainsi l’émergence de nouveaux rétrovirus exogènes félins tels que FeLV-A. De nouveaux rétrovirus félins recombinants d’enveloppe, tels que le FeLV-B, pourraient passer au prédateur primate et y favoriser l’émergence de virus tels que GaLV. On peut donc attendre une parenté étroite entre gènes rétroviraux provenant d’espèces phylogénétiquement éloignées, mais reliées par une « chaîne alimentaire ». De tels échanges de rétrovirus ne dépendraient pas exclusivement de relations de prédation, mais utiliseraient aussi d’autres voies d’échanges entre espèces partageant le même habitat. Ainsi, des recombinaisons au niveau des gènes env ont été décrites entre des rétrovirus de différentes espèces de mammifères pourtant strictement herbivores. Le rétrovirus ovin/caprin JSRV (Jaagsiekte retrovirus), responsable de l’adénocarcinome pulmonaire ovin, porte ainsi des séquences gag de type D et le gène env d’un virus de type B [
18]. Des séquences chimériques de type B et D, apparentées à celle de JSRV, sont présentes non seulement dans le génome du mouton, mais également chez d’autres ongulés, dont la quasi-totalité du genre capra et les vaches domestiquées [
19], suggérant une ancienne capture d’env et une dissémination inter-espèce. Le VQ commun de virus leucémogènes murins et humain Lors de nos travaux sur l’enveloppe du rétrovirus de la leucémie humaine HTLV ( human T-cell leukemia/lymphoma virus), nous rapportions que, bien que les MLV et HTLV soient phylogénétiquement très éloignés sur la base du gène pol, leurs composantes de surface SU présentaient des blocs d’homologies remarquables. Un bloc, en particulier, est relativement conservé entre HTLV et MLV, délimité en aval chez les MLV de Friend et HTLV-1 par les résidus VQ avec, respectivement, les séquences RLLNLVQ et KLLTLVQ ; les domaines de SU délimités par ces séquences sont fonctionnellement interchangeables entre HTLV-1 et MLV de Friend [ 20, 21]. Homologies et conservations de motifs d’Env au sein des génomes MLV et HTLV, par ailleurs très divergents, évoquent la capture d’un gène env ancestral commun. L’analyse phylogénétique de ces séquences devrait fournir de nouveaux indices sur la parenté entre les HTLV, leurs équivalents simiens STLV et les rétrovirus de type C simiens, félins et murins. Des voies de transmission inter-espèces et d’acquisition de nouvelles séquences par les ancêtres de ces virus, par ailleurs très distants, pourraient également être élucidées. Une autre question est celle de la présence de séquences endogènes, env ou autres, apparentées aux HTLV, au sein des génomes de primates, et de leur rôle éventuel dans la physiologie et la physiopathologie humaine. Des env et des génomes Si l’origine de l’enveloppe de certains rétrovirus d’invertébrés est bien établie et repousse la question aux origines mêmes de ces séquences hétérologues, l’origine des Env rétrovirales de vertébrés reste plus énigmatique. Du fait des propriétés fusogènes des Env, une recherche de parenté entre les glycoprotéines Env et les partenaires cellulaires de la fusion ou de l’endocytose membranaire reste privilégiée. Toutefois, les propriétés de la composante de surface SU permettent également d’envisager une parenté avec des gènes codant pour des ligands de récepteurs membranaires (immunoglobulines, cytokines…). Ces deux origines pourraient être conciliées dans le cadre de la formation d’Env par recombinaison stochastique de domaines distincts des protéines [
22,
23], tel que cela est décrit, par exemple, dans le mécanisme de brassage des exons [
24–
26]. La plus forte conservation, au sein des enveloppes rétrovirales, des séquences TM par rapport aux séquences SU, suggère une acquisition modulaire en deux temps, au moins [
27]. À une TM ancestrale bardée de domaines conservés (tels que peptides fusogène, immunodominant et immunosuppresseur [
28,
29]) se serait arrimée une SU d’origine plus variable. Cela est étayé par la possibilité de produire des domaines isolés de SU reconnaissant le récepteur [20,
30,
31] : ces domaines tronqués de la SU nous ont d’ailleurs permis d’identifier le récepteur de la protéine Env de l’HTLV [
32,
33]. Il a également été montré que des fonctions de la protéine Env impliquées dans l’entrée virale pouvaient être reconstituées par un domaine de reconnaissance du récepteur soluble, physiquement dissocié du reste de l’Env [
34]. En ce qui concerne le maintien d’un grand nombre de séquences endogènes env dans les génomes de vertébrés, des fonctions physiologiques essentielles ont été suggérées pour certaines enveloppes endogènes [
35–
37], ainsi que des fonctions vaccinales potentielles (pour revue, voir [14]). À l’inverse, une Env endogène peut aussi faciliter une infection virale. Ainsi, une SU tronquée d’Env endogène de FeLV sert de corécepteur cellulaire lors de l’infection par un isolat thymotrope de FeLV [
38]. L’abondance de rétrovirus chimères naturels et l’altération du spectre d’hôte qui découle de la capture d’env présagent de l’influence permanente des interactions entre rétro-éléments endogènes et exogènes. Cette influence se manifeste par la production de nouveaux rétrovirus et leur impact dans certaines maladies, mais aussi par leur impact sur l’évolution des génomes, du fait de leur capacité à passer dans la lignée germinale de leurs hôtes. Ainsi, l’émergence récente chez les rétrovirus aviaires du sous-groupe J fortement infectieux est au moins en partie due à l’acquisition d’une séquence env fonctionnellement apparentée à une séquence endogène du poulet [
39]. |
Les événements de « capture » d’un gène env sont susceptibles de se produire par au moins trois mécanismes de recombinaison : après co-encapsidation de génomes rétroviraux hétérologues [
40], par brassage intragénomique d’exons par les rétro-éléments [24, 26] et, enfin, après intégration de rétrotransposons ou de rétrovirus dans le génome de virus à ADN tels que les baculovirus ou les virus de type herpès [11, 12]. Ces recombinaisons témoignent de la co-évolution étroite, permanente, des rétrotransposons et des rétrovirus avec les organismes hôtes. L’infection rétrovirale peut aussi être considérée comme l’« introduction d’[une] espèce dans un écosystème : en fonction des avantages sélectifs, la nouvelle espèce peut s’installer de manière stable et permettre un nouvel état de l’ensemble. L’espèce peut aussi s’éteindre ou au contraire proliférer et/ou provoquer un déséquilibre mortel » [
41]. Un des axes actuel de recherche est d’établir des liens entre Env rétrovirales et différentes maladies idiopathiques, dont certaines leucémies, maladies dégénératives et syndromes auto-immuns [
42–
44]. Cependant, bien que la capture d’env, en tant qu’élément clé de la dissémination à de nouveaux organismes, puisse effectivement constituer un événement dont l’impact est délétère, elle reste aussi un acteur essentiel de l’évolution des génomes. |
Nous remercions E. Basyuk, E. Bertrand, C. Denesvre, S. Kinet, A. Lassaux, P. Sonigo, N. Taylor et tous les membres de notre équipe pour les discussions autour de ce manuscrit. Nos travaux ont reçu le soutien de la Fondation de France, l’AFM, l’ANRS, l’ARC et la Fondation Philippe. N.M. est titulaire d’une « Allocation couplée », F.J.K. a reçu le soutien de l’ARC et de la Fondation de France, J.L.B. et M.S. sont chercheurs à l’Inserm.
|
Footnotes |
2. Anxolabéhère D, Nouaud D, Miller WJ. Éléments transposables et nouveautés génétiques chez les eucaryotes. Med Sci (Paris) 2000; 16 : I-IX. 3. Smit AF. Interspersed repeats and other mementos of transposable elements in mammalian genomes. Curr Opin Genet Dev 1999; 9 : 657–63. 4. Tristem M. Identification and characterization of novel human endogenous retrovirus families by phylogenetic screening of the human genome mapping project database. J Virol 2000; 74 : 3715–30. 6. Garoff H, Hewson R, Opstelten DJ. Virus maturation by budding. Microbiol Mol Biol Rev 1998; 62 : 1171–90. 7. Owens RJ, Dubay JW, Hunter E, Compans RW. Human immunodeficiency virus envelope protein determines the site of virus release in polarized epithelial cells. Proc Natl Acad Sci USA 1991; 88 : 3987–91. 8. Basyuk E, Galli T, Mougel M, et al. Retroviral genomic RNAs are transported to the plasma membrane by endosomal vesicles. Dev Cell 2003; 5 : 161–74. 9. Sitbon, M, Nishio J, Wehrly K, Chesebro B. Pseudotyping of dual-tropic recombinant viruses generated by infection of mice with different ecotropic murine leukemia viruses. Virology 1985; 140 : 144–51. 10. Song SU, Gerasimova T, Kurkulos M, et al. An env-like protein encoded by a Drosophila retroelement : evidence that gypsy is an infectious retrovirus. Genes Dev 1994; 8 : 2046–57. 11. Malik HS, Henikoff S, Eickbush TH. Poised for contagion : evolutionary origins of the infectious abilities of invertebrate retroviruses. Genome Res 2000; 10 : 1307–18. 12. Pearson MN, Rohrmann GF. Transfer, incorporation, and substitution of envelope fusion proteins among members of the baculoviridae, orthomyxoviridae, and metaviridae (insect retrovirus) families. J Virol 2002; 76 : 5301–4. 13. Isfort RJ, Witter R, Kung HJ. Retrovirus insertion into herpesviruses. Trends Microbiol 1994; 2 : 174–7. 14. Sitbon M, Denesvre C, Dardalhon V, et al. Les rétrovirus leucémogènes murins (MLV) : pathogènes, gènes, et outils génétiques. Virologie 2001; 5 : 265–93. 15. Stewart MA, Warnock M, Wheeler A, et al. Nucleotide sequences of a feline leukemia virus subgroup A envelope gene and long terminal repeat and evidence for the recombinational origin of subgroup B viruses. J Virol 1986; 58 : 825–34. 16. Van der Kuyl AC, Dekker JT, Goudsmit J. Discovery of a new endogenous type C retrovirus (FcEV) in cats : evidence for RD-114 being an FcEV(Gag-Pol)/baboon endogenous virus BaEV(Env) recombinant. J Virol 1999; 73 : 7994–8002. 17. Battini JL, Heard JM, Danos O. Receptor choice determinants in the envelope glycoproteins of amphotropic, xenotropic, and polytropic murine leukemia viruses. J Virol 1992; 66 : 1468–75. 18. York DF, Vigne R, Verwoerd DW, Querat G. Nucleotide sequence of the jaagsiekte retrovirus, an exogenous and endogenous type D and B retrovirus of sheep and goats. J Virol 1992; 66 : 4930–9. 19. Hecht SJ, Stedman KE, CarlsonJO, DeMartini JC. Distribution of endogenous type B and type D sheep retrovirus sequences in ungulates and other mammals. Proc Natl Acad Sci USA 1996; 93 : 3297–302. 20. Kim FJ, Manel N, Garrido EN, et al. Human T-cell leukemia virus type 1 and 2 envelope SU subdomains and critical determinants in receptor binding. 2004 (soumis pour publication). 21. Kim FJ, Seiliez I, Denesvre C, et al. Definition of an amino-terminal domain of the human T-cell leukemia virus type 1 envelope surface unit that extends the fusogenic range of an ecotropic murine leukemia virus. J Biol Chem 2000; 275 : 23417–20. 22. Lerat E., Brunet F, Bazin C, Capy P. Is the evolution of transposable elements modular ? Genetica 1999; 107 : 15–25. 23. Lerat E, Capy P. Retrotransposons and retroviruses : analysis of the envelope gene. Mol Biol Evol 1999; 16 : 1198–207. 24. Boeke JD, Pickeral OK. Retroshuffling the genomic deck. Nature 2001; 398 : 108–9, 111. 25. Courseaux A, NahonJL. Birth of two chimeric genes in the Hominidae lineage. Science 1999; 291 : 1293–7. 26. Pavlicek, A, Paces J, Elleder D, Hejnar J. Processed pseudogenes of human endogenous retroviruses generated by LINEs : their integration, stability, and distribution. Genome Res 2002; 12 : 391–9. 27. Benit L, Dessen P, Heidmann T. Identification, phylogeny, and evolution of retroviral elements based on their envelope genes. J Virol 2001; 75 : 11709–19. 28. Gallaher WR, Ball JM, Garry RF, et al. A general model for the transmembrane proteins of HIV and other retroviruses. AIDS Res Hum Retrovir 1989; 5 : 431–40. 29. Pancino G, Ellerbrok H, Sitbon M, Sonigo P. Conserved framework of envelope glycoproteins among lentiviruses. Curr Top Microbiol Immunol 1994; 188 : 77–105. 30. Battini JL, Danos O, Heard JM. Receptor-binding domain of murine leukemia virus envelope glycoproteins. J Virol 1995; 69 : 713–9. 31. Fass D, Davey RA, Hamson CA, et al. Structure of a murine leukemia virus receptor-binding glycoprotein at 2.0 angstrom resolution. Science 1997; 277 : 1662–6. 32. Manel N, Kim FJ, Kinet S, et al. The ubiquitous glucose transporter GLUT-1 is a receptor for HTLV. Cell 2003; 115 : 449–59. 33. Manel N, Kinet S, Battini JL, et al. The HTLV receptor is an early T cell activation marker whose expression requires de novo protein synthesis. Blood 2002; 17 : 17. 34. Lavillette D, Ruggieri A, Russell JS, Cosset FL. Activation of a cell entry pathway common to type C mammalian retroviruses by soluble envelope fragments. J Virol 2000; 74 : 295–304. 35. Blond JL, Lavillette D, Cheynet V, et al. An envelope glycoprotein of the human endogenous retrovirus HERV-W is expressed in the human placenta and fuses cells expressing the type D mammalian retrovirus receptor. J Virol 2001; 74 : 3321–9. 36. Kattstrom PO, Bjerneroth G, Nilsson BO, et al. A retroviral gp70-related protein is expressed at specific stages during mouse oocyte maturation and in preimplantation embryos. Cell Differ Dev 1989; 28 : 47–54. 37. Mi SX, Lee X, Li X, et al. Syncytin is a captive retroviral envelope protein involved in human placental morphogenesis. Nature 2000; 403 : 785–9. 38. Anderson MM, Lauring AS, Burns CC, Overbaugh J. Identification of a cellular cofactor required for infection by feline leukemia virus. Science 2000; 287 : 1828–30. 39. Denesvre C, Soubieux D, Pin G, et al. Interference between avian endogenous ev/J 4.1 and exogenous ALV-J retroviral envelopes. J Gen Virol 2003; 84 : 3233–8. 40. Stuhlmann H, Berg P. Homologous recombination of copackaged retrovirus RNAs during reverse transcription. J Virol 1992; 66 : 2378–88. 41. Sonigo P. Pour une biologie moléculaire darwinienne. Med Sci (Paris) 2002; 18 : 1038–9. 42. Lower, R. The pathogenic potential of endogenous retroviruses : facts and fantasies. Trends Microbiol 1999; 7 : 350–6. 43. Perron H, Seigneurin JM. Human retroviral sequences associated with extracellular particles in autoimmune diseases : epiphenomenon or possible role in aetiopathogenesis ? Microbes Infect 1999; 1 : 309–22. 44. Portis JL. Perspectives on the role of endogenous human retroviruses in autoimmune diseases. Virology 2002; 296 : 1–5. 45. Cosset FL, Takeuchi LJ, Battini RA, et al. High-titer packaging cells producing recombinant retroviruses resistant to human serum. J Virol 1995; 69 : 7430–6. 46. Kim FJ, Battini JL, Manel N, Sitbon M. Emergence of vertebrate retroviruses and envelope capture. Virology 2004; 318 : 183–91. |