| |
| Med Sci (Paris). 2002 December; 18(12): 1267–1275. Published online 2002 December 15. doi: 10.1051/medsci/200218121267.Nouveaux aspects de la biologie de la protéine prion Mangé Alain and Lehmann Sylvain* Institut de Génétique Humaine, Cnrs UPR 1142, 141, rue de la Cardonille, 34396 Montpellier Cedex 5, France |
Les encéphalopathies spongiformes subaiguës transmis-sibles (ESST) représentent un groupe d’affections neuro-dégénératives qui comprend principalement, chez l’homme, la maladie de Creutzfeldt-Jakob, et, chez l’animal, la tremblante du mouton et l’encéphalopathie spon-giforme bovine. Également appelées maladies à prion, ces affections peuvent avoir une origine sporadique, infectieuse ou génétique. L’agent infectieux à l’origine de ces maladies est dit non conventionnel et serait de nature protéique [1] (→). Cette hypothèse, qui implique une transmission de l’information indépendante de tout matériel génétique, est encore très controversée. L’événement central de la pathogénie de ces maladies semble être la conversion d’une protéine appelée PrPC (forme cellulaire de la protéine du prion) en une isoforme pathogénique appelée PrPSc (forme scrapie de la PrP) [2]. Les événements qui conduisent à la transmission ou à la propagation des prions ne sont pas détaillés dans cet article. En revanche, cette synthèse se focalise sur la biologie de la PrPC dont la compréhension semble essentielle pour appréhender la fonction de la protéine et les conséquences de sa conversion en PrPSc au cours de la maladie. (→) m/s 2002, n°1, p. 62 |
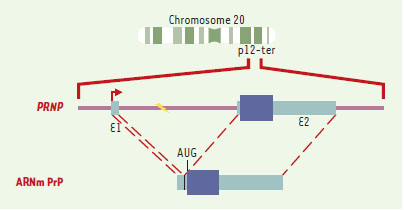
Du gène à la protéine:structure et expression de la protéine prion La PrPC est une glycoprotéine de 33 à 35 kDa codée par un gène unique localisé sur le chromosome 20 humain (PRNP) et le chromosome 2 murin (Prnp) [3]. L’analyse des séquences nucléotidiques a montré que ces gènes étaient morcelés, mais que la phase de lecture codant pour la protéine était comprise dans un seul exon (Figure 1). Le gène de la PrPC est exprimé de manière constitutive dans la plupart des tissus, avec néanmoins une expression plus importante dans le cerveau. Chez les rongeurs, l’expression du gène Prnp varie au cours du développement, avec une augmentation de son expression dès le 10e jour embryonnaire [4]. Il existe à l’heure actuelle très peu de données sur la région régulatrice du gène de la PrPC, qui a les caractéristiques générales d’un gène dit de « ménage » contrôlé par des facteurs transcriptionnels ubiquitaires [5, 6]. 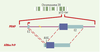 | Figure 1.Du gène à la protéine. Le gène codant pour la PrP C est localisé sur le chromosome 20 humain au locus p12-pter. La région promotrice du gène PRNP humain est suivie par un exon 1 (E1) transcrit non traduit de 134 paires de bases (pb), d’un intron de 12 696 pb et d’un exon 2 (E2) de 2 355 pb incluant la phase ouverte de lecture de 759 pb et une région 3’ transcrite non traduite [ 5]. Chez la souris et chez le mouton, il existe un exon non traduit supplémentaire de 99 pb localisé 2 303 pb en aval de l’exon I [ 51]. Par ailleurs, le gène Prnp de souris possède deux promoteurs distincts en amont des exons non traduits en 5’ [ 52]. Le site d’initiation de la transcription localisé dans l’exon 1 (flèche) permet l’expression d’un ARNm qui contient une région 5’ non traduite de 168 nucléo-tides, un site d’initiation de la traduction (AUG) définissant la phase ouverte de lecture codant pour la PrP C et une région 3’ transcrite non traduite de 1 562 nucléotides contenant un site unique de polyadénylation. |
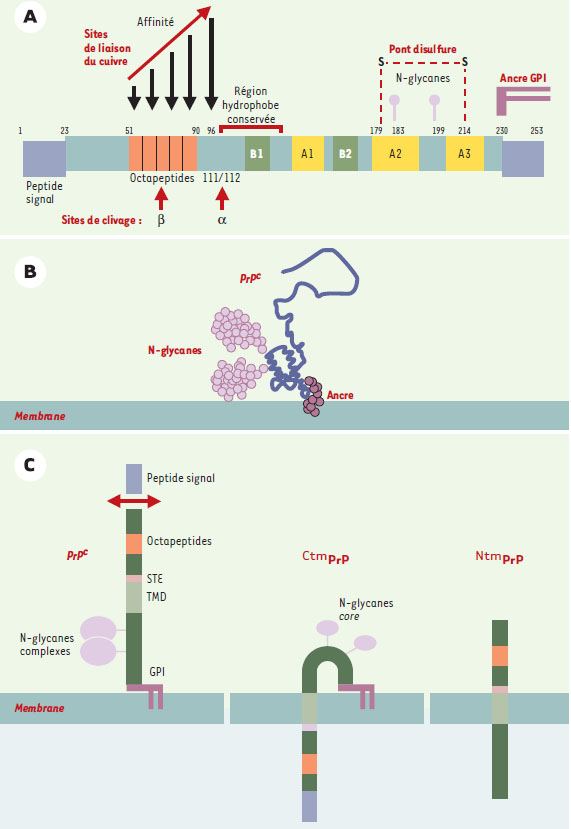
La structure de la PrPC est représentée sur la Figure 2A. Elle possède un peptide signal hydrophobe de 22 acides aminés dans la région amino-terminale qui permet sa translocationdans le réticulum endoplasmique (RE). Dans cette même région, on trouve une répétition de 5 octapeptides de séquence consensus PHGGGWGQ puis, dans une région plus centrale, un domaine hydrophobe très conservé dans toutes les espèces. Une séquence hydrophobe localisée dans la région carboxy-terminale contrôle l’ajout d’une ancre gly-cosyl-phosphatidylinositol (GPI). La PrPC des mammifères contient deux sites de glycosylation et deux résidus cys-téines reliés par un pont disulfure. L’analyse par RMN (résonance magnétique nucléaire) de PrPC recombinantes de souris ou de hamster révèle que la région amino-terminale de ces protéines est très flexible et peu structurée dans les conditions expérimentales utilisées [7]. Le domaine car-boxy-terminal présente une structure globulaire constituée de deux feuillets β et de trois hélices α (Figure 2, A et B) [8]. 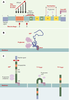 | Figure 2. Structure de la PrPc. A. Structure primaire de la protéine prion chez l’homme. La PrPC humaine possède un peptide signal (résidus 1-22) qui est clivé après l’entrée de la molécule dans le réticulum endoplasmique (RE). Par suite du clivage d’une séquence hydrophobe dans la partie C-terminale, une ancre glycosyl-phosphatidylinositol (GPI) sialylée est attachée au niveau du résidu 230. Un pont disulfure relie les deux cys-téines 179 et 214. La PrPC peut être non-, mono- ou di-glyco-sylée au niveau des deux asparagines 183 et 199. Au cours du trafic intracellulaire, 1 à 5 % des molécules sont clivées entre les résidus 111 et 112 (α) dans une région α hydrophobe très conservée (crochet, résidus 111 à 134). Un deuxième site de clivage (β) semble exister dans une région répétée de 5 octapep-tides (résidus 51 à 90). Cette région qui contient notamment des résidus histidine est capable de lier entre 4 et 6 atomes de cuivre avec une affinité croissante. L’étude en RMN de la protéine recombinante a montré que la moitié N-terminale de la molécule (résidus 23 à 120) était non structurée, alors que deux petits feuillet β (B1 et B2, résidus 128-131 et 161-164) et trois hélices α (A1, A2 et A3, résidus 144-156, 172-193 et 200-227) sont retrouvésdans la région C-terminale. B. Structure de la PrPC à la surface cellulaire. La chaîne polypeptidique de PrPC est ancrée à la membrane plasmique par son ancre GPI (violet foncé). Elle possède deux N-glycanes (violet clair) attachés dans la région C-terminale globulaire de la molécule. C. Formes topologiques de la PrPC. La maturation normale de la PrPC conduit à une protéine privé de son peptide signal N-terminal, ancrée à la surface membranaire par son ancre GPI. Les sites de glycosylation peuvent être occupés par des N-glycanes complexes. Mais la PrPc possède aussi deux séquences qui participent à la production de formes transmembranaires de la protéine: une séquence STE pour stop transfer effector (rose) et une région hydrophobe ou TMD pour trans membrane domain (vert clair). La CtmPrP a son extrémité C-terminale située dans la lumière du RE. Outre le domaine transmembranaire, elle est ancrée par une ancre GPI. Son peptide signal N-terminal n’est pas clivé et ses glycanes ne sont pas matures. La forme NtmPrP, quant à elle, a une orientation inverse et ses sites de glycosylation ne sont donc pas utilisés. Cette forme a été décrite principalement in vitro. |
|
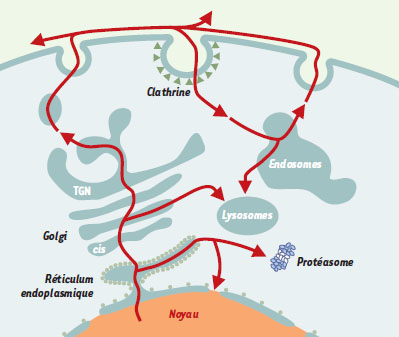
Une protéine pour trois formes topologiques La PrP C, glycoprotéine présente à la surface cellulaire, suit un cheminement biosynthétique classique (Figure 3). Une fois transportée dans le réticulum endoplasmique et après clivage du peptide signal amino-terminal, la protéine transite jusqu’à la membrane plasmique. Néanmoins, il a été décrit deux autres formes topologiques de la molécule ( CtmPrP et NtmPrP) qui possèdent un domaine transmembranaire correspondant à la séquence hydro-phobe conservée de la molécule située entre les positions 111 et 134 (Figure 2C) [ 9]. L’extrémité carboxy-terminale de la CtmPrP est orientée dans la lumière du réticulum endoplasmique.Outre le domaine transmembranaire, la CtmPrP est fixée par une ancre GPI et présente la particularité de conserver son peptide signal amino-terminal [ 10]. La forme NtmPrP présente une orientation inversée par rapport à la CtmPrP et n’est donc ni glycosylée, ni glypiée [ 9]. Ces formes ont été initialement observées dans un système de traduction in vitro [ 11]. Par la suite, la CtmPrP a pu être détectée in vivo et en culture cellulaire [ 9, 12, 13]. Le mécanisme précis aboutissant à ces formes est encore très largement incompris. Il pourrait impliquer trois séquences clés: le peptide signal, une séquence STE ( stop transfer effector) (position 103-111) qui induit une pause dans la translocation de la protéine, et la séquence hydrophobe (TMD, trans membrane domain) (acides aminés 111-134) qui permet l’ancrage de la protéine sous une forme transmembranaire stable (Figure 2C) [ 9, 14]. La proportion relative des trois isoformes serait déterminée à la fois par la séquence STE et par des facteurs cytosoliques [ 15]. 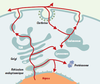 | Figure 3. Cheminement cellulaire de la PrP. Après sa synthèse, la chaîne polypepti-dique de la PrPC est transloquée dans le réticulum endoplasmique (RE). Normalement, cette translocation est complète et conduit au clivage du peptide signal N-terminal et à l’attachement de l’ancre GPI (glycosylphosphatidylinositol). Dans certains cas, la translocation de la molécule n’est pas complète, engendrant ainsi des formes transmembranaires qui s’accumulent dans le RE (Figure 2C) avant d’être dégradées dans le cytoplasme par le protéasome. Ce passage de la PrP dans le cytoplasme pourrait expliquer la détection par certains auteurs de la molécule dans le noyau. La PrPc transloquée poursuit sa maturation dans l’appareil de Golgi où un second contrôle de qualité peut conduire au transport des molécules anormales vers les lysosomes. La PrPC s’associe aux DRM (detergent resistant microdomains) et se retrouve ensuite à la surface plasmique. Elle est l’objet d’un processus continu d’endocytose et de recyclage au cours duquel une fraction des molécules est clivée en deux sites dans leur partie N-terminale. TGN : trans-Golgi network. |
Il a été proposé que l’accumulation de la forme CtmPrP soit responsable de la neurodégénérescence observée dans les ESST génétiques et infectieuses [10, 12, 13]. Cependant, ce lien de causalité ne semble pas absolu car certaines mutations pathologiques de la PrP ne semblent pas favoriser la production de CtmPrP [10]. Contrôle/qualité des protéines et protéasome Les ESST humaines ont une incidence très faible, indiquant que la formation spontanée de PrP Sc est un processus rare. Une des caractéristiques de ces maladies réside dans la capacité de la PrP C d’adopter des conformations multiples, soit de manière spontanée (forme sporadique), soit à la suite de mutations (forme génétique) ou encore par conversion (forme infectieuse). Des mécanismes cellulaires assurent normalement la détection et la dégradation rapide des protéines de conformation incorrecte. Chez les eucaryotes, un des principaux points de contrôle est localisé dans le réticu-lum endoplasmique où les protéines anormales sont stockées avant d’être exportées dans le cytoplasme puis dégradées par le protéasome (pour revue, voir [ 16]). Plusieurs études ont montré récemment qu’un tel système était impliqué dans la dégradation de PrP mutées pathologiques [ 13, 17], mais également de la PrP C elle-même [ 6, 18]. Ainsi, quand l’activité du système de contrôle/qualité des protéines est altérée artificiellement, notamment par des inhibiteurs du protéasome, environ 10 % de la PrP C se retrouve dans le cytoplasme. Cette forme, qui est déglycosylée et ubiquitinylée, se présente en agrégats insolubles et résistants à la protéi-nase K, deux des caractéristiques de la PrP Sc. Ces résultats suggèrent qu’il existe un flux constant de PrP C duréticulum endoplasmique vers le cytoplasme en situation non pathologique. Ce flux pourrait augmenter dans le cas de dysfonctionnements du protéasome dû au vieillissement ou à un stress et avoir des conséquences pathologiques. Tout d’abord, cette PrP C qui présente des caractéristiques proches de la PrP Sc pourrait expliquer l’apparition des ESST sporadiques. Par ailleurs, elle pourrait rendre compte de la neurodégénérescence observée dans les formes génétiques où les PrP mutantes s’accumulent dans le réticulum endoplas-mique et pourraient activer certaines voies de signalisation liées au stress du réticulum. Cette localisation cytoplasmique de la PrP c pourrait également expliquer, d’une part, l’interaction de la PrP c avec des partenaires tels que Bcl-2 ou Hsp60 (Tableau I) et, d’autre part, la présence de la protéine dans le noyau par un transport actif via un signal de localisation nucléaire présomptif situé dans la région amino-terminale [ 19– 21]. Pour conclure, il semble exister également une deuxième étape de contrôle au niveau de l’appareil de Golgi, au cours de laquelle les PrP c possédant une conformation anormale seraient redirigées vers les lysosomes pour y être dégradées [ 22].  | Tableau I. Protéines interagissant avec la PrP. |
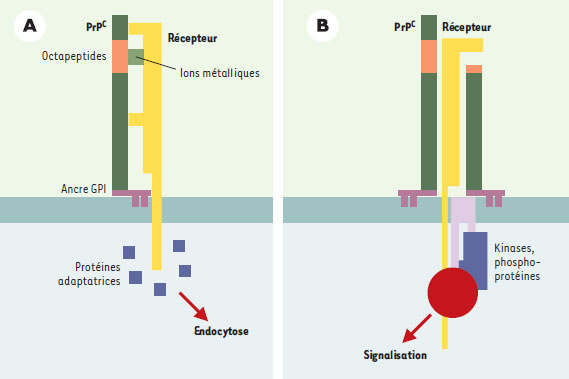
Cheminement intracellulaire, clivage et dégradation Tout au long de son cheminement dans le réticulum endoplasmique puis dans l’appareil de Golgi, la PrP C va subir des modifications post-traductionnelles (glycosy-lation, ancrage GPI, formation d’un pont disulfure (pour revue, voir [ 23]). Dès son passage dans l’appareil de Golgi, la PrP C est concentrée, comme la plupart des autres protéines à ancre GPI, dans de larges domaines enrichis en cholestérol et en sphingolipides. Ces complexes correspondent à des microdomaines spécialisés de la membrane plasmique appelés raft ou DRM ( detergent resistant microdomains). Outre la PrP C, ces DRM contiennent également plusieurs molécules impliquées dans des voies de signalisation comme les tyrosine kinases de la famille Src et les protéines G. En revanche, les DRM contenant la PrP C semblent avoir une composition lipidique différente de ceux contenant des molécules à ancre GPI classique comme Thy-1 [ 24]. Cela pourrait expliquer pourquoi la PrP C, ainsi synthétisée et exprimée à la membrane, est rapidement « endocytée » de la surface membranaire à la différence de Thy-1 qui y est très stable. Cette endocytose semble suivre principalement les puits de clathrine [ 25]. Cette voie nécessite une interaction directe avec la clathrine et suggère ainsi que la PrP interagisse directement ou indirectement avec des molécules transmembranaires (Figure 4). La région amino-terminale de la molécule joue un rôle crucial dans cette étape d’internalisation [ 26]. En effet, le clivage physiologique ( voir plus loin) ou la délétion de ce domaine abolit l’endocytose. Après l’internalisation de la PrP C, la majorité des molécules va être recyclée à la surface cellulaire et un faible pourcentage est dirigé vers les lysosomes pour y être dégradé [ 27]. Au cours de ce cycle, 1 à 5 % des molécules vont subir un clivage dans la région centrale hydrophobe entre les acides aminés 111 et 112 (site α, Figure 2A). Ce clivage semble réglé par la protéine kinase C et implique les métalloprotéases TACE et ADAM10 [ 28, 29]. Il est intéressant de noter que ce clivage détruit la région hydrophobe de la PrP C qui est décrite comme neurotoxique et que la PrP Sc, probablement en raison du changement conformationnel de la protéine, n’est pas clivée au site α. La molécule clivée est ensuite ré-exprimée à la surface cellulaire où elle reste jusqu’à sa dégradation. Il existe un autre site de clivage de la PrP situé en amont du site α, dans la région des octapeptides (site β, Figure 2A). Ce clivage, principalement décrit pour la PrP Sc [ 30], semble dépendre à la fois de la présence de cuivre et de radicaux libres [ 31]. 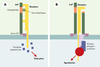 | Figure 4. Interaction de la PrP avec d’autres protéines à la surface cellulaire. A. Endocytose. La PrPC (vert foncé) est endocytée rapidement à la surface cellulaire contrairement à d’autres molécules à ancre GPI telle que Thy-1. Cette endocytose s’effectue principalement dans des puits de clathrine. La PrPC ne possédant pas de domaine intracyto-plamique, elle doit interagir avec une autre molécule transmembranaire (récepteur jaune) qui induira l’endocytose grâce à des protéines adaptatrices (cubes bleus). La partie N-terminale de la PrPC et la région des octapeptides (en orange) jouent un rôle prépondérant dans ce processus. En effet, la délétion de cette région entraîne une diminution de l’endocytose, alors que la fixation d’ions métalliques (cube vert) module la quantité de PrPC présente à la surface cellulaire. B. Signalisation. Plusieurs travaux ont montré que la PrPC pouvait être impliquée et dans des phénomènes de signalisation cellulaire. Différents modèles peuvent être proposés pour rendre compte d’une transduc-tion de signal induite par la PrPC. Comme pour l’endocytose, l’intervention d’un récepteur capable de transduire un signal semble le plus probable. Ce récepteur (jaune) pourrait interagir avec la PrPC entière, ou clivée, ou encore présente à la surface cellulaire sous la forme d’un dimère. Ce récepteur pourrait faire partie d’un complexe de trans-duction plus large comprenant des protéines membranaires (violet clair) et cytoplasmiques (cubes bleus, sphère rouge), dont notamment des phosphoprotéines et des kinases de la famille Src. |
|
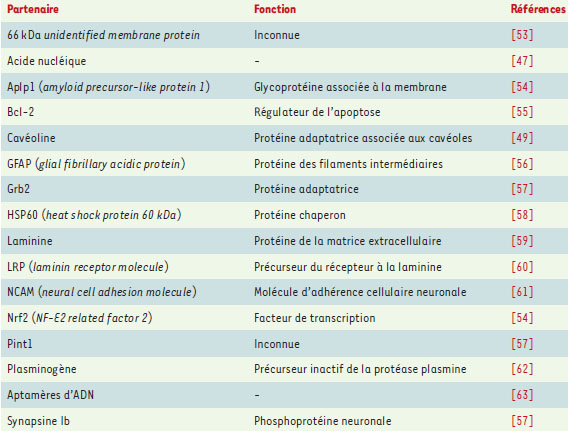
PrP C : une protéine multifonctionnelle? À la recherche d’une fonction inconnue Malgré plusieurs pistes sérieuses, la fonction de la PrP C reste encore hypothétique. Compte tenu de son ancre GPI, de son trafic intracellulaire et de son transport par le flux axonal rapide à l’extrémité pré-synaptique, il a été proposé que la PrP C pourrait contribuer à la stabilisation ou à la transmission synaptique. Néanmoins, d’autres hypothèses ont été avancées, notamment un rôle dans l’adhérence ou la reconnaissance intercellulaires, dans la transduction de signal ou encore comme récepteur. L’approche classique consistant à invalider le gène de la PrP chez la souris (PrP−/−) a fourni des résultats contrastés. Ces souris dont le gène de la PrP a été invalidé ont permis de montrer que la protéine du prion était nécessaire au développement et à la transmission des ESST [32], mais n’ont en revanche pas permis de donner une réponse satisfaisante quant à sa fonction. En effet, sur trois lignées transgéniques indépendantes, deux présentent un phénotype quasi normal et sont parfaitement viables [33, 34] et la dernière présente des symptômes neurologiques de type ataxie ainsi qu’une dégénérescence des cellules de Purkinje [35]. Ce dernier phéno-type résulte en fait de l’expression ectopique dans le cerveau d’un gène codant pour une protéine appelée Doppel, localisée juste en aval du gène de la PrPc [36]. Doppel présente des analogies avec la PrPC, notamment au niveau structural. La réexpression de la PrPC dans le cerveau de ces souris est capable d’inverser la dégénérescence induite par Doppel, suggérant que les deux molécules pourraient avoir des effets antagonistes et interagir avec un récepteur commun. Pour les autres souris PrP−/−, il existe toujours des controverses concernant des anomalies phénotypiques mineures, notamment électrophysiologiques. Il reste que la délétion du gène de la PrPc n’entrave pas le développement et n’altère pas significativement le comportement des souris. Cette absence évidente de phénotype suggère que la PrPC n’exerce pas de fonction vitale pour l’organisme et/ou que les fonctions assurées par cette protéine peuvent être compensés par d’autres molécules. La région amino-terminale : domaine clé de la PrPc De nombreux arguments aussi bien génétiques, biochimiques que structuraux montrent l’importance de la région N-terminale de la PrP C dans la physiologie cellulaire et la pathologie des ESST. Cette région contient notamment une série d’octapeptides capables de se lier à des ions métalliques bivalents, en particulier le cuivre et avec des affinités moindres au zinc et au manganèse (Figure 2A) [ 37]. La PrP C pourrait ainsi lier, de façon coopérative, quatre à six atomes de cuivre avec des affinités de l’ordre du micromolaire jusqu’au femtomolaire [ 38, 39]. De telles affinités suggèrent que le cuivre est effectivement lié à la PrP C in vivo. Les résidus histi-dines des octapeptides, ainsi que ceux situés plus en amont, notamment en position 96, pourraient être impliqués dans la coordination des ions. La signification physiologique de l’interaction PrP C/cuivre peut-être interprétée de diverses manières. Tout d’abord, le cuivre pourrait contribuer à l’acquisition d’une structure tridimensionnelle fonctionnelle de la PrP C [ 40]. L’association du cuivre et de la PrP C a conduit à l’hypothèse d’un rôle de la protéine dans le transport et le métabolisme de cet ion. Cette hypothèse repose sur l’observation d’une endocytose de la PrP C dépendante d’ions métalliques (cuivre et zinc essentiellement) et sur une étude montrant une réduction de 10 à 15 fois de la concentration en cuivre dans le cerveau de souris transgéniques PrP−/− par rapport aux animaux témoins [ 41, 42]. Cependant, une deuxième étude n’a pas permis de retrouver une telle différence ni de trancher quant au rôle de la PrP C comme protéine majeure de liaison et de transport du cuivre [ 43]. De nombreux résultats impliquent cependant la PrP C dans les mécanismes de défense cellulaire contre le stress oxydant, probablement en relation avec les ions métalliques. Il est en effet établi que les métaux de transition comme le cuivre, le zinc ou le fer peuvent être impliqués dans des réactions oxydantes au niveau cellulaire. De telles réactions jouent un rôle prépondérant dans les processus aboutissant à la mort cellulaire dans de nombreuses maladies neurodégénératives telles que les maladies de Parkinson, d’Alzheimer et de Charcot. Dans plusieurs modèles expérimentaux d’ESST, le niveau d’expression de la PrP C a pu être lié à la résistance cellulaire au stress induit par des radicaux libres ou des ions métalliques, et à l’augmentation de l’activité d’une enzyme clé du système de défense cellulaire contre le stress, la superoxyde dismutase (SOD) [ 44]. Ces observations seraient dues à une activation de l’enzyme SOD dépendante du cuivre et du zinc via la PrP C [ 44] et/ou à une activité SOD- like de la PrP C [ 45]. L’étude de cellules et d’animaux infectés par les prions a montré par ailleurs que la réplication de l’agent infectieux altérait également la réponse cellulaire au stress oxydant, l’activité d’enzymes anti-oxydantes et le métabolisme des ions métalliques (pour revue, voir [ 46]). En conclusion, bien que la relation entre la PrPC, le cuivre et les radicaux libres reste à éclaircir, la fonction de la PrPC pourrait être liée aux mécanismes de défense cellulaire contre le stress oxydant. Dans ce contexte, le clivage de la PrPC au niveau du site β pourrait intervenir dans le métabolisme du cuivre en permettant notamment le relargage de l’ion dans un compartiment intracellulaire, ou encore être impliqué dans l’activation d’une voie de signalisation aboutissant à une réponse cellulaire de défense contre le stress oxydant. Dans cette hypothèse, le clivage subséquent au niveau du site α pourrait permettre l’inactivation de la PrPC. Les partenaires de la PrPC De nombreuses stratégies, principalement de double hybride, ont été mises en place pour identifier des protéines susceptibles d’interagir avec la PrP C. Plus de douze partenaires ont été identifiés et sont donc suppo sés participer à la fonction de la PrP C ou aux mécanismes moléculaires aboutissant à la neurodégénérescence observée dans les ESST (Tableau I). Pour la plupart de ces protéines, la réalité physiologique de ces interactions reste à démontrer. Cependant, les récents résultats sur la localisation cytoplasmique et nucléaire de la PrP C suggèrent que certaines de ces interactions, notamment avec les acides nucléiques, ne sont pas fortuites [ 47]. Parmi les candidats les plus sérieux, on peut citer les molécules d’adhérence cellulaire neuronales (N-CAM), le précurseur du récepteur de la laminine (LRP), le récepteur de la laminine (LR) et la laminine elle-même (Tableau I). La laminine, protéine de la matrice extracellulaire, joue un rôle central, tout comme les N-CAM, dans la régulation de la prolifération et de la différenciation cellulaires et dans la mort neuronale. On peut donc supposer que l’interaction entre la PrP C et ces molécules puisse avoir des conséquences directes sur la survie neuronale, l’extension des neurites ou la différenciation. Par ailleurs, l’interaction entre la PrP C et les protéines membranaires LRP/LR induit l’internalisation de la PrP C, processus qui pourrait être lié aussi à l’activa-tion d’une voie de signalisation [ 48]. L’implication potentielle de la PrP C dans une cascade de signalisation via la tyrosine kinase de la famille Src, p59 Fyn, a été décrite dans un modèle cellulaire neuronal [ 49]. Récemment, une autre étude est venue renforcer ces données en montrant que la PrP C est capable d’interagir in vitro et in vivo avec deux molécules impliquées dans des processus de signalisation neuronale: la synapsine Ib et Grb2 (Tableau I). Bien qu’encore parcellaires, toutes ces données concordent avec la localisation de la PrP C dans les DRM qui sont connus pour concentrer tous les acteurs de la transduction du signal. Dans le cadre des ESST, ces mécanismes pourraient être altérés et induire des effets délétères sur la cellule. Des données très récentes montrent ainsi que l’infection de cultures cellulaires neuronales par des prions affecte les voies de signalisation activées par le récepteur de l’insuline et augmente de façon importante le niveau de la kinase Erk2 sous sa forme phosphorylée [ 50]. Ce dysfonctionnement induit par la PrP Sc pourrait être une cause de la neurodégénérescence observée dans les ESST. |
En dépit d’avancées significatives en biologie cellulaire et moléculaire de la PrPC, nos connaissances sont encore parcellaires. Cette protéine étant le précurseur de la forme anormale PrPSc, la compréhension de la physiopa-thologie des ESST passe par l’étude complète de la biologie de la PrPC. À l’heure actuelle, il manque une vision globale de la fonction normale de la PrPC dont l’altération pourrait jouer un rôle majeur dans la neuropathologie des ESST. Cet article fournit une vue générale de la biologie de la PrPC, et en particulier de ses nouveaux aspects. Plusieurs champs de recherche découlent de ces données, notamment concernant le rôle du système ubiquitine/protéasome dans les ESST, les mécanismes d’endocytose de la PrPC, les interactions de la PrPC avec le cuivre ou ses protéines partenaires, ou encore le lien entre la PrPC et les systèmes cellulaires de défense contre le stress oxydant. |
Note ajoutée aux épreuves Depuis l’acceptation de cet article pour publication dans médecine/sciences, plusieurs articles ont souligné l’importance du transport rétrograde de la PrPc au niveau du réticu-lum endoplasmique. Comme nous l’avons indiqué précédemment, à l’état physiologique, 10 à 15% de PrPC mal repliées dans le réticulum endoplasmique sont dégradées par le pro-téasome. Dans le cas où l’activité du protéasome est altérée, ces formes mal repliées de la PrPC s’accumulent dans le cyto-sol et provoquent la mort neuronale par apoptose comme le décrivent deux travaux récents commentés dans la Brève de F. Béranger, p. 1207 de ce numéro [64, 65]. De plus, nous avons montré récemment que la PrPc pouvait être transportée de manière rétrograde de la surface cellulaire jusqu’au réticulum endoplasmique et que ce compartiment intracellulaire pourrait jouer un rôle très important dans la conversion de la PrPC en PrPSc [66]. Dans la mesure où la conversion s’effectuerait effectivement dans le réticulum endoplasmique, la proportion de PrPc mal repliée pourrait alors augmenter dans ce compartiment, entraînant, à la suite d’une accumulation de PrPc dans le cytoplasme, la neurodégénérescence cérébrale. |
Nous tenons à remercier les membres du laboratoire pour la lecture critique du manuscrit, ainsi que le Cnrs, le GIS-Prion et l’Union européenne (BIO4CT98-6055) pour leur soutien financier.
|
1. Prusiner SB. Novel proteinaceous infectious particles cause scrapie. Science 1982; 216: 136–44. 2. Prusiner SB, Scott MR, Dearmond SJ, Cohen FE. Prion protein biology. Cell 1998; 93: 337–48. 3. Sparkes RS, Simon M, Cohn VH, et al. Assignment of the human and mouse prion protein genes to homologous chromosomes. Proc Natl Acad Sci USA 1986; 83: 7358–62. 4. Manson J, West JD, Thomson V, et al. The prion protein gene: a role in mouse embryogenesis? Development 1992; 115: 117–22. 5. Puckett C, Concannon P, Casey C, Hood L. Genomic structure of the human prion protein gene. Am J Hum Genet 1991; 49: 320–9. 6. Ma J, Lindquist S. Wild-type PrP and a mutant associated with prion disease are subject to retrograde transport and proteasome degradation. Proc Natl Acad Sci USA 2001; 98: 14955–60. 7. Donne DG, Viles JH, Groth D, et al. Structure of the recombinant full-length hamster prion protein PrP (29-231): the N-terminus is highly flexible. Proc Natl Acad Sci USA 1997; 94: 13452–7. 8. Riek R, Hornemann S, Wider G, et al. NMR structure of the mouse prion protein domain PrP (121-231). Nature 1996; 382: 180–2. 9. Hegde RS, Mastrianni JA, Scott MR, et al. A transmembrane form of the prion protein in neurodegenerative disease. Science 1998; 279: 827–34. 10. Stewart RS, Harris DA. Most pathogenic mutations do not alter the membrane topology of the prion protein. J Biol Chem 2001; 276: 2212–20. 11. Hay B, Barry RA, Lieberburg I, Prusiner SB, Lingappa VR. Biogenesis and transmembrane orientation of the cellular isoform of the scrapie prion protein. Mol Cell Biol 1987; 7: 914–20. 12. Hegde RS, Tremblay P, Groth D, et al. Transmissible and genetic prion diseases share acommon pathway of neurodegeneration. Nature 1999; 402: 822–6. 13. Stewart RS, Drisaldi B, Harris DA. Atransmembrane form of the prion protein contains an uncleaved signal peptide and is retained in the endoplasmic reticulum. Mol Biol Cell 2001; 12: 881–9. 14. Kim SJ, Rahbar R, Hegde RS. Combinatorial control of prion protein biogenesis by the signal sequence and transmembrane domain.J Biol Chem 2001; 276: 26132–40. 15. Hegde RS, Voigt S, Lingappa VR. Regulation of protein topology by trans-acting factors at the endoplasmic reticulum. Mol Cell 1998; 2: 85–91. 16. Coux O, Piechaczyk M. Le systèmeubiquitine/protéasome: un ensemble (de) complexe(s) pour dégrader les protéines. Med Sci 2000; 16: 623–9. 17. Zanusso G, Petersen RB, Jin T, et al. Proteasomal degradation and N-terminal protease resistance of the codon 145 mutant prion protein. J Biol Chem 1999; 274: 23396–404. 18. Yedidia Y, Horonchik L, Tzaban S, Yanai A, Taraboulos A. Proteasomes and ubiquitin are involved in the turnover of the wild-type prion protein. EMBO J 2001; 20: 5383–91. 19. Pfeifer K, Bachmann M, Schroder HC, Forrest J, Muller WE. Kinetics of expression of prion protein in uninfected and scrapie- infected N2a mouse neuroblastoma cells. Cell Biochem Funct 1993;11 : 1–11. 20. Jaegly A, Mouthon F, Peyrin JM, et al. Search for a nuclear localization signal in the prion protein. Mol Cell Neurosci 1998; 11: 127–33. 21. Rybner C, Finel-Szermanski S, Felin M, et al. The cellular prion protein: a new partner of the lectin CBP70 in the nucleus of NB4 human promyelocytic leukemia cells. J Cell Biochem 2002;84: 408–19. 22. Gilch S, Winklhofer KF, Groschup MH, et al. Intracellular re-routing of prion protein prevents propagation of PrP (Sc) and delays onset of prion disease. EMBO J 2001; 20: 3957–66. 23. Lehmann S, Milhavet O, Mange A. Trafficking of the cellular isoform of the prion protein. Biomed Pharmacother 1999; 53: 39–46. 24. Madore N, Smith KL, Graham CH, et al. Functionally different GPI proteins are organized in different domains on the neuronal surface. EMBO J 1999; 18: 6917–26. 25. Shyng SL, Heuser JE, Harris DA. A glycolipid-anchored prion protein is endocytosed via clathrin-coated pits. J Cell Biol 1994; 125: 1239–50. 26. Shyng SL, Moulder KL, Lesko A, Harris DA. The N-terminal domain of a glycolipid-anchored prion protein is essential for its endocytosis via clathrin-coated pits. J Biol Chem 1995; 270: 14793–800. 27. Shyng SL, Huber MT, Harris DA. A prion protein cycles between the cell surface and an endocytic compartment in cultured neuroblastoma cells. J Biol Chem 1993; 268: 15922–8. 28. Vincent B, Paitel E, Frobert Y, et al. Phorbol ester-regulated cleavage of normal prion protein in HEK293 human cells and murine neurons. J Biol Chem 2000; 275: 35612–6. 29. Vincent B, Paitel E, Saftig P, et al. The disintegrins ADAM10 and TACE contribute to the constitutive and phorbol ester-regulated normal cleavage of the cellular prion protein. J Biol Chem 2001; 276: 37743–6. 30. Chen SG, Teplow DB, Parchi P, et al. Truncated forms of the human prion protein in normal brain and in prion diseases. J Biol Chem 1995; 270: 19173–80. 31. McMahon HE, Mange A, Nishida N, et al. Cleavage of the amino-terminus of the prion protein by reactive oxygen species. J Biol Chem 2001; 276: 2286–91. 32. Bueler H, Aguzzi A, Sailer A, et al. Mice devoid of PrP are resistant to scrapie. Cell 1993; 73: 1339–47. 33. Bueler H, Fischer M, Lang Y, et al. Normal development and behaviour of mice lacking the neuronal cell-surface PrP protein. Nature 1992; 356: 577–82. 34. Manson JC, Clarke AR, Hooper ML, et al. 129/Ola mice carrying a null mutation in PrP that abolishes mRNA production are developmentally normal. Mol Neurobiol 1994; 8: 121–7. 35. Sakaguchi S, Katamine S, Nishida N, et al. Loss of cerebellar Purkinje cells in aged mice homozygous for a disrupted PrP gene. Nature 1996; 380: 528–31. 36. Moore RC, Lee IY, Silverman GL, et al. Ataxia in prion protein (PrP)-deficient mice is associated with upregulation of the novel PrP-like protein doppel. J Mol Biol 1999; 292: 797–817. 37. Hornshaw MP, McDermott JR, Candy JM, Lakey JH. Copper binding to the N-terminal tandem repeat region of mammalian and avian prion protein: structural studies using synthetic peptides. Biochem Biophys Res Commun 1995; 214: 993–9. 38. Viles JH, Cohen FE, Prusiner SB, et al. Copper binding to the prion protein: structural implications of four identical cooperative binding sites. Proc Natl Acad Sci USA 1999; 96: 2042–7. 39. Kramer ML, Kratzin HD, Schmidt B, et al. Prion protein binds copper within the physiological concentration range. J Biol Chem 2001; 276: 16711–9. 40. Jackson GS, Murray I, Hosszu LL, et al. Location and properties of metal-binding sites on the human prion protein. Proc Natl Acad Sci USA 2001; 3 : 3. 41. Brown DR, Qin K, Herms JW, et al. The cellular prion protein binds copper in vivo. Nature 1997; 390: 684–7. 42. Pauly PC, Harris DA. Copper stimulates endocytosis of the prion protein. J Biol Chem 1998; 273: 33107–10. 43. Waggoner DJ, Drisaldi B, Bartnikas TB, et al. Brain copper content and cuproenzyme activity do not vary with prion protein expression level. J Biol Chem 2000; 275: 7455–8. 44. Brown DR, Besinger A. Prion protein expression and superoxide dismutase activity. Biochem J 1998; 334: 423–9. 45. Brown DR, Wong BS, Hafiz F, et al. Normal prion protein has an activity like that of superoxide dismutase. Biochem J 1999; 344: 1–5. 46. Milhavet O, McMahon HE, Rachidi W, et al. Prion infection impairs the cellular response to oxidative stress. Proc Natl Acad Sci USA 2000; 97: 13937–42. 47. Gabus C, Derrington E, Leblanc P, et al. The prion protein has RNA binding and chaperoning properties characteristic of nucleocapsid protein NCP7 of HIV-1. J Biol Chem 2001; 276: 19301–9. 48. Gauczynski S, Peyrin JM, Haik S, et al. The 37-kDa/67-kDa laminin receptor acts as the cell-surface receptor for the cellular prion protein. EMBO J 2001; 20: 5863–75. 49. Mouillet-Richard S, Ermonval M, Chebassier C, et al. Signal transduction through prion protein. Science 2000; 289: 1925–8. 50. Ostlund P, Lindegren H, Pettersson C, Bedecs K. Altered insulin receptor processing and function in scrapie-infected neuroblastoma cell lines. Brain Res Mol Brain Res 2001; 97: 161–70. 51. Lee IY, Westaway D, Smit AF,et al. Complete genomic sequence and analysis of the prion protein gene region from three mammalian species. Genome Res 1998; 8: 1022–37. 52. Baybutt H, Manson J. Characterisation of two promoters for prion protein (PrP) gene expression in neuronal cells. Gene 1997; 184: 125–31. 53. Martins VR, Graner E, Garcia-Abreu J, et al. Complementary hydropathy identifies a cellular prion protein receptor. Nat Med 1997; 3: 1376–82. 54. Yehiely F, Bamborough P, Da Costa M, et al. Identification of candidate proteins binding to prion protein. Neurobiol Dis 1997; 3: 339–55. 55. Kurschner C, Morgan JL. The cellular prion protein (PrP) selectively binds to Bcl-2 in the yeast two-hybrid system Brain Res Mol Brain Res 1995; 30: 165–8 56. Oesch B, Teplow DB, Stahl N, et al. Identification of cellular proteins binding to the scrapie prion protein. Biochemistry 1990; 29 : 5848–55. 57. Spielhaupter C, Schatzl HM. PrPC directly interacts with proteins involved in signaling pathways. J Biol Chem 2001; 276: 44604–12. 58. Edenhofer F, Rieger R, Famulok M, Wendler W, Weiss S, Winnacker EL. Prion protein PrPc interacts with molecular chaperones of the Hsp60 family. J Virol 1996; 70: 4724–8. 59. Graner E, Mercadante AF, Zanata SM, et al. Cellular prion protein binds laminin and mediates neuritogenesis. Brain Res Mol Brain Res 2000; 76: 85–92. 60. Rieger R, Edenhofer F, Lasmezas CI, Weiss S. The human 37-kDa laminin receptor precursor interacts with the prion protein in eukaryotic cells. Nat Med 1997; 3 : 1383–8. 61. Schmitt-Ulms G, Legname G, Baldwin MA, et al. Binding of neural cell adhesion molecules (N-CAMs) to the cellular prion protein. J Mol Biol 2001; 314: 1209–25 62. Fischer MB, Roeckl C, Parisek P, et al. Binding of disease-associated prion protein to plasminogen. Nature 2000; 408 : 479–83. 63. Weiss S, Proske D, Neumann M, et al. RNA aptamers specifically interact with the prion protein PrP. J Virol 1997; 71 : 8790–7. 64. Ma J, Lindquist S. Conversion of PrP to a self-perpetuating PrPSc-like conformation in the cytosol. Science 2002 (online). 65. Ma J, Wollmann R, Lindquist S. Neurotoxicity and neurodegeneration when PrP accumulates in the cytosol. Science 2002 (online). 66. Beranger F, Mangé A, Goud B, Lehmann S. Stimulation of PrPC retrograde transport toward the endoplasmic reticulum increases accumulation of PrPSc in prion-infected cells. J Biol Chem 2002; 277 : 38972–7. |