| |
| Med Sci (Paris). 2002 August; 18(8-9): 831–840. Published online 2002 August 15. doi: 10.1051/medsci/20021889831.Approches génétiques de la mort cellulaire programmée : succès et questions Myriam Adam,1 Jean-Pierre Levraud,2 and Pierre Golstein1* 1Centre d’Immunologie de Marseille-Luminy, Cnrs-Inserm, Université Méditerranée, Parc Scientifique de Luminy, Case 906, 13288 Marseille Cedex 9, France 2Institut Pasteur, 25, rue du Docteur Roux, 75015 Paris, France |
Imaginez une cellule, et, dans cette cellule, un nuage qui contient les différentes voies moléculaires et les organites impliqués dans la « mort cellulaire programmée ». Différents signaux d’origine extracellulaire pénètrent ce nuage, le destin cellulaire, mort ou survie, s’y décide, cette décision aboutissant éventuellement à la mort de la cellule. Comment déterminer ce qui se passe à l’intérieur du nuage ? Comme souvent dans l’analyse d’un phénomène complexe, la voie royale est la voie génétique, que renforcent maintenant les progrès de la biologie moléculaire. Nous traiterons ici des molécules et des mécanismes identifiés par des moyens génétiques utilisés en première intention (obtention et analyse de mutants altérant les mécanismes de mort cellulaire), par opposition à ceux qui sont mis en évidence secondairement par leurs homologies interspécifiques, le plus souvent en consultant des banques de données à l’aide d’outils tels que BLAST1. Les approches génétiques classiques ont été essentielles à la compréhension des processus de mort cellulaire faisant intervenir les caspases, et commencent à être utilisées dans la recherche de ceux qui contrôlent la mort indépendamment des caspases. Nous constaterons - ce qui n’allait pas forcément de soi -que les découvertes primaires ont été fonction de l’organisme modèle utilisé, pour la mise en évidence de telle ou telle voie de mort cellulaire et des molécules impliquées soit dans les voies de signalisation conduisant à ce processus, soit dans l’exécution de la mort cellulaire proprement dite. Nous discuterons brièvement les raisons de certains de ces résultats. |
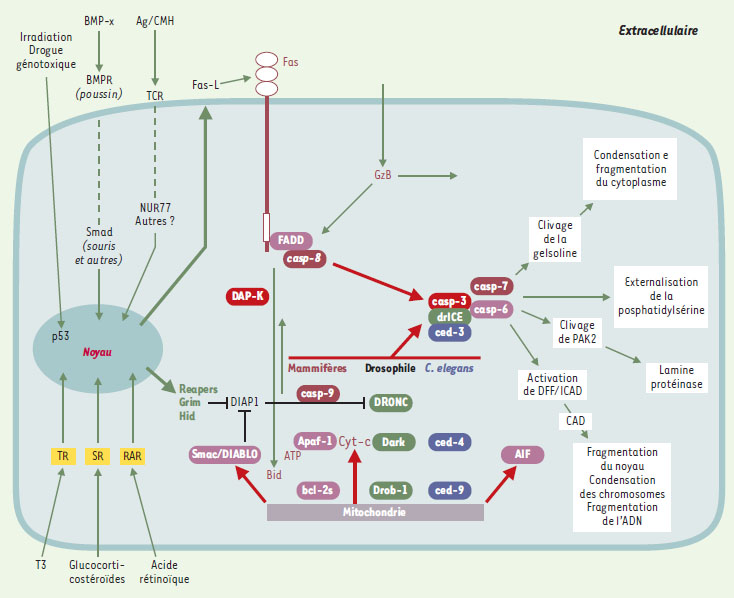
Comment définir la mort cellulaire Quelques définitions sont indispensables. Les processus de mort cellulaire peuvent être programmés ou non. Une mort cellulaire non programmée, dont la survenue est accidentelle et résulte parfois d’une lésion primaire de la membrane externe de la cellule, pourra être la conséquence de l’exposition de la cellule à un produit toxique. La mort cellulaire qui survient lors du développement embryonnaire est « programmée » à la fois quant à sa survenue dans l’organisme, et quant à son déroulement dans la cellule qui meurt. Cependant, programmations de survenue et de déroulement peuvent être dissociées : en pathologie, par exemple lors d’épisodes infectieux ou ischémiques, la mort cellulaire peut survenir de façon anormale, mais se dérouler selon un processus normal. On pourrait ainsi inclure dans la classification des pathologies un nouveau chapitre traitant des « maladies de survenue de la mort cellulaire », qui demanderaient une gestion thérapeutique adaptée. Les morts cellulaires peuvent être hétérogènes également quant à leur morphotype. Le terme apoptose [1] désigne un tableau morphologique associant condensation cytoplasmique et chromatinienne, fragmentation de l’ADN, externalisation de la phosphatidylsérine membranaire ; la synthèse de macromolécules est parfois requise, et ces changements sont rapidement suivis de la phagocytose des cellules mortes. Il existe d’autres tableaux morphologiques de mort cellulaire, même chez les mammifères, comme la mort nécrotique [2, 3] ou la mort par vacuolisation [4–6]. Les morts cellulaires peuvent différer aussi quant à leur mécanisme, selon qu’il fait ou non intervenir des cystéine protéases particulières appelées caspases (→) (Figure 1). Au total, la plupart des morts cellulaires lors du développement embryonnaire sont programmées pour leur survenue et pour leur déroulement dans les cellules qui meurent, et incluent une activation des caspases conduisant à un démantèlement des structures cellulaires, se traduisant morphologiquement par un aspect d’apoptose. Cependant, les relations entre morphologie, mécanismes et programme de survenue peuvent être moins schématiques qu’il n’est classiquement considéré. Par exemple, la mort nécrotique peut n’être pas qu’accidentelle, elle peut survenir également au cours du développement, au moins chez certains mutants chez la souris [7]. Il s’agit là de la survenue normale d’une mort dont le déroulement, lui, est, sinon anormal, du moins inattendu. (→) m/s 1998, n°1, p. 9  | Figure 1. Quelques voies gouvernant la mort cellulaire dépendante des caspases. Dans la partie droite de la figure, en aval de l’activation des caspases, le clivage des substrats de caspases conduit au démantèlement systématique de structures cellulaires (modifications cellulaires inscrites sur fond blanc), ce qui se traduit par un morphotype apoptotique. Dans la partie gauche de la figure, en amont de l’activation des caspases, une série complexe d’étapes conduit de signaux extracellulaires à l’activation des caspases, par l’intermédiaire de récepteurs, puis de facteurs de transcription, et enfin de la synthèse de molécules intermédiaires telles reaper. La partie centrale de la figure est le stade d’intégration, avec activation des caspases. Des caspases effectrices, comme la caspase 3, sont activées par des caspases initiatrices. Celles-ci incluent la caspase 8, dont l’activation est la conséquence de la constitution d’un complexe moléculaire après engagement de récepteurs de la famille TNFR/Fas, et la caspase 9, dont l’activation est la conséquence de la constitution d’un apoptosome comprenant entre autres APAF-1. Dans cette partie centrale de la figure, les molécules de C. elegans, de dro-sophile ou de mammifères impliquées dans la mort cellulaire sont indiquées en lettres blanches sur fonds respectivement bleu (C. elegans), vert (drosophile), rose, rouge et violet (mammifères). La conservation semble maximale dans la zone « cœur » du mécanisme, les variations interspécifiques semblent prédominer dans les régions de contrôle en amont. Soulignons que ce schéma n’inclut pas les morts indépendantes des caspases. TR : thyroid hormone receptor ; SR : steroid receptor ; RAR : retinoic acid receptor. |
|
Au commencement moléculaire était le ver Quelle était la compréhension de la mort cellulaire dans les années 1980 ? Malgré la systématisation du morphotype majeur de mort cellulaire qu’est l’apoptose [1], faisant pressentir la programmation de son déroulement, la mort cellulaire n’était encore qu’un phénomène. Une respectabilité moléculaire lui a été conférée par la découverte de son contrôle génétique chez C. ele-gans. R. Horvitz et ses collègues ont obtenu dans cet organisme des mutations pouvant prévenir la mort cellulaire : des cellules qui mouraient lors du développement du ver sauvage, survivaient chez ces mutants. Cela démontrait l’intervention de certaines molécules, altérées chez ces mutants, indispensables pour qu’il y aitmort cellulaire. La conclusion, très importante, est que la mort cellulaire n’est pas une détérioration passive comme on l’avait souvent cru jusqu’alors, mais une fonction active, une voie de différenciation terminale de la cellule, nécessitant l’intervention de molécules spécialisées. Dans les années 1970, l’étude du nématode C. elegans avait déjà conduit le laboratoire de Sidney Brenner à Cambridge (Royaume-Uni), à l’identification d’une mort cellulaire par l’examen direct en microscopie Nomarski2. Une mutagenèse chimique, s’appuyant sur les avantages génétiques de cet organisme, permit à J.E. Sulston d’identifier, dès 1976, une mutation appelée nuc-1 empêchant la fragmentation de l’ADN dans des cellules mourantes [8], puis les mutations ced-1 et ced-2, qui interférent avec la phagocytose des cellules mortes [9] (→). R. Horvitz, dans ce même laboratoire, puis dans son propre laboratoire à Cambridge (MA, USA), découvrit en particulier les mutations ced-3 et ced-4 [10, 11]. (→) m/s 2002, n°8-9, p. 853 Les protéines ced-3 et ced-4 sont nécessaires à la mort cellulaire qui survient au cours du développement, introduisant, comme nous l’avons souligné plus haut, la notion que la mort cellulaire est un processus actif. Chez C. elegans, une approche de cartographie par RFLP (restriction fragment length polymorphism) et marche chromosomique a permis de déterminer que ced-3 est une cystéine protéase, dont un homologue chez la souris est la cystéine protéase ICE [12, 13], ce qui a conduit à la description d’une série de membres de cette même sous-famille de cystéine protéases appelées caspases [14]. L’activation des caspases est sous le contrôle de ced-4 chez C. elegans et de son homologue Apaf-1 chez les mammifères [15]. Du laboratoire de R. Horvitz provient la description, non seulement de ced-3 et de ced-4 comme molécules effectrices de la mort cellulaire chez C. elegans, mais aussi de ced-9 comme molécule contrôlant la survenue de la mort [16]. Ced-9 appartient à la famille Bcl-2, dont un membre chez l’homme, découvert dans des cellules tumorales porteuses d’une translocation chromosomique, avait été identifié comme gène de survie dès 1988 en Australie [17]. D’autres molécules, impliquées dans la mort cellulaire, les voies de signalisation qui y conduisent, ou la phagocytose des cellules post-mortem ont été découvertes chez C. elegans [18, 19]. Cette approche constitue par ailleurs un excellent exemple de recherche fondamentale, appliquant à un organisme, alors relativement obscur, une approche génétique classique, et qui devrait maintenant conduire dans un très proche avenir à des applications très importantes dans le domaine médical. Les recherches sur C. elegans ont abouti à un dogme mécanistique quant au processus de mort cellulaire, qui a considérablement influencé les recherches ultérieures. L’aspect positif est que ce modèle a conduit à la résolution, chez les animaux, de mécanismes de mort cellulaire dépendants des caspases ; cependant, curieusement, le ver s’avère parfois un modèle plus particulier qu’universel [20] : aucune libération de cytochrome c par les mitochondries n’a pu y être démontrée, ced-4 est plus court que ses homologues mammifères connus, il n’y a pas été identifié d’IAP (inhibitor of apoptosis proteins) ni de « voie récepteur »… Mais ce succès a eu un effet pervers : l’éclairage du « nuage » mort cellulaire évoqué dans l’introduction par les seules données obtenues chez C. elegans a conduit très longtemps à laisser dans l’ombre d’autres aspects, biaisant ainsi les recherches. La mise en évidence précoce chez C. elegans de l’ensemble ced-9-ced-4-ced-3 a fait de cet organisme pendant longtemps, le gardien du dogme d’une mort cellulaire programmée apoptotique et dépendante des caspases. Intéressant retour des choses, chez cet organisme sont maintenant étudiés, aussi par des méthodes génétiques, certains mécanismes de mort non apoptotique et indépendante des caspases [21, 22]. |
Puis vinrent : la mouche… Assez curieusement, la drosophile n’a pas été mise à contribution très tôt pour l’élucidation des mécanismes de mort cellulaire. Ce n’est qu’au début des années 1990 (malgré des observations éparses, dont les références sont citées dans [23]) qu’une approche systématique de la mort cellulaire a été entreprise dans cet organisme. Le laboratoire de H. Steller au Massachusetts Institute of Technology (MA, USA) a alors démontré qu’un tel processus intervient lors du développement de la drosophile, et que les cellules mortes présentent les caractères morphologiques d’une apoptose, facilement identifiée dans de nombreuses régions après coloration à l’acridine orange de l’embryon entier [24], et faisant intervenir les caspases. L’existence, chez la drosophile, d’une collection de souches présentant un spectre très étendu de délétions chromosomiques a permis d’identifier certaines des molécules en jeu. Un criblage de ces souches utilisant le marquage à l’acridine orange a été entrepris à la recherche d’embryons qui ne présenteraient pas de cellules mortes. C’est le cas dans une souche présentant une délétion chromosomique appelée H99. La région chromosomique correspondant à cette délétion porte un gène appelé reaper [23], ainsi que deux autres gènes, Hid et grim [25, 26], tous impliqués dans le contrôle de la mort cellulaire. Les molécules codées par ces trois gènes présentent une certaine homologie de séquence de leur extrémité N-terminale. C’est par ce domaine N-terminal qu’elles peuvent s’associer à des molécules de type IAP qui, elles, inhibent l’activation des caspases, ce qui leur confère une fonction anti-apoptotique. C’est peut-être par leur capacité d’inhiber ces molécules IAP anti-apoptotiques que reaper, hid et grim peuvent être pro-apoptotiques [27]. Curieusement, on ne connaît d’homologues structuraux de reaper, hid et grim ni chez C. elegans ni chez les mammifères. Chez ces derniers, la molécule appelée Smac/DIABLO [28, 29], et la sérine protéase Omi/HtrA2 [30], libérées par les mitochondries et qui se lient aux IAP, semblent jouer un rôle homologue à celui de reaper/hid/grim. Certes, des composants des mécanismes de mort cellulaire caractérisés chez les animaux existent chez la drosophile, en particulier des caspases et des membres de la famille Bcl-2 [31], mais ces données sont le fruit d’une recherche in silico d’homologues et non pas d’un abord génétique direct. Par ailleurs, un homologue du domaine de mort FADD [32] et une molécule à death effector domain [33] ont également été décrits chez la drosophile, ce qui soulève l’hypothèse intéressante, voire provocante, d’une possible existence chez la drosophile d’une voie de mort relayée par des récepteurs. Au total, l’approche génétique de première intention a bien identifié chez la drosophile des gènes impliqués dans la mort cellulaire ; mais, curieusement, les gènes ainsi identifiés ne semblent pas être conservés phylogénétiquement dans l’ensemble du règne animal, et ont plus un rôle de contrôle du mécanisme de mort cellulaire qu’un rôle direct dans le processus de mort proprement dit. Ce sont d’autres approches qui ont conduit à l’identification, chez la drosophile, des familles moléculaires contrôlant le mécanisme de la mort cellulaire apoptotique dépendante des caspases telle qu’on l’observe dans le règne animal. |
Les mammifères, qui ne possèdent pas les avantages génétiques de C. elegans ou de la drosophile, ne se prêtent pas facilement à la recherche, par une approche génétique classique in vivo, de molécules impliquées dans la mort cellulaire. Certaines approches génétiques sont néanmoins possibles in vitro, à partir de lignées cellulaires, en parant aux inconvénients de la diploïdie par des méthodes produisant l’équivalent de mutations dominantes. Ainsi, A. Kimchi et ses collaborateurs à l’Institut Weizmann (Rehovot, Israël) ont transfecté des cellules humaines HeLa avec une banque d’ADNc de cellules HeLa. Les ADNc de cette banque sont insérés en orientation antisens (leur expression après transfection devrait donc avoir un effet dominant négatif sur l’expression endogène du produit du gène ciblé) dans un vecteur épisomique dérivé de l’EBV (Epstein-Barr virus), ce qui assure un niveau d’expression suffisant. Après transfection, ces cellules sont exposées à une forte concentration d’interféron γ (IFNγ) qui normalement entraîne leur mort apoptotique. Certaines cellules s’avèrent résistantes. L’ADN épisomique en est extrait, le plasmide isolé et l’insertion d’ADNc identifiée. Un tel ADNc, dont la séquence antisens bloque la mort cellulaire, identifie donc un gène codant pour une molécule nécessaire à la mort cellulaire. Plusieurs gènes de ce type et leurs produits ont été détectés par cette approche, et une des plus remarquables et des plus étudiées de ces molécules est la DAP-kinase [34]. Il s’agit d’une protéine kinase ayant une spécificité sérine/thréonine, comprenant, outre le domaine kinase, huit répétitions ankyrine, une région régulatrice sous le contrôle de la calmoduline et un death domain. Cette DAP-kinase est associée au cytosquelette, plus précisément aux microfilaments [34, 35]. Son expression semble diminuée dans certaines tumeurs à fort pouvoir métastatique [36] et elle s’oppose à la transformation induite par un oncogène, en activant un point de contrôle du cycle cellulaire contrôlé par la boucle p19ARF/p53 [37]. De façon assez inattendue, par l’intermédiaire de son death domain, elle est nécessaire à la mort cellulaire induite via Fas ou le TNF-R1 (tumor necrosis factor receptor I)[38], et aussi à la mort cellulaire induite par le TGF-β (transforming growth factor β) qui entraîne son activation transcriptionnelle [39] ; dans ces deux cas, l’activité DAP-kinase nécessaire à cette mort s’exerce en amont des événements mitochondriaux. La façon dont cette DAP-kinase s’articule avec les autres molécules, définies par d’autres approches, intervenant dans la mort cellulaire, en particulier les intermédaires de la voie « récepteur », n’est pas évidente. Il s’agit là d’un bon exemple d’une molécule clairement importante dans la mort cellulaire, identifiée par une approche génétique inhabituelle, et à laquelle il est actuellement difficile d’attribuer une place précise dans le réseau des molécules dont l’intervention dans le processus de mort est connue. Cela suggère l’existence de molécules et de voies de mort encore insoupçonnées. Une autre approche « dominante », contournant la difficulté liée au caractère diploïde des cellules de mammifères, consiste à transfecter dans les cellules un vecteur, par exemple un rétrovirus, contrôlé par un promoteur fort capable d’activer un gène au voisinage du point d’insertion de ce vecteur dans le génome. Si le gène en question est anti-apoptotique, les cellules dans lesquelles son expression est induite seront résistantes à la mort cellulaire et pourront ainsi être sélectionnées. C’est par une telle approche qu’une fonction anti-apoptotique majeure de Bcl-X dans certaines circonstances a été identifiée [40]. Rappelons aussi que des mutations non expérimentales survenant chez les mammifères ont été à l’origine de la caractérisation de certaines molécules impliquées dans la mort cellulaire, ou de la confirmation de leur rôle soupçonné sur d’autres arguments. Par exemple, chez la souris, la mutation spontanée lpr [41, 42] correspond à une mutation de la molécule Fas découverte lors du criblage d’anticorps monoclonaux [43, 44]. Et, comme nous l’avons indiqué plus haut, notre connaissance de la famille Bcl-2 dérive à la fois d’une mutagenèse expérimentale chez C. elegans [16] et de la caractérisation d’une translocation chromosomique pathologique chez l’homme [17]. |
…et des organismes hors du règne animal Rappelons aussi qu’un nuage peut en cacher un autre. Il existe dans des cellules de mammifères un, voire des mécanismes de mort cellulaire qui ne répondent pas aux critères de définition d’une apoptose et qui ne font pas intervenir les caspases (voir par exemple [7, 45]). La caractérisation de ces morts « indépendantes des cas-pases » a rarement été explorée sous l’angle génétique chez les animaux (voir cependant [21, 22]). Or, il se trouve que, chez tous les êtres vivants, et donc hors du règne animal, existent des phénomènes de mort cellulaire. Comme on ne connaît pas de caspases stricto sensu en dehors du règne animal [46], il faut nécessairement postuler l’intervention d’autres mécanismes, indépendants des caspases. Ceuxci peuvent être identifiés dans des organismes différents des animaux (plantes, champignons, Dictyostelium) et les résultats peuvent être extrapolés aux animaux s’il y a conservation phylogénétique. La mort cellulaire chez les plantes suscite maintenant beaucoup d’intérêt [47–51], en particulier pour des raisons agro-économiques. Les molécules caractérisées interviennent surtout dans les voies de signalisation induites lors de la réaction d’hypersensibilité à des pathogènes, et liées à des gènes de résistance [52–54], et elles concernent moins les mécanismes de la mort proprement dite, mais cette situation peut évoluer très vite. Beaucoup de ces travaux sont en cours dans le modèle végétal Arabidopsis thaliana, mais d’autres modèles, comme l’algue Volvox [55], sont particulièrement tentants. La levure Saccharomyces cerevisiae pose un problème particulier. Il n’est pas évident qu’il existe une mort cellulaire spontanée chez la levure dans le sens habituel (la « sénescence » d’une levure, après de multiples bourgeonnements, semble être d’une autre nature [56]). Cependant, la surexpression de molécules « de mort » actives chez les mammifères, comme Bax, induit une mort cellulaire chez la levure (et cette utilisation de la levure comme tube à essais peut être analytiquementutile [57–59]). La mort de cellules de levure peut aussi survenir dans certaines conditions génétiques [60] ou d’agression oxydative [61]. Dans ces conditions, curieusement, les cellules de levure qui meurent peuvent alors présenter des signes [60] voisins de ceux qui sont observés au cours de l’apoptose de cellules animales, alors même que la levure ne possède pas de gènes codant pour des caspases (certains de ces signes peuvent cependant être communs à toutes les circonstances de démantèlement cellulaire, voir la discussion dans [62]). L’isolement du gène Bl-1 représente une des rares circonstances, voire la seule, où une approche génétique chez la levure (il s’agissait de la recherche de mutants résistants à la surexpression de Bax), a abouti à l’identification d’une molécule semblant interférer avec la mort cellulaire dans la levure et dans d’autres organismes [58]. Une approche génétique intéressante des mécanismes de la mort cellulaire hors du règne animal a été menée sur les champignons filamenteux, dont un exemple est Podospora anserina. Ces organismes sont les seuls qui soient naturellement capables de fusion cellulaire, aboutissant à la constitution d’hétérocaryons. Dans les situations d’incompatibilité, il y a mort des hétérocaryons, ce qui se traduit dans chaque cellule qui meurt par une vacuolisation importante, et par un phénomène de barrage lors de la rencontre de deux colonies incompatibles [63]. La viabilité des hétérocaryons est génétiquement contrôlée par des locus appelés het [64]. Les gènes correspondants, clonés par des approches classiques, s’avèrent hétérogènes dans leur séquence et leurs éventuelles fonctions. Une situation particulièrement intéressante est celle du gène het-s [63], dont le produit a un comportement de prion [65, 66]. Des approches dérivées ont consisté à isoler, par des méthodes génétiques, des gènes suppresseurs s’opposant à cette incompatibilité comme mod-E1, un membre de la famille HSP90 [67], ou, par approche soustractive, des gènes induits lors d’une réaction d’incompatibilité [68]. Ces efforts ont ainsi permis d’identifier un certain nombre de gènes responsables de la mort, mais, comme précédemment, intervenant plus au niveau des voies de signalisation conduisant à la mort, que dans le mécanisme d’exécution proprement dite de cette mort. Il est à noter que Podospora subit un autre phénomène, peut-être apparenté à la mort cellulaire, appelé sénescence (mais qui diffère de la sénescence chez la levure), et dont le déterminisme semble mitochondrial [69–71]. Clairement, l’approche génétique chez Podospora devrait continuer à fournir des informations de portée générale sur l’induction et le mécanisme de la mort cellulaire. Un autre organisme hors du règne animal dont l’analyse promet d’être féconde pour l’étude génétique de mécanismes de la mort cellulaire est le protiste Dictyostelium discoideum. Cet organisme se multiplie sous forme unicellulaire (« pseudo-amibes ») lorsque les conditions nutritionnelles sont favorables. En situation de carence, les cellules s’agrègent, se différencient et subissent une rapide et frappante morphogenèse : l’agrégat initial se transforme en une limace migratoire, puis en une structure verticale formée d’une tige qui supporte une masse de spores. Les cellules de la tige subissent un processus de mort cellulaire, qui est donc lié au développement, par définition. L’étude, par des moyens génétiques, du mécanisme de cette mort est facilitée par les caractéristiques du génome de ce protiste : il est à peu près 100 fois plus petit que celui des eucaryotes supérieurs, et son séquençage est en cours d’achèvement ; il est haploïde, ce qui facilite l’obtention et la sélection de mutants de gènes impliqués dans la mort cellulaire ; ceux-ci peuvent ensuite être identifiés grâce aux outils génétiques disponibles chez Dictyostelium [72, 73]. Du fait de la séparation temporelle entre pousse végétative et développement, de tels mutants, qui ne touchent que la phase développementale, ne seront pas affectés en condition végétative, et se comporteront donc comme des mutants conditionnels [72]. On peut déclencher in vitro une différenciation sans morphogenèse [74], permettant ainsi l’obtention des cellules de Dictyostelium isolées et mourantes, donc faciles à identifier et à étudier. Il s’agit donc d’analyser la mort cellulaire programmée chez un organisme assez distant phylogénétiquement des organismes modèles classiquement utilisés pour cette analyse, et possédant des avantages génétiques au moins aussi marqués. Cette prise de distance pourrait aider à définir des mécanismes de base de la mort cellulaire, plus accessibles chez cet organisme que chez d’autres. La mort cellulaire chez Dictyostelium est vacuolaire, avec des lésions membranaires tardives, mais sans la fragmentation en échelle de l’ADN [75] (voir aussi [76, 77]), et sans l’intervention de caspases conventionnelles [78]. La recherche de gènes gouvernant la mort cellulaire chez Dictyostelium s’est faite d’abord par mutagenèse insertionnelle, dans sa variante REMI (restriction-enzyme mediated insertional)[73], appliquée à des cellules de Dictyostelium en monocouche dont on peut induire la mort. Un vecteur est électroporé dans ces cellules de Dictyostelium en pousse végétative et, en s’insérant dans le génome de rares cellules, inactive un gène nécessaire à la mort. Les cellules transfectées sont ensuite transférées dans un milieu carencé, ce qui induit leur mort ; les seules qui survivent étant celles dans lesquelles un gène nécessaire à la mort a été inactivé par l’insertion du vecteur. Ces cellules survivantes vont se multiplier après leur transfert dans un milieu riche, et le vecteur inséré, ainsi que les séquences flanquantes correspondantes, en est extrait par plasmid rescue [79, 80]. Plusieurs mutants indépendants relativement résistants à la mort cellulaire ont ainsi été isolés. Parmi ceux-ci, certains s’avèrent particulièrement intéressants : DDM16, qui affecte les capacités de survie en milieu carencé seul, et surtout DDM4, où la molécule mutée, appelée DeliriumA, contrôle l’expression de la protéine kinase A (PKA) en inhibant l’instabilité observée dans la région codante de la PKA elle-même. Dans ce modèle aussi, les approches génétiques de première intention n’ont pour l’instant identifié que des molécules impliquées dans la signalisation de la mort cellulaire, et non dans le mécanisme de mort. |
L’analyse génétique des mécanismes moléculaires de la mort cellulaire a fourni des renseignements essentiels en ce qui concerne la mort cellulaire apoptotique dépendante des caspases, et est actuellement utilisée dans l’étude d’autres voies de mort cellulaire. En ce qui concerne l’apoptose dépendante des caspases, la recherche dans C. elegans de mutants de mort cellulaire par une approche génétique classique a été couronnée de succès, révélant certains des gènes-clés du mécanisme de base de cette mort. Cependant, C. elegans constitue une exception presqu’aussi éclatante, non seulement en raison des quelques particularités rappelées plus haut quant aux molécules en jeu, mais surtout parce que c’est, semble-t-il, le seul organisme (voir cependant [40]) dans lequel l’approche génétique classique a permis d’identifier par mutagenèse des molécules impliquées dans le mécanisme proprement dit de la mort cellulaire. On pourrait a priori s’attendre à obtenir plus de mutants touchant les voies de signalisation que de mutants altérant les effecteurs directs de la mort : en effet, de multiples molécules et de plus longues étapes moléculaires constituent les voies de signalisation, qui toutes convergent vers un mécanisme moléculaire « central » de la mort. Le dérèglement de ce dernier est plus susceptible d’être létal très tôt dans le développement, et s’il y a redondance, elle pourra être plus fortement sélectionnée qu’une redondance des voies de signalisation. Or, cette prédiction d’une obtention préférentielle de mutants de signalisation s’avère exacte pour la plupart des organismes considérés ici, mais pas pour C. elegans. Pourquoi ? Une plus grande avancée des recherches sur C. elegans par rapport aux recherches sur d’autres organismes pourrait l’expliquer et, dans ce cas, cette discordance serait toute provisoire : c’est probablement dans cet organisme que le plus de mutants ont sans doute été criblés, et très peu d’organismes autres que C. elegans (mammifères, drosophile…) ont été étudiés en détail. Reste cependant que la série des mutants ced a été obtenue tôt dans l’histoire de ces criblages chez C. elegans. L’explication de l’obtention préférentielle de mutants du mécanisme de mort proprement dit chez C. elegans pourrait en fait résider dans la combinaison de deux facteurs. D’une part, il existe peu ou pas de redondance moléculaire en ce qui concerne le mécanisme de mort cellulaire chez C. elegans (en particulier ced-3, ced-4, ced-9 sont uniques), et chez ces mutants le phénotype d’absence de mort cellulaire est sans ambiguïté. D’autre part, cet organisme tolère fort bien ces mutations. Une particularité de C. elegans est en effet que la suppression de la mort cellulaire par mutagenèse n’empêche pas la survie quasi normale des vers mutants [11]. Plus précisément, chez C. elegans la mutation de gènes nécessaires à la mort cellulaire semble n’interférer sévèrement ni avec le fonctionnement cellulaire (indiquant que les gènes en question, s’ils sont nécessaires à la mort des cellules, ne le sont pas à leur vie), ni avec celui de l’organisme entier (indiquant que la survie de cellules qui normalement meurent n’empêche pas la survie de l’organisme, à quelques exceptions près liées à des problèmes sans lien avec le développement [81]). La facilité d’obtention d’un phénotype de mort cellulaire et sa bonne tolérance par l’organisme entier contribueraient ainsi à la fréquence relative des mutations de mécanisme de mort cellulaire chez C. elegans. De plus, chez C. elegans, les cellules résistant à la mort, témoignant d’une mutation, sont identifiées par criblage au microscope, après le développement au moins partiel de l’organisme. Il est vraisemblable que des mutants de signalisation affecteraient sévèrement le développement de l’organisme, indépendamment de leur effet sur la mort (cette remarque vaut sans doute également pour tout autre organisme). Au total, chez C. elegans, les mutants de signalisation seraient contre-sélectionnés, alors que les mutants du mécanisme de mort proprement dit sont à la fois détectés et tolérés. Cette catégorie de mutants est donc retrouvée chez cet organisme. Cette observation n’est pas sans implications sur les stratégies d’obtention de mutants touchant les mécanismes de mort dans d’autres organismes. Chez les mammifères, des molécules essentielles à la vie des cellules, comme certaines molécules intervenant dans le cycle cellulaire, semblent aussi nécessaires à la mort cellulaire [82–86]. Les conséquences des mutations de ces molécules seraient en règle non détectables. Est-ce « la fin de l’histoire » ? En d’autres termes, a-ton recensé l’ensemble des types de molécules impliquées dans la mort cellulaire programmée dépendante des caspases ? Le fait que la mort cellulaire ait été étudiée jusqu’à présent dans très peu d’organismes, et que l’étude dans chacun de ces organismes ait souvent conduit à la découverte de nouvelles molécules (impliquées cependant dans la signalisation plus que dans le mécanisme de mort proprement dit), suggère fortement que non. Les molécules « orphelines » ainsi découvertes font suspecter là aussi l’existence de mondes moléculaires encore ignorés. En ce qui concerne les mécanismes moléculaires intervenant dans la mort cellulaire indépendamment des caspases, qui, rappelons-le, représentent toutes les morts cellulaires hors du règne animal, et une partie des morts cellulaires dans ce règne, l’étendue de l’ignorance est notable (voir cependant dans des cellules de mammifères [87–89]). L’abord génétique, peu exploité pour l’instant dans ces morts indépendantes des caspases (chez C. elegans [21, 22] ; et chez Dictyostelium, voir cidessus), peut bénéficier de modèles appropriés dans le règne végétal et chez certains protistes. Il est possible, mais cela reste à prouver, que des mécanismes de mort indépendants des caspases soient conservés à travers les règnes autant que le mécanisme dépendant des caspases l’est à l’intérieur du règne animal. |
Nous remercions pour leur contribution M.F. Luciani et C. Roisin. Les travaux de ce laboratoire cités ici sont soutenus par la dotation récurrente de l’Inserm et du Cnrs au Centre d’Immunologie, par l’Institut Pasteur pour J.P. Levraud, et par l’ARC, le MRT et la Communauté Européenne (ERBFMRX CT97 0153 ; QLG1-CT-1999-00739).
|
Footnotes |
1. Kerr JFR, Wyllie AH, Currie AR. Apoptosis: a basic biological phenomenon with wide-ranging implications in tissue kinetics. Br J Cancer 1972; 26 : 239–57. 2. Searle J, Kerr JFR, Bishop CJ. Necrosis and apoptosis: distinct modes of cell death with fundamentally different significance. Pathol Annu 1982; 17 : 229–59. 3. Fiers W, Beyaert R, Declercq W, Vandenabeele P . More than one way to die: apoptosis, necrosis and reactive oxygen damage. Oncogene 1999; 18 : 7719–30. 4. Clarke PGH. Developmental cell death: morphological diversity and multiple mechanisms. Anat Embryol 1990; 181 : 195–213. 5. Schwartz LM, Smith SW, Jones MEE, Osborne BA. Do all programmed cell deaths occur via apoptosis? Proc Natl Acad Sci USA 1993; 90 : 980–4. 6. Zakeri Z, Bursch W, Tenniswood M, Lockshin RA. Cell death: programmed, apoptosis, necrosis, or other ? Cell Death Differ 1995; 2 : 87–96. 7. Chautan C, Chazal G, Cecconi F, Gruss P, Golstein P . Interdigital cell death can occur through a necrotic and caspase-independent pathway. Curr Biol 1999; 9 : 967–70. 8. Sulston JE. Post-embryonic development in the ventral cord of Caenorhabditis elegans. Philos Trans R Soc Lond B Biol Sci 1976; 275 : 287–97. 9. Hedgecock EM, Sulston JE, Thomson JN. Mutations affecting programmed cell death in the nematode Caenorhabitis elegans. Science 1983; 220 : 1277–9. 10. Horvitz HR, Sternberg PW, Greenwald IS, Fixsen W, Ellis HM. Mutations that affect neural cell lineages and cell fates during the development of the nematode Caenorhabditis elegans. Cold Spring Harb Symp Quant Biol 1983; 48 : 453–63. 11. Ellis HM, Horvitz HR. Genetic control of programmed cell death in the nematode C. elegans. Cell 1986; 44 : 817–29. 12. Yuan J, Horvitz HR. The Caenorhabditis elegans genes ced-3 and ced-4 act cell autonomously to cause programmed cell death. Dev Biol 1990; 138 : 33–41. 13. Miura M, Zhu H, Rotello R, Hartwieg EA, Yuan J. Induction of apoptosis in fibroblasts by IL-1 β-converting enzyme, a mammalian homolog of the C.elegans cell death gene ced-3. Cell 1993; 75 : 653–60. 14. Alnemri ES, Livingston DJ, Nicholson DW, et al. Human ICE/CED-3 protease nomenclature. Cell 1996; 87 : 171. 15. Zou H, Henzel WJ, Liu X, Lutschg A, Wang X. Apaf-1, a human protein homologous to C. elegans CED-4, participates in cytochrome c-dependent activation of caspase-3. Cell 1997; 90 : 405–13. 16. Hengartner MO, Ellis RE, Horvitz HR. Caenorhabditis elegans gene ced-9 protects cells from programmed cell death. Nature 1992; 356 : 494–9. 17. Vaux DL, Cory S, Adams JM. Bcl-2 gene promotes haemopoietic cell survival and cooperates with c-myc to immortalize pre-B cells. Nature 1988; 335 : 440–2. 18. Metzstein MM, Stanfield GM, Horvitz HR. Genetics of programmed cell death in C.elegans: past, present and future. Trends Genet 1998; 14 : 410–6. 19. Gumienny TL, Hengartner MO. How the worm removes corpses: the nematode C. elegans as a model system to study engulfment. Cell Death Differ 2001; 8 : 564–8. 20. Aravind L, Dixit VM, Koonin EV. Apoptotic molecular machinery: vastly increased complexity in vertebrates revealed by genome comparisons. Science 2001; 291 : 1279–84. 21. Chung S, Gumienny TL, Hengartner MO, Driscoll M. A common set of engulfment genes mediates removal of both apoptotic and necrotic cell corpses in C. elegans. Nat Cell Biol 2000; 2 : 931–7. 22. Xu K, Tavernarakis N, Driscoll M. Necrotic cell death in C. elegans requires the function of nalreticulin and regulators of Ca2+ release from the endoplasmic reticulum. Neuron 2001; 31 : 957–71. 23. White K, Grether ME, Abrams JM, Young L, Farrell K, Steller H. Genetic control of programmed cell death in Drosophila. Science 1994; 264 : 677–83. 24. Abrams JM, White K, Fessler LI, Steller H. Programmed cell death during Drosophila embryogenesis. Development 1993; 117 : 29–43. 25. Grether ME, Abrams JM, Agapite J, White K, Steller H. The head involution defective gene of Drosophila melanogaster functions in programmed cell death. Genes Dev 1995; 9 : 1694–708. 26. Chen P, Nordstrom W, Gish B, Abrams JM. grim, a novel cell death gene in Drosophila. Genes Dev 1996; 10 : 1773–82. 27. Goyal L, McCall K, Agapite J, Hartwieg E, Steller H. Induction of apoptosis by Drosophila reaper, hid and grim through inhibition of IAP function. EMBO J 2000; 19 : 589–97. 28. Du C, Fang M, Li Y, Li L, Wang X. Smac, a mitochondrial protein that promotes cytochrome c-dependent caspase activation by eliminating IAP inhibition. Cell 2000; 102 : 33–42. 29. Verhagen AM, Ekert PG, Pakusch M, et al. Identification of DIABLO, a mammalian protein that promotes apoptosis by binding to and antagonizing IAP proteins. Cell 2000; 102 : 43–53. 30. Martins LM, Iaccarino I, Tenev T, et al. The serine protease Omi/HtrA2 regulates apoptosis by binding XIAP through a reaper-like motif. J Biol Chem 2001; 15 : 15. 31. Bangs P, Franc N, White K. Molecular mechanisms of cell death and phagocytosis in Drosophila. Cell Death Differ 2000; 7 : 1027–34. 32. Hu S, Yang X. dFADD, a novel death domain-containing adapter protein for the drosophila caspase DREDD. J Biol Chem 2000; 275 : 30761–4. 33. Inohara N, Koseki T, Hu Y, Chen S, Nunez G. CLARP, a death effector domain-containing protein interacts with caspase-8 and regulates apoptosis. Proc Natl Acad Sci USA 1997; 94 : 10717–22. 34. Deiss L P, Feinstein E, Berissi H, Cohen O, Kimchi A. Identification of a novel serine/threonine kinase and a novel 15-kDa protein as potential mediators of the gamma interferon-induced cell death. Genes Dev 1995; 9 : 15–30. 35. Cohen O, Feinstein E, Kimchi A. DAP-kinase is a Ca2+/calmodulin-dependent, cytoskeletal-associated protein kinase, with cell death-inducing functions that depend on its catalytic activity. EMBO J 1997; 16 : 998–1008. 36. Inbal B, Cohen O, Polak-Charcon S, et al. DAP kinase links the control of apoptosis to metastasis. Nature 1997; 390 : 180–4. 37. Raveh T, Droguett G, Horwitz MS, DePinho RA, Kimchi A. DAP kinase activates a p19ARF/p53-mediated apoptotic checkpoint to suppress oncogenic transformation. Nat Cell Biol 2001; 3 : 1–7. 38. Cohen O, Inbal B, Kissil JL, et al. DAP-kinase participates in TNF-alpha- and Fas-induced apoptosis and its function requires the death domain. J Cell Biol 1999; 146 : 141–8. 39. Jang CW, Chen CH, Chen CC, Chen JY, Su YH, Chen RH. TGF-beta induces apoptosis through Smad-mediated expression of DAP-kinase. Nat Cell Biol 2002; 4 : 51–8. 40. Thomas J, Leverrier Y, Marvel J. Bcl-X is the major pleiotropic anti-apoptotic gene activated by retroviral insertion mutagenesis in an IL-3 dependent bone marrow derived cell line. Oncogene 1998; 16 : 1399–408. 41. Watanabe-Fukunaga R, Brannan CI, Copeland NG, Jenkins NA, Nagata S. Lymphoproliferation disorder in mice explained by defects in Fas antigen that mediates apoptosis. Nature 1992; 356 : 314–7. 42. Murphy ED, Roths JB. A single gene model for massive lymphoproliferation with immune complex disease in new mouse strain MRL. Excerpta Medica International Congress. Amsterdam : Elsevier, 1977; 415 : 69–72. 43. Trauth BC, Klas C, Peters AMJ, et al. Monoclonal antibody-mediated tumor regression by induction of apoptosis. Science 1989; 245 : 301–5. 44. Yonehara S, Ishii A, Yonehara M. A cell-killing monoclonal antibody (Anti-Fas) to a cell surface antigen co-downregulated with the receptor of tumor necrosis factor. J Exp Med 1989; 169 : 1747–56. 45. Vercammen D, Beyaert R, Denecker G, et al. Inhibition of caspases increases the sensitivity of L929 cells to necrosis mediated by tumor necrosis factor. J Exp Med 1998; 187 : 1477–85. 46. Uren AG, O’Rourke K, Aravind L, et al. Identification of paracaspases and metacaspases. Two ancient families of caspase-like proteins, One of which plays a key role in MALT lymphoma. Mol Cell 2000; 6 : 961–7. 47. Hadfield KA, Bennett AB. Programmed senescence of plant organs. Cell Death Differ 1997; 4 : 662–70. 48. Fukuda H. Programmed cell death during vascular system formation. Cell Death Differ 1997; 4 : 684–8. 49. Beers EP. Programmed cell death during plant growth and development. Cell Death Differ 1997; 4 : 649–61. 50. Mittler R. Cell death in plants. In: Lockshin RA, Zakeri Z, Tilly JL, eds. When cells die. New York : Wiley-Liss Inc, 1998 : 147–73. 51. Lam E, Pontier D, Del Pozo O. Die and let live: programmed cell death in plants. Curr Opin Plant Biol 1999; 2 : 502–7. 52. Hammond-Kosack KE, Jones JD. Resistance gene-dependent plant defense responses. Plant Cell 1996; 8 : 1773–91. 53. Bonas U, Van den Ackerveken G. Gene-for-gene interactions: bacterial avirulence proteins specify plant disease resistance. Curr Opin Microbiol 1999; 2 : 94–8. 54. Holt BF, Mackey D, Dangl JL. Recognition of pathogens by plants. Curr Biol 2000; 10 : R5–7. 55. Tam LW, Kirk DL. The program for cellular differentiation in Volvox carteri as revealed by molecular analysis of development in a gonidialess / somatic regenerator mutant. Development 1991; 112 : 571–80. 56. Jazwinski SM. The genetics of aging in the yeast Saccharomyces cerevisiae. Genetica 1993; 91 : 35–51. 57. Fraser A, James C. Fermenting debate: do yeast undergo apoptosis? Trends Cell Biol 1998; 8 : 219–21. 58. Xu Q, Reed JC. Bax inhibitor-1, a mammalian apoptosis suppressor identified by functional screening in yeast. Mol Cell 1998; 1 : 337–46. 59. Hawkins CJ, Wang SL, Hay BA. A cloning method to identify caspases and their regulators in yeast: identification of Drosophila IAP1 as an inhibitor of the Drosophila caspase DCP-1. Proc Natl Acad Sci USA 1999; 96 : 2885–90. 60. Madeo F, Frohlich E, Frohlich KU. A yeast mutant showing diagnostic markers of early and late apoptosis. J Cell Biol 1997; 139 : 729–34. 61. Madeo F, Frohlich E, Ligr M, et al. Oxygen stress: a regulator of apoptosis in yeast. J Cell Biol 1999; 145 : 757–67. 62. Daugas E, Candé C, Kroemer G. Erythrocytes : death of a mummy. Cell Death Differ 2001; 8 : 1131–3. 63. Rizet G. Les phénomènes de barrage chez Podospora anserina. I. Analyse génétique des barrages entre les souches S et s. Rev Cytol Biol Veget 1952; 13 : 51–92. 64. Saupe SJ. Molecular genetics of heterokaryon incompatibility in filamentous ascomycetes. Microbiol Mol Biol Rev 2000; 64 : 489–502. 65. Coustou V, Deleu C, Saupe S, Begueret J. The protein product of the het-s heterokaryon incompatibility gene of the fungus Podospora anserina behaves as a prion analog. Proc Natl Acad Sci USA 1997; 94 : 9773–8. 66. Coustou V, Deleu C, Saupe SJ, Begueret J. Mutational analysis of the [Het-s] prion analog of Podospora anserina. A short N-terminal peptide allows prion propagation. Genetics 1999; 153 : 1629–40. 67. Loubradou G, Begueret J, Turcq B. A mutation in an HSP90 gene affects the sexual cycle and suppresses vegetative incompatibility in the fungus Podospora anserina. Genetics 1997; 147 : 581–8. 68. Bourges N, Groppi A, Barreau C, Clave C, Begueret J. Regulation of gene expression during the vegetative incompatibility reaction in Podospora anserina. Characterization of three induced genes. Genetics 1998; 150 : 633–41. 69. Jamet-Vierny C, Contamine V, Boulay J, Zickler D, Picard M. Mutations in genes encoding the mitochondrial outer membrane proteins Tom70 and Mdm10 of Podospora anserina modify the spectrum of mitochondrial DNA rearrangements associated with cellular death. Mol Cell Biol 1997; 17 : 6359–66. 70. Begel O, Boulay J, Albert B, Dufour E, Sainsard-Chanet A. Mitochondrial group II introns, cytochrome c oxidase, and senescence in Podospora anserina. Mol Cell Biol 1999; 19 : 4093–100. 71. Dufour E, Boulay J, Rincheval V, Sainsard-Chanet A. A causal link between respiration and senescence in Podospora anserina. Proc Natl Acad Sci USA 2000; 97 : 4138–43. 72. Loomis WF. Genetic tools for Dictyostelium discoideum. Meth Cell Biol 1987; 28 : 31–65. 73. Kuspa A, Loomis WF. Tagging developmental genes in Dictyostelium by restriction enzyme-mediated integration of plasmid DNA. Proc Natl Acad Sci USA 1992; 89 : 8803–7. 74. Kay RR. Cell differentiation in monolayers and the investigation of slime mold morphogens. Meth Cell Biol 1987; 28 : 433–48. 75. Cornillon S, Foa C, Davoust J, Buonavista N, Gross JD, Golstein P. Programmed cell death in Dictyostelium. J Cell Sci 1994; 107 : 2691–704. 76. Arnoult D, Tatischeff I, Estaquier J, et al. On the evolutionary conservation of the cell death pathway: mitochondrial release of an apoptosis-inducing Factor during Dictyostelium discoideum cell death. Mol Biol Cell 2001; 12 : 3016–30. 77. Tatischeff I, Petit PX, Grodet A, Tissier JP, Duband-Goulet I, Ameisen JC. Inhibition of multicellular development switches cell death of Dictyostelium discoideum towards mammalian-like unicellular apoptosis. Eur J Cell Biol 2001; 80 : 428–41. 78. Olie RA, Durrieu F, Cornillon S, et al. Apparent caspase independence of programmed cell death in Dictyostelium. Curr Biol 1998; 8 : 955–8. 79. Cornillon S, Olie RA, Golstein P. An insertional mutagenesis approach to Dictyostelium cell death. Cell Death Differ 1998; 5 : 416–25. 80. Levraud JP, Adam M, Cornillon S, Golstein P. Methods to study cell death in Dictyostelium discoideum. In : Schwartz LM, Ashwell J, eds. Cell death. Methods in cell biology, 2e ed. San Diego: Academic Press, 2001 : 469–97. 81. Aballay A, Ausubel FM. Programmed cell death mediated by ced-3 and ced-4 protects Caenorhabditis elegans from Salmonella typhimurium-mediated killing. Proc Natl Acad Sci USA 2001; 98 : 2735–9. 82. Freeman RS, Estus S, Johnson EM Jr. Analysis of cell cycle-related gene expression in postmitotic neurons: selective induction of cyclin D1 during programmed cell death. Neuron 1994; 12 : 343–55. 83. Chen G, Shi L, Litchfield DW, Greenberg AH. Rescue from granzyme B-induced apoptosis by Wee1 kinase. J Exp Med 1995; 181 : 2295–300. 84. Kranenburg O, Van der Eb A, Zantema A. Cyclin D1 is an essential mediator of apoptotic neuronal cell death. EMBO J 1996; 15 : 46–54. 85. Wang J, Walsh K. Resistance to apoptosis conferred by Cdk inhibitors during myocyte differentiation. Science 1996; 273 : 359–61. 86. Harvey KJ, Lukovic D, Ucker DS. Caspase-dependent Cdk activity is a requisite effector of apoptotic death events. J Cell Biol 2000; 148 : 59–72. 87. Kitanaka C, Kuchino Y. Caspase-independent programmed cell death with necrotic morphology. Cell Death Differ 1999; 6 : 508–15. 88. Leist M, Jaattela M. Four deaths and a funeral: from caspases to alternative mechanisms. Nat Rev Mol Cell Biol 2001; 2 : 589–98. 89. Wyllie AH, Golstein P. More than one way to go. Proc Natl Acad Sci USA 2001; 98 : 11-3. |