Le dosage de l’expression des gènes est essentiel au bon fonctionnement cellulaire ainsi qu’au développement et au maintien des organismes pluricellulaires. De fait, des mécanismes biologiques permettent d’activer ou de réprimer l’expression des gènes sans affecter l’intégrité du support de l’information génétique. Au cours des dernières années, les biologistes se sont notamment intéressés aux phénomènes épigénétiques aboutissant à la répression de l’expression d’un gène. Ces régulations négatives, qui affectent aussi bien la transcription (TGS : inhibition épigénétique de la transcription) que les processus subséquents de l’expression des gènes (stabilité des ARN messagers, PTGS : inhibition épigénétique post-transcriptionelle), sont communes à tous-les eucaryotes [1]. Parmi les approches qui permettent d’étudier la fonction proprement dite d’un gène, l’une d’entre elles consiste à supprimer l’expression de ce dernier, dans des cellules ou dans un organisme. De nouvelles techniques, comme l’interférence par ARN (ARNi [2]), fondées sur l’utilisation d’ARN double brin (ARNdb), sont en voie de développement.
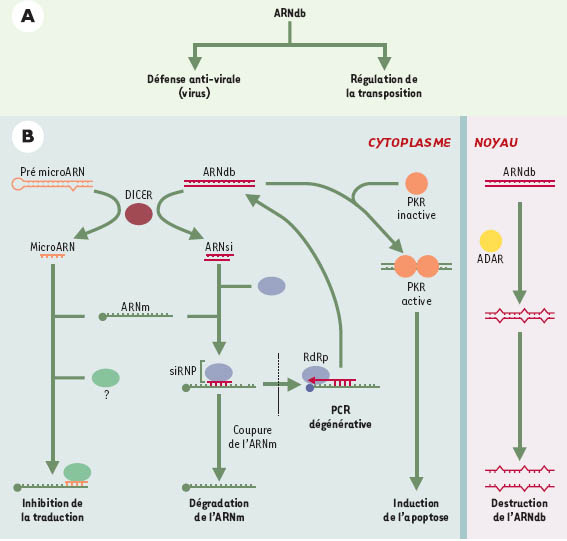
La réaction ARNi détourne des enzymes impliquées dans le métabolisme d’ARNdb produits par les virus et les transposons. En effet, ces ARNdb de grande taille induisent des mécanismes cellulaires antiviraux ou des phénomènes de régulation de la transposition (Figure 1A). La réaction cellulaire met en jeu au niveau nucléaire des enzymes (ADAR, adénosine désaminase) capables de désaminer en inosine les adénines de l’ARNdb [3]. Ces modifications induisent la déstructuration des ARNdb et la séparation des deux brins d’ARN, conduisant à leur dégradation par des ribonucléases (Figure 1B). D’autre part, l’introduction d’un long ARNdb dans le cytoplasme active la production d’interféron, ainsi qu’une protéine kinase ARN dépendante (PKR). PKR est activée par dimérisation en présence de longs ARNdb, et autophosphorylation. PKR activée réprime la traduction du facteur d’initiation de la traduction eIF2α. De plus, l’ARNdb induit la dégradation des ARN cellulaires en stimulant la ribonucléase L (RNaseL). Finalement, l’activation de la PKR, en conjonction avec la voie des interférons, induit l’apoptose cellulaire (Figure 1B) [3]. Ce mécanisme est déficient dans le cas des cellules embryonnaires telles que les cellules souches et les embryotératocarcinomes. Dans ces dernières, c’est une autre protéine, appelée Dicer, qui est fortement exprimée [4]. Dicer est une enzyme comportant plusieurs domaines dont deux motifs ribonucléase III catalysant la coupure des longs ARNdb en petits fragments de 21 à 23 nucléotides appelés petits ARN interférents (ARNsi) [5]. Cette enzyme est importante au cours du développement embryonnaire puisqu’elle est impliquée dans la maturation des microARN nécessaires à la transition entre les stades larvaires chez C. elegans [6]. Ces deux formes d’ARN, ARNsi et microARN, représentent des signaux moléculaires essentiels lors des phénomènes de régulation post-transcriptionnelle de l’expression des gènes.
| Figure 1.
A. ARN double brin (ARNdb) cellulaires dans les cellules mammifères. B. Métabolisme des ARNdb et microARN : modèles de la réaction d’ARN interférence (ARNi). ADAR : adénosine désaminase ; PKR : protéine kinase ARN dépendante ; RdRp : ARN polymérase ARN dépendante ; ARNsi : petit ARN interférent ; siRNP : complexe ribonucléoprotéique impliquant les ARNsi ; ARNm : ARN messager. |
Deux processus biologiques distincts découlent de l’activité de ces deux formes de petits ARN (Figure 1B). Le mécanisme biologique d’action des microARN, actuellement très mal caractérisé, impliquerait d’autres facteurs protéiques dont l’association permettrait spécifiquement l’inhibition de la traduction de gènes cibles [7]. L’ARNi, processus beaucoup mieux décrypté, présente la particularité d’agir par l’intermédiaire de voies de signalisation différentes en fonction des organismes. Chez la drosophile, l’ARNi nécessite la formation d’un complexe (siRNP) entre l’ARNsi et des protéines. Le complexe siRNP catalyse la réaction de clivage de l’ARN cible correspondant à la séquence complémentaire à l’ARNsi. La conséquence de cette coupure est la déstabilisation et la dégradation de l’ARN cible. Dans le cas d’un ARN messager (ARNm), le gène correspondant est réduit au silence. Dans d’autres organismes, tel C. elegans, une enzyme est capable de catalyser la synthèse d’un brin ARN à partir d’une matrice ARN. Cette ARN polymérase ARN dépendante (RdRp) [8] utilise comme amorce l’un des brins de l’ARNsi. Le résultat de cette réaction est la formation d’un ARNdb. Ce dernier est alors utilisé comme substrat par Dicer. Ce modèle dit de « PCR dégénérative » aboutit à une amplification du signal ARNsi.
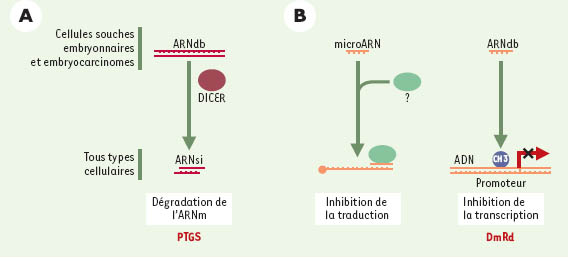
L’étude de ces phénomènes biologiques et de ces mécanismes enzymatiques a permis de développer différentes stratégies pour inhiber spécifiquement l’expression d’un gène par ARNi dans des cellules mammifères en culture (Figure 2A). D’une part, dans les cellules différenciées, il existe un système permettant de passer outre les mécanismes d’induction de l’apoptose cellulaire décrits précédemment en apportant un ARNdb de petite taille. Ainsi, l’introduction d’ARNsi est suffisante pour que la réaction de clivage de l’ARN cible se produise de façon efficace [9]. D’autre part, une interférence ARNi à partir d’un long ARNdb peut être réalisée in vitro dans des cellules souches embryonnaires ou d’embryotératocarcinomes (exemple de gènes inactivés, eGFP, intégrines α3 et β1) [4].
| Figure 2.
A. Stratégies d’introduction de la réaction ARNi dans un système utilisant des cellules de mammifères. B. Voie potentielle d’inhibition artificielle de l’expression de gènes dans les cellules de mammifères (méthylation ARN dépendante). ARNdb : ARN double brin ; ARNsi : petit ARN interférent ; DmRd : réaction ARNdb dépendante de la méthylation ; PTGS : post-transcriptional genetic silencing. |
Les récentes études décrivant les effets des microARN, et une connaissance plus approfondie des mécanismes d’action de ces derniers, permettront peutêtre de développer d’autres stratégies. L’utilisation d’ARN artificiel pourrait permettre de cibler l’expression d’un gène par inhibition de la traduction (Figure 2B). Par ailleurs, les plantes disposent d’un mécanisme qui permettrait de catalyser une réaction ARNdb dépendante de la méthylation de l’ADN (DmRd) [10]. La méthylation d’une séquence promotrice implique généralement une inhibition de la transcription d’un gène. On pourrait donc envisager de réprimer l’expression d’un gène, par méthylation de ces séquences régulatrices, en utilisant comme support signalétique un ARNdb spécifique d’une séquence donnée.