| |
| Med Sci (Paris). 2002 March; 18(3): 366–374. Published online 2002 March 15. doi: 10.1051/medsci/2002183366.Modélisation des données biologiques Bio-informatique (2) Anne Morgat and François Rechenmann Inria Rhône-Alpes, 655, avenue de l’Europe, Montbonnot, 38334 Saint Ismier, France |
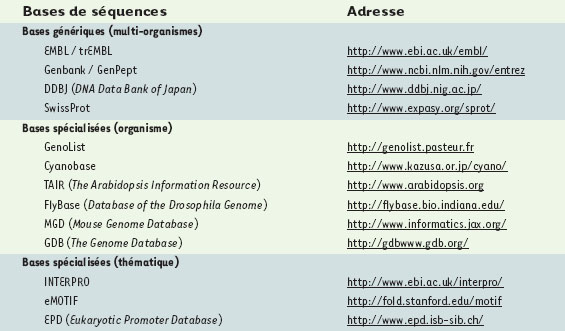
Les bases de données dédiées à la biologie moléculaire sont un complément essentiel aux données de la littérature. Elles constituent un des tout premiers points de contact des chercheurs biologistes avec l’informatique. Dès le milieu des années 1970, les banques de séquences se sont ainsi organisées pour accueillir l’ensemble des séquences publiques, ainsi que les annotations qui leur sont associées. Le terme « banque» » rappelle ici que les séquences y sont déposées directement par les chercheurs qui les ont obtenues, sous leur seule responsabilité. Il existe actuellement trois banques principales, EMBL, Genbank et DDBJ, accessibles via Internet, qui partagent sensiblement les mêmes données et constituent de ce fait trois points d’entrée d’une seule et même banque mondiale. Pour donner un ordre d’idée, en février 2002 la base EMBL contenait environ 17 milliards de nucléotides. À côté de ces bases généralistes, multi-espèces, redondantes et de qualité variable, de très nombreuses bases de données spécialisées se sont développées (Tableau I). La plus célèbre est sans aucun doute SwissProt qui contient plus de 100 000 protéines traduites d’EMBL, expertisées et réannotées manuellement. Beaucoup de banques spécialisées sont dédiées à une espèce ou un sous-ensemble d’espèces, telles que GenoList à l’Institut Pasteur pour les bactéries E. coli, B. subtilis, M. tuberculosis, M. leprae et H. pylori, Cyanobase pour la cyanobactérie Synechosystis, ou TAIR pour la plante A. thaliana. Outre un ensemble vérifié et non redondant de séquences, elles recueillent des annotations remises à jour en cas d’erreurs détectées ultérieurement. D’autres rassemblent des annotations complémentaires qu’elles relient aux séquences contenues dans les banques. C’est par exemple le cas de FlyBase pour la drosophile D. melanogaster, MGD pour la souris ou encore GDB pour l’homme. Enfin, d’autres bases sont thématiquement spécialisées. Par exemple, la base EPD (eukaryotic promoter database) rassemble les séquences de promoteurs eucaryotes, et les bases INTERPRO et eMOTIF décrivent des motifs et des profils de familles de protéines.  | Tableau I. Quelques adresses de bases de données de séquences, génériques ou dédiées à l’étude d’un organisme ou d’une thématique particulière. |
|
Une multiplicité de bases de données, hétérogènes et distribuées Il est difficile d’estimer le nombre de bases de données actuellement dédiées à la biologie moléculaire, mais il est probablement de l’ordre du millier. Chaque année, le numéro de janvier de la revue Nucleic Acids Research [http://nar.oupjournals.org] est composé de près d’une centaine d’articles décrivant les bases les plus importantes et introduisant les nouvelles. La version électronique de la revue permet d’accéder à une liste de presque 300 d’entre elles [http://www3.oup.co.uk/nar/database/c/]. Par ailleurs, plusieurs sites Web ont pour vocation de répertorier l’ensemble des bases disponibles comme par exemple le site DBcat (The Public Catalog of Database) géré par Infobiogen [http://www.info biogen.fr/services/dbcat] ou le serveur ExPASy [http://www.expasy.ch] géré par l’Institut Suisse de Bioinformatique (SIB). Les données biologiques ne se limitent toutefois pas aux séquences. Le Tableau II présente un aperçu des différents types de bases spécialisées dédiées à l’étude du métabolisme, de la régulation de la transcription, des interactions protéine-protéine, de la structure tridimensionnelle de macromolécules ou encore d’une famille particulière de gènes ou de protéines.  | Tableau II. Quelques exemples de bases de données spécialisées. |
S’il existe autant de bases de données biologiques, c’est parce qu’elles sont conçues pour répondre à des objectifs différents. De ce fait, même quand leurs contenus se recouvrent, leurs schémas conceptuels (voir glossaire) peuvent différer. Rappelons qu’un schéma conceptuel constitue un modèle dont la conception est pilotée par les questions qu’il doit permettre d’aborder. Il est donc tout à fait illusoire de penser construire un jour « le » système d’informations biologiques universel : il faut accepter la pluralité des problématiques, qui induit une pluralité des bases de données. Malheureusement, cette diversité, nécessaire, a des conséquences pratiques assez fâcheuses. En effet, lors d’une recherche d’informations un peu complexe, le chercheur est très vite conduit à interroger plusieurs bases et à tenter de relier entre elles les données qu’il en extrait. Avec le développement d’Internet, l’accès proprement dit ne constitue pas un obstacle : la quasi-totalité des bases de données biologiques sont accessibles sur le Web, les modalités variant éventuellement suivant la nature des données et le statut du demandeur. Lors d’une analyse classique, qui consiste à rassembler l’ensemble des informations disponibles sur une protéine ou un gène particulier, le biologiste devra intégrer les informations de différentes bases de données. Cette recherche, bien que fastidieuse, peut encore être effectuée « à la main ». En revanche, cette démarche devient inenvisageable lorsqu’il s’agit d’analyser des résultats à grande échelle (génome complet, transcriptome, protéome…), tels que ceux fournis par les nouvelles technologies (séquençage exhaustif, puce à ADN, électrophorèse 2D…). La diversité des modèles et des formats des bases concernées constitue ainsi un véritable problème. Dans le meilleur des cas, il faut s’adapter au modèle de chacune d’entre elles. Le plus souvent cependant, les dites « bases de données » sont constituées de simples fichiers munis d’un langage d’interrogation et de manipulation spécifique. De ce fait, l’écriture de petits programmes de lecture et de reformatage de données est une des activités majeures des bio-informaticiens. De plus, afin de formuler les bonnes requêtes pour chacune de ces bases, il est à l’évidence nécessaire de connaître leur structure, leur schéma conceptuel. Or, ce schéma n’est pas toujours disponible ; dans le cas des fichiers, il est même inexistant. Et il est malheureusement illusoire de penser qu’il suffit de connaître le nom d’un champ d’une relation ou d’un enregistrement de fichier pour savoir ce qu’il désigne et ce que ce champ contient réellement. En effet, un même nom est souvent utilisé avec des acceptions différentes. L’exemple le plus frappant est sans conteste le terme « gène ». On s’attend à ce qu’au sein d’un même domaine de connaissances, un terme aussi fondamental désigne de façon non ambiguë un seul et même concept. Il n’en est rien et, là encore, c’est la diversité des problématiques qui explique la diversité des interprétations. Au-delà de l’évidente différence conceptuelle entre le gène mendélien et le gène moléculaire, le terme « gène » est susceptible de recouvrir des réalités fort différentes d’une base à une autre. Ainsi, le problème majeur de la gestion des données biologiques ne résulte pas tant de leur volume, encore bien inférieur à ce que les techniques développées dans le domaine des bases de données permettent d’assumer, mais bien de leur hétérogénéité, tant en termes de nature que de format. Le mot-clé est donc ici « intégration » et la question fondamentale est : comment intégrer ces données biologiques, hétérogènes et distribuées, afin qu’elles soient accessibles et exploitables aussi facilement que si elles figuraient dans une seule et même base ? Deux grandes catégories de solution sont envisageables. La première consiste à ajouter, au-dessus des bases existantes, une couche logicielle qui offre les interfaces nécessaires entre les bases et fasse apparaître l’ensemble comme une seule base virtuelle. Cette approche, dite « fédérative », assure d’accéder à tout instant à des données qui sont à jour. La seconde approche est celle des « entrepôts de données » (data wharehousing) : les données des différentes bases concernées sont copiées de leurs bases d’origine et restructurées au sein d’un schéma unique. Les performances en temps de traitement des requêtes sont évidemment meilleures qu’avec la première démarche, mais il faut assurer la mise à jour de l’entrepôt afin de suivre les mises à jour des bases d’origine. Ainsi, les deux approches requièrent que soient résolus les problèmes d’incompatibilité syntaxique et sémantique éventuels : la première « à la volée », c’est-à-dire à chaque requête ; la seconde à chaque fois que l’entrepôt est mis à jour. La compatibilité syntaxique, c’est-à-dire des formats de données, nécessite que soient disponibles des programmes d’interface pour chaque couple de bases qui sont appelées à communiquer. Afin de préserver la communication entre les bases de données, il est indispensable de ne plus modifier leurs interfaces, même lorsque leurs schémas évoluent. C’est sur ce principe dit « d’encapsulation » que repose la motivation pour le développement de « standards » tels que CORBA (common object request broker architecture) [1]. Ces standards ne résolvent pas, en tant que tels, le problème de la compatibilité syntaxique, mais lui offrent un contexte logiciel favorable. Au-delà de ces problèmes, certes fondamentaux, mais essentiellement techniques, il reste à résoudre le problème de la compatibilité sémantique : comment les entités modélisées dans un certain schéma conceptuel peuvent-elles être mises en correspondance avec leurs homologues supposées dans le schéma d’une autre base ? Comme cela est expliqué précédemment, la seule connaissance des noms ne saurait fournir cette correspondance. Même dans le cas le plus favorable, où il existe un schéma conceptuel accessible et correctement documenté, cette mise en correspondance reste une opération délicate qui repose sur une expertise biologique. La difficulté provient essentiellement du fait qu’un schéma conceptuel décrit les entités de la base sous une forme nécessaire au traitement des requêtes et à l’administration de la base. Il n’explicite donc pas nécessairement les définitions de ces entités, autrement que sous la forme de textes dans la documentation associée, quand elle existe. Expliciter et formaliser ces définitions constituent donc un objectif à atteindre si le problème de l’inter-opérabilité sémantique des bases de données biologiques doit trouver une solution générique. |
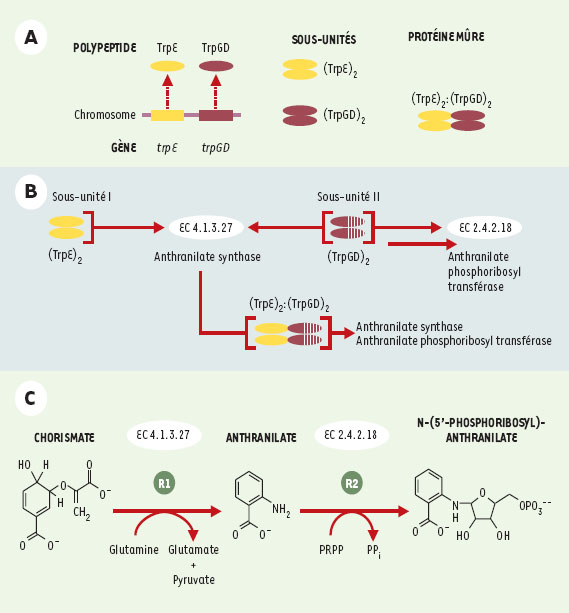
Des bases de données aux bases de connaissances Ainsi, modéliser plus finement les classes d’entités, ainsi que les relations qu’elles entretiennent, non plus seulement à des fins de requêtes et de gestion, mais pour expliciter formellement leurs définitions, fait passer de la notion de base de données à celle de base de connaissances. Là encore, il faut écarter toute illusion sur l’unicité d’une base de connaissances en biologie moléculaire, d’une « ontologie » à vocation universelle. Rappelons qu’une ontologie correspond à la formalisation des concepts d’un domaine et des relations qu’ils entretiennent [2]. En pratique, le schéma d’une base de connaissances correspond à la mise en œuvre de l’ontologie retenue. La recherche en informatique s’est attaquée très tôt au problème de la modélisation des connaissances et a développé plusieurs formalismes dans lesquels les connaissances d’un domaine peuvent être décrites afin d’être simultanément lisibles par une personne et exploitables par la machine. Parmi ces formalismes, ceux dits « à objets », qui reposent sur la notion de classes d’objets, structurées en hiérarchies de classes et de sous-classes de plus en plus spécifiques, ellesmêmes décrites en termes d’attributs typés, se sont progressivement imposés, sans cependant qu’un véritable standard n’ait émergé [3]. En pratique, il est judicieux de recourir à un formalisme de plus haut niveau, tel que UML (unified modeling language), pour écrire le schéma conceptuel d’une base, puis de le « traduire » dans un modèle relationnel ou à objets particulier, propre à un certain SGBD [4]. Une première catégorie de motivations au développement d’une base de connaissances est d’assurer l’interopérabilité sémantique de plusieurs bases de données liées à des problématiques portant sur des domaines qui se recouvrent, en explicitant la définition des concepts qu’elles mettent en œuvre. Il est alors possible de réexprimer automatiquement une requête complexe portant sur des données contenues dans des bases distinctes en une série de sous-requêtes qui leur sont adressées, puis de reconstituer, à partir des réponses reçues, une réponse à la requête complexe initiale. C’est par exemple l’approche suivie par le système TAMBIS développé à l’université de Manchester (Royaume-Uni) [5]. Les modèles de connaissances offrent une capacité d’expression qui permet d’aborder la représentation de données plus complexes que celles qui apparaissent traditionnellement dans les bases. Il est ainsi fréquent que des champs, potentiellement très informatifs, ne soient remplis dans des bases de données qu’avec du texte en langage naturel du fait de l’incapacité d’en exprimer le contenu formellement. Si ces textes peuvent évidemment être lus et interprétés par les personnes qui consultent ces bases, leur exploitation automatique est très délicate. Ces champs sont, de ce fait, exclus de toute requête. C’est très fréquemment le cas du champ « fonction » associé à un gène ou à une protéine. Cependant, les travaux fondateurs de P. Karp et M. Riley montrent qu’il est possible d’en faire une description explicite et formelle et donc facilement « requêtable » [6, 7]. Ces mêmes modèles de connaissances facilitent par ailleurs la description des relations entre les classes d’entités et entre les entités elles-mêmes. Il est ainsi possible de représenter explicitement des réseaux, tels que les réseaux de régulation de l’expression des gènes ou les réseaux métaboliques. Il est par exemple intéressant de comparer la base EcoCyc [8], dans laquelle les voies métaboliques de la bactérie E. coli sont explicitement décrites, et la base KEGG [9], où ces voies sont essentiellement stockées sous la forme d’images et de textes. Il est clair que la première forme facilite grandement le traitement de requêtes évoluées et l’application de mécanismes d’inférence. Un exemple simple mais complet de modélisation est détaillé dans les Figures 1 à3 . La problématique consiste à faire une description fonctionnelle de la protéine anthranilate synthase de la bactérie Escherichia coli. L’anthranilate synthase est la première enzyme de la phase terminale de la voie de biosynthèse de l’acide aminé tryptophane (voie métabolique permettant la transformation du chorismate en tryptophane). Cette voie métabolique est bien caractérisée, elle a fait l’objet de nombreuses études dans divers organismes depuis de nombreuses années et peut être considérée comme un « cas d’école » [10]. 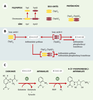 | Figure 1. Description fonctionnelle de l’enzyme anthranilate synthase (E. coli). A. Constituants moléculaires. L’anthranilate synthase de la bactérie E. coli est constituée de deux sous-unités (I et II). La sous-unité I correspond à la dimérisation du polypeptide codé par le gène trpE, alors que la sous-unité II correspond à la dimérisation du polypeptide codé par le gène trpGD. L’anthranilate synthase d’E. coli est donc un tétramère. B. Activités enzymatiques. La sous-unité I est impliquée dans la catalyse de la réaction qui transforme une molécule de chorismate en anthranilate. Cette activité de catalyse est associée à l’identifiant EC 4.1.3.27 (classification enzymatique internationale). La sous-unité II est dite bifonctionnelle, car elle est responsable de la catalyse de deux réactions biochimiques. D’une part, elle participe, avec la sous-unité I, à la production d’anthranilate à partir de chrorismate (EC 4.1.3.27) ; d’autre part, elle catalyse la transformation du produit de la réaction précédente - l’anthranilate - en dérivé phosphoribosyl-anthranilate (EC 2.4.2.18). Cette seconde activité enzymatique est indépendant de la première, elle peut être effectuée en l’absence d’interaction avec la sous-unité I. En résumé, le complexe moléculaire (TrpE)2 : (TrpGD)2 possède à la fois une activité anthranilate synthase et une activité anthranilate phosphoribosyl transférase. C. Réactions biochimiques catalysées par les enzymes EC 4.1.3.27 et EC 2.4.2.18. Afin de rendre plus explicites les composés intervenant comme subtrat(s) ou produit(s) dans une réaction biochimique, il est souvent nécessaire de les représenter par leur formule plane (2D). |
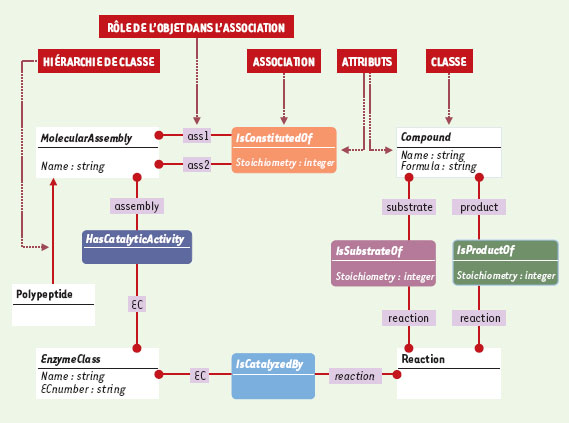
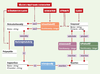 | Figure 2.Formalisation du problème : le diagramme de classes et associations. Le processus de modélisation consiste à identifier les concepts mis en jeu et à les traduire à l’aide d’un système de représentation de connaissances. Le système AROM est fondé sur un formalisme dérivé d’UML. Les classes sont représentées par des rectangles à angles droits, les associations sont représentés par des rectangles à bords arrondis. Un code couleur permet de distinguer les différentes associations (ce même code couleur est repris dans le diagramme d’instances, Figure 3). Dans cet exemple, il s ’agit de décrire :
- des polypeptides : un polypeptide représente le produit d’un gène codant pour une protéine. la classe Polypeptide représente l’ensemble des polypeptides.
- des assemblages moléculaires : un assemblage moléculaire est constitué de polypeptides ou d’assemblages moléculaires. Un assemblage moléculaire est caractérisé par le nombre d’entités qui le constitue. la classe MolecularAssembly est une généralisation de la classe Polypeptide. Autrement dit, la classe Polypeptide représente une spécialisation de la classe MolecularAssembly. Les classes Polypeptide et Molecular Assembly sont décrites par l’attribut Name (chaîne de caractères). L’association IsConstitutedOf permet de décrire de façon récursive la composition d’un assemblage moléculaire. Le nombre de constituants d’un assemblage moléculaire est décrit par l’attribut Stoichiometry (nombre entier).
- des enzymes : la dénomination des enzymes est définie par les règles de la nomenclature internationale (IUBMB : International Union of Biochemistry and Molecular Biology). Un identifiant (EC number) est associé à chaque enzyme répertoriée. La classification enzymatique internationale regroupe six grandes classes d’enzymes. la classe EnzymeClass représente les éléments de la classification enzymatique internationale. L’association HasCatalyticActivity permet de connecter les classes MolecularAssembly et EnzymeClass.
- des réactions biochimiques : une réaction biochimique décrit la transformation de substrat(s) en produit(s). Une réaction biochimique est généralement catalysée par une enzyme.
la classe Reaction représente les réactions biochimiques. L’association IsCatalyzedBy connecte les classes Reaction et EnzymeClass. Cette association permet d ’établir le lien entre une réaction biochimique et son catalyseur enzymatique. La classe Compound regroupe l’ensemble des composés intervenant dans une réaction biochimique. Les associations IsSubstrateOf et IsproductOf permettent de spécifier le rôle du composé dans la réaction (composés substrats de la réaction ou composés produits de la réaction). |
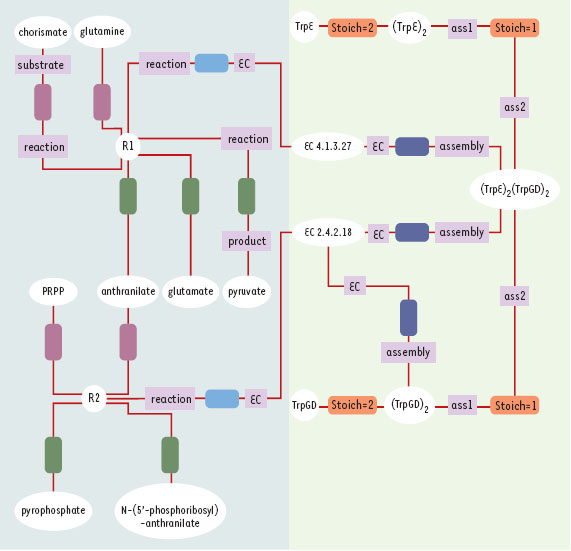
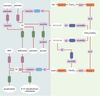 | Figure 3. Diagramme d’instances. Les objets (instances de classes) sont représentés par leur nom. Les tuples (instances d’associations) sont représentées par des rectangles à bords arrondis. Dans AROM, les tuples n’ont pas d’identifiant, ils sont uniquement définis par les rôles. Afin de les discerner, les tuples sont représentés avec le code couleur correspondant dans le diagramme de classes (Figure 2). Comment lire un diagramme d’instances : La partie gauche du diagramme décrit les réactions chimiques. Les composés chorismate et glutamine sont les substrats de la réaction R1. Les instances chorismate et glutamine (représentées par des ovales) sont des éléments de la classe Compound. Chacun de ces éléments est relié à l’instance R1 de la classe Reaction par l’association IsSubstrateOf matérialisée par des rectangles violets. D’une façon similaire, les composés - produits de la réaction R1- sont représentés par les instances anthranilate, glutamate et pyruvate. Chacun de ces éléments de la classe Compound est relié à l’instance R1 par l’association IsProductOf matérialisée par un rectangle vert. Le composé anthranilate peut être considéré comme un intermédiaire réactionnel puisqu’il est à la fois produit de la première réaction et substrat de la seconde réaction. Dans la base, une seule instance anthranilate est reliée à la fois à l’instance R1, par l’association IsProductOf, et à l’instance R2 par l’association IsSubstrateOf. La partie droite du diagramme décrit la composition des assemblages moléculaires et les activités enzymatiques associées. Par exemple, l’instance (TrpGD)2 de la classe MolecularAssembly est reliée à l’instance TrpGD de la classe Polypeptide par l’association IsConstitutedOf dont la valeur de l’attribut Stoichiometry est égale à 2. Cet assemblage moléculaire possède une activité enzymatique propre. L’instance (TrpGD)2 est donc reliée à l’instance EC2.4.2.18 de la classe EnzymeClass par l’association HasCatalyticActivity. Par ailleurs, (TrpGD)2 est impliqué dans la constitution de la protéine mûre (instance [TrpE]2:[TrpGD]2 qui possède deux activités enzymatiques). |
La première étape d’un processus de modélisation consiste à formaliser le domaine biologique d’intérêt et à identifier les concepts que l’on veut représenter. Dans cet exemple, la description fonctionnelle de la protéine est restreinte à la description de sa composition moléculaire et de son activité enzymatique (réaction biochimique catalysée par cette enzyme). Cette étape est du ressort du biologiste, expert du domaine. Ainsi la Figure 1 donne un résumé, présenté sous forme d’une description textuelle et graphique, des informations concernant cette protéine. La seconde étape consiste à traduire les concepts manipulés dans un formalisme UML. Pour cet exemple, le système de représentation de connaissances AROM (Allier Relations et Objets pour Modéliser) [11] a été utilisé. Succinctement, ce système de modélisation dispose de deux entités de représentations complémentaires appelées « classe » et « association ». Une classe représente un concept ; il lui est associé un ensemble d’éléments ayant la même structure et la même sémantique. Un élément d’une classe est appelé « objet » ou « instance » de la classe. Une association permet de relier deux classes ou plus, distinctes ou non. Un élément d’une association est appelé tuple ; il est défini par les rôles des objets de chacune des classes impliquées dans l’association. Les classes et associations peuvent être caractérisées par des attributs typés et peuvent être organisées de façon hiérarchique, des plus générales au plus spécifiques. La conception et le développement de tels systèmes relèvent du domaine de l’informatique. En revanche, la « traduction » des concepts manipulés sous forme d’un schéma de classes et d’associations tel qu’il est présenté sur la Figure 2 relève de la bio-informatique. Enfin, la troisième étape consiste à instancier le modèle, c’est-à-dire à alimenter la base avec les données disponibles. Ce processus est illustré par le diagramme d’instances (Figure 3). Une fois cette dernière étape réalisée, il est alors possible de poser, via un langage de requête, des questions précises de complexité variable sur l’ensemble des instances de la base. Ce travail de modélisation peut sembler complexe et fastidieux dans le cas de la description d’une seule protéine. Cependant, il est clair que le modèle obtenu est en fait générique et pourra ainsi être utilisé pour décrire la grande majorité des enzymes, quel que soit l’organisme et quelle que soit la classe enzymatique. |
Face à la production systématique de données, le recours indispensable à l’informatique est désormais reconnu par la très grande majorité des biologistes. Mais le plus souvent, l’informatique est vue, à travers sa matérialisation qu’est l’ordinateur, comme un « simple outil » destiné à stocker ces données et à calculer des résultats. Cette perception est extrêmement réductrice ; elle écarte de fait les apports de l’informatique en matière de modélisation. Identifier et définir formellement tous les objets et leurs relations pertinents pour la résolution d’une classe de problèmes est en effet une des premières étapes de tout projet informatique. Que ce soit dans le domaine des bases de données, de la représentation des connaissances et du génie logiciel, les avancées de l’informatique se sont traduites par des propositions de modèles et de démarches de conception. Les modèles actuellement disponibles, qu’ils soient conceptuels (UML), opérationnels (SGBD) ou bien destinés à la construction de bases de connaissances (AROM), permettent d’expliciter et de décrire formellement les objets particulièrement complexes de la biologie. Cette modélisation préalable à toute manipulation in silico est nécessaire et bénéfique. Elle doit permettre, on l’a vu, de résoudre le problème posé par la diversité naturelle des bases de données et par la nécessité de recouper à la demande les informations qu’elles contiennent. Elle doit également précéder la conception et le développement de tout logiciel destiné à manipuler ou à construire ces objets. Mais, comme dans toute démarche de modélisation, le bénéfice réside tout autant dans le produit que dans le processus. Le fait même d’expliciter les objets d’intérêt et les relations qu’ils entretiennent fait progresser dans la connaissance et dans la compréhension de la problématique qui est à l’origine de cette modélisation, et par là du domaine ui-même. |
| Système de gestion de bases de données (SGBD) |
Une base de données est constituée d’un contenant et d’un contenu. Le contenant est la description de la nature et de la structure du contenu, c’est-à-dire des données elles-mêmes. Concevoir une base de données, c’est tout d’abord spécifier le contenant : le schéma conceptuel. Une fois ce schéma établi et implémenté à l’aide d’un système de gestion de bases de données, un SGBD, la base est opérationnelle : elle peut être alimentée en données et des requêtes peuvent être soumises par les utilisateurs. Une requête est en fait une question particulière posée à la base ; la réponse est un ensemble de données qui satisfont les critères énoncés dans la requête. Bien entendu, à tout moment, les données de la base peuvent être modifiées ou supprimées et de nouvelles données peuvent être ajoutées : ce sont les tâches d’édition de la base.L’intérêt d’utiliser un SGBD est qu’une fois le schéma conceptuel décrit dans le modèle de données associé, toutes les opérations d’édition, de vérification, de sécurisation et de traitement des requêtes sont « offertes ». À l’inverse, gérer un ensemble de données directement sur des fichiers, aussi sophistiqués soient-ils, oblige à programmer spécifiquement toutes ces opérations. Néanmoins, force est de constater que de très nombreuses « bases de données » biologiques ne sont encore que des ensembles de fichiers plus ou moins bien structurés. |
|
1. Jungfer K, Cameron G, Flores T. EBI : CORBA and the EBI databases in bioinformatics. In : Letovsky, ed. Databases and systems. New York: Kluwer Academic Publishers, 2000 : 245–54. 2. Baker PG, Goble CA, Bechhofer S, Paton NW, Stevens R, Brass A. An ontology for bioinformatics applications.Bioinformatic s 1999; 15 : 510–20 3. Ducournau R, Euzenat J, Masini G, Napoli A. Langages et modèles à objets : état des recherches et perspectives. Collection Didactique. Paris: INRIA, 1998. 4. Muller PA, Gaertner N. Modélisation objet avec UML. Paris: Éditions Eyrolles, 2000. 5. Baker PG, Brass A, Bechhofer S, Goble C, Paton N, Stevens R. TAMBIS : transparent access to multiple bioinformatics information sources. Proc Int Conf Intell Syst Mol Biol 1998; 6 : 25–34 6. Karp P. An ontology for biological function based on molecular interactions. Bioinformatics 2000; 16 : 269–85. 7. Karp P, Riley M. Representation of metabolic knowledge. Proc Int Conf Intell Syst Mol Biol 1993; 1 : 207–15. 8. Karp P. Pathway databases : a case study in computational symbolic theories. Science 2001; 293 : 2040–4. 9. Kanehisa M. Post-genome informatics. Oxford (GBR) : Oxford University Press, 2000. 10. Pittard AJ. Biosynthesis of aromatic amino acids. In : Neidhardt FC, et al., eds. Escherichia coli and Salmonella typhimurium: cellular and molecular biology, 2nd ed. Washington DC : ASM, 1996 : 458–84. 11. Page M, Gensel J, Capponi C, Bruley C, Genoud P, Ziébelin D. Représentation de connaissances au moyen de classes et d’associations : le système AROM. Actes du colloque Langages et Modèles à Objets (LMO), Mont Saint-Hilaire. Canada : Éditions Hermes, 2000 : 91–106. |