De multiples fonctions sont attribuées aux ARN. Les plus connues concernent la synthèse protéique : les ARN ribosomiques forment l’ossature des ribosomes et catalysent l’allongement des chaînes polypeptidiques, les ARN de transfert amino-acyés reconnaissent les codons des ARN messagers, l’ARN 7SL chez les eucaryotes, fait partie de la particule SRP (signal recognition particle) qui entraîne vers le réticulum endoplasmique les peptides signaux sortant des ribosomes. Des ARN interviennent dans la maturation posttranscriptionnelle des ARN eux-mêmes, des petits ARN nucléaires (snARN) sont impliqués dans l’épissage des pré-messagers eucaryotes et des petits ARN nucléolaires (snoARN) servent de guide pour modifier des nucléotides sur les autres ARN (→). Des ARN modulent spécifiquement la régulation de l’expression génétique : des micro-ARN antisens de 21 à 23 nucléotides limitent la traductibilité et la stabilité de transcrits [1, 2], d’autres, plus grands, répriment la synthèse de protéines spécifiques, c’est le cas de l’ARN OxyS qui contrôle la synthèse des protéines induites en réponse à un stress oxydant chez E. coli en réprimant la synthèse de la sous-unité sigma S de l’ARN polymérase et de l’activateur transcriptionnel fhlA (pour revue, voir [3]). Une série de travaux récents introduit un nouveau type de fonction : certains co-facteurs de la transcription sont des ARN.
(→) m/s 2001, n°6-7, p. 730
Des ARN non codants réglent la transcription de domaines chromosomiques voire de chromosomes entiers, c’est le cas du chromosome X dont il faut compenser le nombre de copies qui diffère entre mâles et femelles. Ainsi, les ARN roX activent la transcription du chromosome X chez les mouches mâles alors que l’ARN Xist inactive l’un des chromosome X chez les souris femelles (pour revue, voir [4]). Ces transcrits recouvrent les chromosomes X autour de leur site de transcription. Les ARN roX s’associent aux protéines MSL (male-specific lethal) de la mouche pour stimuler l’acétylation des histones. En revanche, le mode d’action de Xist chez la souris n’est pas connu, mais il est lui-même réglé négativement par un transcrit antisens, Tsix (→).
(→) m/s 2000, n°6-7, p. 818
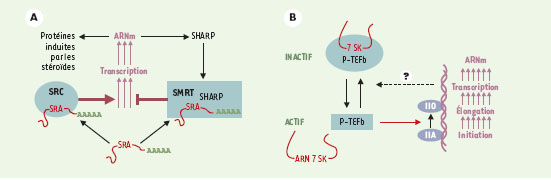
Des ARN modulent directement l’activité de facteurs de transcription spécifiques. Ainsi, dans l’œuf de xénope, l’association avec un (des) ARN maintiendrait dans le cytoplasme le facteur de transcription CBTF (CCAAT-box transcription factor) [5] et l’association avec des ARN messagers polyadénylés supprime la capacité de liaison à l’ADN du facteur de transcription Yin Yang 1 (YY1) [6]. En utilisant un crible double-hybride, l’équipe de Bert O’Malley (Houston, TX, USA), a découvert de façon inattendue un ARN, le SRA (ste-roïd receptor RNA activator), co-activa-teur de la transcription des gènes activés par les récepteurs des stéroïdes [7] Cet ARN polyadénylé présente de courtes phases de lecture ouverte, mais la destruction des codons potentiellement initiateurs de ces phases n’a pas de conséquence fonctionnelle. L’ARN SRA peut s’associer à une protéine à « boîte DEAD »1 du complexe co-activateur des récepteurs des stéroïdes [8]. Plus récem-ment, l’équipe de Ron Evans (La Jolla, CA, USA), a caractérisé une protéine (SHARP ou SMRT/HDAC1 associated repressor protein) qui se lie à l’ARN SRA et à SMRT, le co-répresseur des récepteurs stéroïdiens [9]. Selon que l’ARN SRA s’associe à SHARP ou au complexe co-activateur, il y aurait soit répression, soit activation de la transcription. Puisque l’expression de SHARP est induite par les stéroïdes, l’interaction de l’ARN SRA avec SHARP pourrait contribuer au rétrocontrôle de la réponse hormonale (Figure 1 A).
| Figure 1. Deux exemples d’ARN modulant la transcription. A. La transcription des gènes induits par les stéroïdes est activée par un complexe activateur (SRC) comprenant le récepteur des stéroïdes et l’ARN polyadénylé SRA. Cependant, cette transcription est inhibée lorsque le même ARN SRA s’associe au complexe répresseur SMRT à travers la protéine SHARP. Puisque la synthèse de SHARP est elle-même induite par les stéroïdes, l’interaction de SHARP avec l’ARN SRA pourrait contribuer à une rétro-inhibition de l’induction. B. La transcription des ARN messagers débute (initiation) avec l’ARN polymérase IIA (non phosphorylée), celle-ci doit ensuite être phosphorylée (IIO) par le facteur de transcription P-TEFb pour que les transcrits soient allongés (élongation). L’activité kinase du P-TEFb est inhibée par son association avec l’ARN 7SK, un petit transcrit de l’ARN polymérase III. Cette association est réversible, elle dépend de l’activité transcriptionnelle globale par un mécanisme encore inconnu, et pourrait constituer l’un des éléments d’une boucle de rétrocontrôle global de la transcription des ARN messagers. |
(→) m/s 1999, n°10, p. 1153
Des petits ARN règlent directement l’activité de la machinerie générale de transcription. Ainsi, le petit ARN 6S s’associe à l’ARN polymérase bactérienne et en diminue l’activité [10]. Chez les mammifères, un petit ARN nucléaire module l’efficacité de l’allongement des transcrits. En effet, deux complexes du facteur P-TEFb (positive transcription elongation factor b) viennent d’être isolés simultanément à partir de cellules en culture par le groupe de Qiang Zhou (Berkeley, CA, USA), et par notre équipe, à Paris (France) avec la collaboration de Tamás Kiss à Toulouse (France) [11, 12]. L’un de ces complexes est actif ; il est constitué de la kinase CDK9 et d’une cycline T. L’autre complexe, inactif, comprend en outre un petit ARN nucléaire transcrit par l’ARN polymérase III, le snARN 7SK. La dégradation in vitro de l’ARN 7SK convertit le complexe inactif en complexe actif (Figure 1B). Ces deux complexes sont en équilibre in vivo ; un traitement des cellules avec des inhibiteurs de la transcription ou une irradiation aux UV augmente la proportion de forme active du P-TEFb par un mécanisme encore inconnu. Le PTEFb phosphoryle le domaine carboxy-terminal (CTD) de l’ARN polymérase II, il estnécessaire pour transcrire la plupart des ARN messagers (→) [13], il serait recruté sur les complexes de transcription par les facteurs de transcription [14, 15]. Aucune fonction n’avait été proposée jusqu’ici pour le snARN 7SK. Pourtant, c’est un ARN abondant de 331 nucléotides, décrit depuis plus de 25 ans [16, 17]. Comme l’ARN 7SK est dissocié du P-TEFb en réponse à une inhibition, cette interaction pourrait constituer l’un des éléments d’une boucle de rétrocontrôle global de la transcription (Figure 1 B).
(→) m/s 1998, n°10, p. 167
L’existence d’un complexe cellulaire ARN/P-TEFb éclaire d’un jour nouveau l’activation transcriptionnelle du virus de l’immunodéficience humaine. En effet, le recrutement du P-TEFb sur le promoteur est une étape clé de cette activation (→), elle utilise la formation d’un complexe ribonucléoprotéique entre le P-TEFb, la protéine virale Tat et l’ARN TAR situé à l’extrémité 5’ du transcrit viral. Or, on peut obtenir une activation du promoteur du VIH, indépendante de TAR, en soumettant des cellules à des traitements qui perturbent globalement la transcription [18]. Puisque ceux-ci provoquent l’activation du P-TEFb, il est plausible qu’ils favorisent son recrutement sans l’assistance du complexe TAR/Tat.
(→) m/s 1999, n°10, p. 1173
Ces travaux illustrent l’existence de rétrocontrôles de l’efficacité de la transcription des ARN messagers, ils s’inscrivent dans une évolution très récente de la biologie moléculaire tendant à attribuer une importance fonctionnelle croissante aux ARN qui ne codent pas pour des protéines.