| |
| Med Sci (Paris). 2002 February; 18(2): 237–250. Published online 2002 February 15. doi: 10.1051/medsci/2002182237.L’annotation in silico des séquences génomiques Bio-informatique (1) Claudine Médigue,1 Stéphanie Bocs,1 Laurent Labarre,1 Catherine Mathé,2 and David Vallenet1 1URA 8030, Atelier de génomique comparative, Genoscope, 2, rue Gaston Crémieux, 91000 Évry, France 2Institut InterUniversitaire Flamand de Biotechnologie (VIB), Département de Génétique Végétale, K.L. Ledeganckstraat, 35, 9000 Gand, Belgique |
Le séquençage des génomes est aujourd’hui perçu comme un exploit technologique qui pourrait permettre, à terme, de guérir un grand nombre de maladies associées à des gènes. Déterminer la séquence complète d’un génome, c’est avant tout établir le catalogue des gènes qui sont nécessaires à la survie et à la reproduction d’un organisme vivant. Mais au-delà de ce catalogue, les projets de séquençage des génomes peuvent nous conduire au cœur du vivant, à condition toutefois que nous puissions comprendre les relations fonctionnelles entre les gènes et/ou leurs produits. De ce point de vue, la systématisation du séquençage ouvre une voie nouvelle à la découverte scientifique : les hypothèses sur les fonctions et le rôle des gènes sont de plus en plus issues de l’analyse in silico (c’est-à-dire une analyse entièrement réalisée au moyen de l’ordinateur). Cette analyse in silico permet alors de débuter une expérimentation biologique en laboratoire (c’est-à-dire une analyse in vitro ou in vivo). L’informatique va donc jouer un rôle clé au cours des différentes étapes de l’étude des génomes qui vont de l’acquisition à l’exploitation des données de séquences et à leur gestion « intelligente ». Cette dernière importante facette recouvre le développement de bases de données de nature très variée : les séquences et leurs caractéristiques, les informations sur l’ensemble des transcrits ou des protéines exprimées dans la cellule, les informations sur leurs interactions, ou encore sur les chemins métaboliques et les circuits de régulations mis en œuvre dans un organisme. Ces derniers aspects seront traités dans d’autres articles de ce lexique. La présente synthèse porte sur une activité située en aval de ces problématiques : l’annotation d’un génome brut. Elle est destinée à donner une idée générale de la façon dont le processus d’annotation est aujourd’hui conduit dans le cas des séquences d’organismes procaryotes et eucaryotes mais aussi, et surtout, de montrer que le chemin est encore long avant que nous puissions exploiter un jour pleinement toute l’information portée par ces longs textes génomiques. |
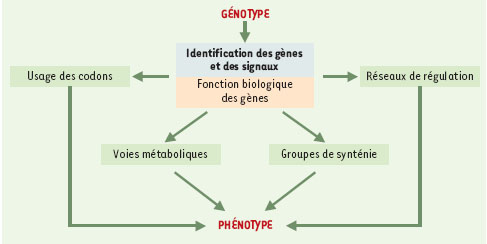
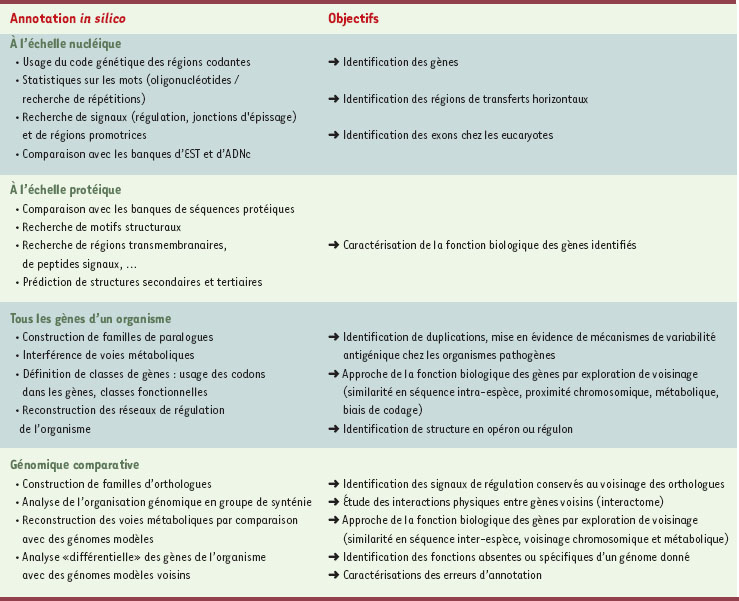
Qu’est-ce que l’annotation des génomes ? L’accès à l’information portée par un texte génomique s’est d’abord fait au moyen des techniques expérimentales de la génétique qui, à partir de caractères observables (phénotypiques), compare des types altérés (mutants) au type normal (sauvage). Les phénotypes observés n’avaient alors pas vraiment de réalité physique. Aujourd’hui, nous avons directement accès à ce support physique, l’ADN (donc le génotype d’un organisme), la séquence d’une région d’ADN étant alors obtenue avant même de caractériser les gènes qui y sont localisés. L’annotation d’une séquence génomique peut être abordée à différents niveaux d’analyse, une première phase incontournable consistant à identifier les gènes de l’organisme, c’est-à-dire à trouver leur localisation précise sur la séquence du génome. Dans une seconde étape, on cherche ensuite à assigner une (ou plusieurs) fonction biologique à chacun de ces gènes hypothétiques (Figure 1 et Tableau I). Cette seconde étape est généralement conduite par comparaison des séquences des gènes hypothétiques avec les séquences de gènes de fonction déjà connue. Les organismes dont le génome est aujourd’hui entièrement séquencé nous ont révélé que près de 40 % des gènes déterminés au cours de la première étape n’ont pas de fonction attribuée, soit parce qu’ils ne ressemblent à aucun gène connu, soit (pour près de la moitié d’entre eux) qu’ils ressemblent à d’autres gènes mais eux-mêmes de fonction inconnue. Avec le séquençage de nouveaux génomes, la proportion de gènes constituant des «familles conservées» de fonction inconnue tend à croître, ce qui suggère qu’elles remplissent des fonctions biologiques importantes. 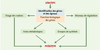 | Figure 1. Stratégies d’annotation in silico des génomes. L’annotation des séquences génomiques consiste à extraire, à partir d’outils informatiques, le maximum d’information des données de séquences afin de prédire des caractéristiques phénotypiques permettant de guider le travail expérimental. Dans une première étape, les régions codantes et les signaux (promoteurs et terminateurs de la transcription, ribosome binding site ou signal RBS) sont recherchés. Les gènes identifiés sont alors traduits en séquences peptidiques et la mise en œuvre de méthodes fondées sur la recherche de similarités avec les protéines répertoriées dans les banques de séquences permet de caractériser la fonction biologique de près de la moitié d’entre eux. On s’intéresse alors, dans un deuxième temps, aux relations qui lient ces objets biologiques : caractérisation des réseaux de régulation, des groupes de synténie, des voies métaboliques, et de l’usage des codons des gènes des organismes étudiés. |
Décrire l’organisation linéaire d’un génome constitue une première étape de l’annotation, parfois très laborieuse, et cependant loin d’être complète. Audelà de la simple succession des gènes sur un chromosome, on voudrait par exemple prédire également la compartimentation des protéines dans la cellule, identifier leur signaux d’adressage, caractériser les facteurs et les signaux qui contrôlent la transcription des gènes, etc. Une seconde étape de l’annotation consiste donc à identifier les relations entre les objets génomiques mis en évidence au cours des étapes précédentes (Tableau I). Ces relations peuvent être de nature très variée : des interactions physiques protéines/ADN, pro-téines/ARN et protéines/protéines, des réseaux de régulation de l’expression génique, des voies métaboliques, des synténies conservées entre organismes (voir glossaire), ou encore des groupes de gènes dont l’usage du code génétique est voisin (Figure 1). Avec le séquençage de génomes entiers, une abondante quantité d’informations devient potentiellement disponible pour mener à bien une analyse approfondie de l’ensemble de l’information génétique.
 | Tableau I. Niveaux d’analyse informatique des séquences génomiques. |
|
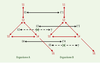
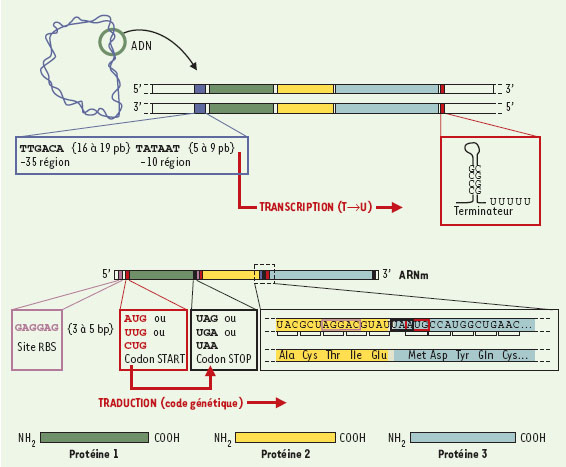
Le premier problème qui se pose consiste à repérer les gènes dans le texte brut du génome constitué par l’enchaînement des nucléotides. Le génome humain, environ 3 milliards de nucléotides, comporte probablement 30 000 à 35 000 gènes dont les parties codant pour des protéines ont une longueur moyenne d’environ 1 500 nucléotides. La proportion du texte qui serait ainsi associée à des fonctions protéiques représenterait seulement 1 % de la totalité du génome. On comprend alors mieux pourquoi les efforts ont longtemps porté sur l’étude de génomes beaucoup plus compacts comme ceux des bactéries ou encore de la levure. Les séquences de certains gènes étant très conservées chez toutes les espèces, le déchiffrage des génomes d’organismes inférieurs aide alors beaucoup à l’interprétation de ceux des organismes supérieurs. Structure des gènes Les séquences génomiques abritent plusieurs types de gènes : les gènes codant pour des protéines, mais aussi des gènes codant pour des ARN structuraux, molécules indispensables au processus de la traduction des ARN messagers en protéines. Il s’agit des ARN ribosomiques, constituants essentiels des sous-unités ribosomiques impliquées dans le processus de la traduction, et des ARN de transfert qui permettent d’établir la correspondance entre les codons d’un gène en cours de traduction et les acides aminés qui composent la protéine finale. Il existe aussi d’autres gènes codant pour de petits ARN fonctionnels qui interviennent dans le mécanisme d’excision des introns, ou qui sont impliqués dans la maturation d’ARN ribosomiques ou la régulation de l’expression génique. Dans la suite de cet article, nous focaliserons essentiellement notre attention sur les gènes codant pour les protéines. La structure des gènes codant pour les protéines est très différente chez les organismes procaryotes et eucaryotes. Chez les bactéries, les processus moléculaires fondamentaux se déroulent dans un même compartiment et sont le plus souvent simultanés (Figure 2). Les gènes sont constitués d’une seule pièce, et sont fréquemment organisés en opérons (c’est-à-dire en groupes de gènes exprimés simultanément sous le contrôle d’une protéine régulatrice). Les protéines alors produites sont souvent impliquées dans un même processus métabolique, ou peuvent constituer les différentes sous-unités d’une protéine multimérique qui présentera la fonction biologique requise. Chez les organismes eucaryotes, les étapes de transcription et de traduction ne sont que très exceptionnellement simultanées, la transcription se déroulant dans le noyau et la traduction dans le cytoplasme (Figure 3). Par ailleurs, les gènes eucaryotes présentent une structure morcelée : ils sont constitués de régions non codantes parfois très longues, les introns, qui alternent avec des portions effectivement traduites en protéines, les exons. 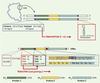 | Figure 2. Structures des gènes chez les organismes procaryotes. Les processus moléculaires associés à la réplication (ADN->ADN), à la transcription (ADN->ARN) et à la traduction (ARN->protéine) se déroulent dans un même compartiment représenté par la cellule de l’organime. Plusieurs signaux permettent la reconnaissance des zones à transcrire, à savoir la région promotrice sur laquelle se fixe l’ARN polymérase pour déclencher la transcription (boîtes -35 et -10) et une région de terminaison (dite indépendante de rho) qui correspond à une structure secondaire en tigeboucle au niveau de laquelle l’ARN polymérase se décroche (Terminateur). Les gènes sont le plus souvent rassemblés en opéron, c’est-à-dire un groupe de gènes exprimés en même temps sous le contrôle d’une protéine régulatrice. Les mécanismes de transcription et de traduction se produisent de façon simultanée : dès que le ribosome peut, au niveau du site RBS (ribosome binding site), se fixer sur la molécule d’ARN messager en cours de fabrication, la traduction de la protéine est mise en route avant même que la transcription soit achevée. Cette traduction débute au niveau du codon d’initiation formé le plus souvent des trois lettres AUG (et plus rarement CUG ou UUG), et se termine par un des trois codons de terminaison universels, UAA, UAG et UGA. |
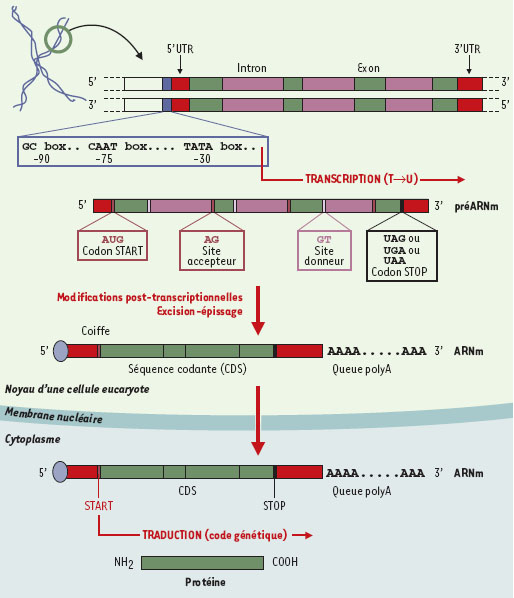
L’analyse informatique visant à identifier les gènes d’un organisme combine des méthodes qui permettent d’une part de caractériser les signaux nécessaires à l’expression des gènes (c’est-à-dire des motifs particuliers dans la séquence d’ADN, Figures 2 et 3) et d’autre part, de prédire les régions codantes. Recherche de signaux Avant même de se poser la question de la localisation d’éléments fonctionnels dans une séquence génomique (sites de fixation des ribosomes, régions promotrices, etc), il est nécessaire de donner une description précise des signaux que l’on recherche. Une description très simple repose sur la notion de «motif consensus» qui constitue une sorte de résumé du signal recherché. Par exemple, le motif consensus [AG]GAG[CG] signifie que le signal à chercher doit être de la forme : « une base A ou G, suivi de G, de A, de G puis des bases C ou G ». Généralement, le motif consensus est établi à partir d’un ensemble de séquences bien caractérisées dont on sait qu’elles contiennent le signal à caractériser. Celui-ci est appelé jeu ou ensemble d’apprentissage et est généralement construit à partir de résultats expérimentaux. La mise en œuvre de programmes d’alignements multiples sur ce jeu d’apprentissage permet de caractériser des segments de séquences conservés et d’en déduire le (ou les) motifs consensus. Une autre description, plus fine que la précédente, repose sur la notion de «tableau poids-positions». Un tel tableau est constitué de quatre lignes correspondant aux quatres bases (A,C,G et T) et d’autant de colonnes qu’il y a de positions dans le signal que l’on souhaite représenter. L’entrée T(b,i) de ce tableau indique simplement la fréquence relative d’apparition de la base b à la position i du motif, observée sur l’ensemble d’apprentissage [1], c’est-à-dire une mesure de l’importance relative de cette base dans le motif. Lorsque les séquences de l’ensemble d’apprentissage sont suffisamment proches ou lorsque le motif recherché est très conservé, l’alignement ne pose pas de problème et la définition du signal (sous forme de motif consensus ou de tableau poids-positions) est généralement conduite manuellement. Lorsque ces conditions ne sont pas satisfaites, la «découverte» du motif dans l’ensemble d’apprentissage devient plus délicate et nécessite la mise en œuvre d’algorithmes dédiés (on parle alors «d’inférence de motif») [2, 3]. Les étapes de construction de l’ensemble d’apprentissage (rôle du biologiste) et de choix de l’algorithme utilisé pour inférer le consensus (rôle du bioinformaticien), constituent les étapes limitatives du processus de caractérisation de signaux. Rechercher ensuite les occurrences du motif (consensus ou tableau poidsposition) sur une séquence d’ADN ne présente en réalité pas de difficulté majeure. Enfin, il convient de remarquer que d’autres représentations, plus sophistiquées, ont également été proposées, notamment celles fondées sur les «modèles de Markov» (voir glossaire) [4]. D’une manière générale, ces représentations constituent un modèle du signal recherché. Plus ce modèle est sophistiqué (et donc proche de la réalité) plus le nombre de paramètres augmente et plus la question de l’estimation de ces paramètres sur l’ensemble d’apprentissage (de taille nécessairement limité) se pose avec acuité. Enfin, il est important de noter que les signaux dont nous avons parlé jusqu’ici sont essentiellement de nature «lexicale» (seule compte l’identité des bases en chacune des positions). Certains signaux peuvent être de nature plus complexe. Tel est le cas, par exemple, des signaux «structuraux» dans lesquels intervient non pas l’identité des bases mais leur facilité à former une structure secondaire précise (généralement au niveau de l’ARN). Chez les organismes procaryotes, les signaux de terminaison de la transcription (indépendante de rho) en fournissent un exemple. Ce motif étant bien décrit (Figure 2) et conservé entre les espèces procaryotes (eubactéries), l’utilisation du programme Pétrin [5] permet d’obtenir une bonne idée du nombre de séquences transcrites dans une séquence génomique. Chez les organismes eucaryotes, les séquences associées aux différents signaux sont souvent trop courtes et/ou trop peu conservées pour constituer un critère absolu. Les signaux les plus «forts» seraient les sites donneurs et accepteurs du mécanisme d’excision des introns : les introns commencent généralement en 5’ par GT et finissent en 3’ par AG (Figure 3). Cependant, le nombre de mots AG et GT dans une séquence génomique est très important, et la seule présence de ces deux di-nucléotides n’indique donc pas nécessairement la présence d’un intron. On a alors recours à une combinaison de stratégies prenant en compte d’autres séquences plus ou moins conservées voisines des AG et GT, les caractéristiques des exons selon leur position dans le gène, ainsi que d’autres signaux comme les sites de fixation des facteurs de transcription, les îlots CpG situés en amont des gènes, ou encore le site de reconnaissance localisé en aval des gènes, nécessaire à la poly-adénylation de l’ARN messager (Figure 3). De nombreuses méthodes informatiques ont été développées pour rechercher les régions promotrices de la transcription [6], pour identifier spécifiquement les sites d’épissage [7, 8], ou encore les sites d’initiation de la traduction [9]. Elles n’ont donc pas la prétention de déterminer la structure des gènes, mais seulement d’affecter à chaque site potentiel un indice de fiabilité. 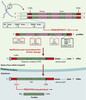 | Figure 3. Structure des gènes chez les organismes eucaryotes. Si la réplication et la transcription se déroulent dans le noyau de la cellule, la traduction, elle, opère au sein du cytoplasme. L’ARN polymérase utilise des signaux particuliers sur la séquence d’ADN pour déclencher le processus de la transcription qui conduit à la fabrication d’un pré-ARN messager contenant les régions non codantes ou introns, et les régions codantes ou exons. Les introns sont alors excisés avant la traduction grâce à un ensemble moléculaire complexe (le splicéosome) qui reconnaît plusieurs signaux au niveau de la séquence: les sites donneurs et accepteurs à la jonction des exons et des introns, et le site de branchement responsable de la structure en lasso que prend l’intron excisé. Un autre signal primordial chez les eucaryotes est celui qui spécifie le codon d’initiation de la traduction, car il n’y a pas de séquence RBS en amont pour faciliter sa reconnaissance par les ribosomes. Aussi une «coiffe» (ou molécule de guanine monophosphate) est ajoutée à l’extrémité 5’ de la molécule d’ARN messager mûre. Elle protège cette dernière des attaques par les nucléases et permet une meilleure reconnaissance de l’extrémité 5’ par le ribosome. Enfin, la séquence qui pourrait servir de repère de fin de gène à l’ARN polymérase chez les eucaryotes (séquence AATAAA) serait aussi le signal qui commande l’ajout d’une queue de plusieurs dizaines de nucléotides A à l’extrêmité 3’ de l’ARN messager. Cette queue polyA joue un rôle dans la stabilité de l’ARNm, dans son mécanisme de sortie du noyau, et dans la stimulation de l’initiation de la traduction. |
Prédiction de régions codantes Une protéine étant codée par une succession de codons, une stratégie simple pour repérer les gènes, au moins chez les bactéries, consiste à rechercher les plus longues phases ouvertes de lecture (ORF : open reading frame), c’est-à-dire des régions entre deux codons de terminaison de la traduction, en phase. Au risque d’ignorer les plus petits gènes, on ne considère généralement que les ORF d’au moins 300 nucléotides, la probabilité qu’une telle ORF soit due au hasard devenant alors suffisamment faible chez la plupart des bactéries. Malheureusement, ces critères ne suffisent pas toujours à localiser correctement les régions codantes et, en tout état de cause, ne permettent pas d’identifier de manière certaine le codon correct de démarrage de la traduction. On doit alors utiliser une propriété statistique des régions codantes qui permet de mieux les discriminer des régions non codantes. Il a en effet été observé que la distribution des «mots», par exemple la fréquence d’apparition d’oligonucléotides de longueur 4, 5, ou 6, est différente dans les régions codantes et les régions non codantes : ces régions de l’ADN présentent donc des différences de «style» qui sont mises à profit de façon très efficace par les programmes fondés sur l’utilisation des chaînes de Markov, tels que GeneMark [ 10] et Glimmer [ 11]. La discrimination entre les phases ouvertes codantes et les autres est facilitée, chez les organismes procaryotes, par le fait que ces phases sont généralement longues et dépourvues d’introns. Chez les organismes eucaryotes, la situation est plus complexe. Les régions codantes des exons sont parfois trop courtes pour qu’une simple tendance statistique permette de les repérer. Par ailleurs, il n’y a pas de raison pour que leur taille soit un multiple de trois ni qu’elles soient comprises entre deux codons de fin de traduction. La structure fine des gènes, c’est-à-dire leur découpage précis en exons et introns ainsi que leurs extrêmités 5’ et 3’, est prédite par des programmes qui combinent généralement un ensemble de méthodes. Plusieurs mesures sont utilisées pour rendre compte des différences entre les régions codantes et non codantes : des méthodes probabilistes tels que les modèles de Markov déjà mentionnés ci-dessus [12, 13], des méthodes de discrimination tels que les réseaux neuronaux [14] ou les arbres de décision [15]. Ce type de technique a pour objectif de trouver les critères permettant de discriminer deux ensembles de données : les exemples (tels que des séquences correspondant à des sites d’épissage fonctionnels), et les contre-exemples (des séquences qui ressemblent à des sites d’épissage mais qui n’en sont pas). La mise en œuvre de ces techniques va permettre, en général, d’identifier tous les éléments constitutifs des gènes (exons, sites d’épissage, codons initiateurs) individuellement. L’assemblage de ces éléments, pour reconstituer la structure globale d’un gène, est généralement réalisé par d’autres programmes, fondés, par exemple, sur des modèles de Markov à états cachés [16,17]. Outre les approches «intrinsèques» que nous venons de mentionner (c’est-à-dire ne mettant en œuvre que les propriétés statistiques de la séquence d’ADN chromosomique), on peut également recourir, lorsqu’elles sont disponibles, à des informations expérimentales concernant les gènes exprimés dans la cellule. Par exemple, les ADN complémentaires (ADNc) obtenus par transcription inverse de l’ARN messager ne contiennent que les parties exoniques d’un gène. Une autre source essentielle d’information se trouve dans les EST (expressed sequence tags), obtenus par séquençage systématique des extrémités d’ADNc. La mise en œuvre de programmes de recherche de similarité, à partir de banques d’ADNc [18] ou d’EST [19], permet alors de mettre en évidence des régions de la séquence génomique analysée contenant des exons, et par là même les frontières intron-exon deviennent plus aisées à identifier. La nature de cette similarité pose cependant des problèmes car les séquences d’EST sont non seulement très courtes, mais aussi parfois, de plus mauvaise qualité que l’ADN génomique. Une autre solution consiste alors à comparer les six séquences peptidiques, obtenues après traduction de la séquence génomique sur les six cadres de lecture possibles, aux protéines répertoriées dans une banque de protéines connues telle que SwissProt [20]. Un des atouts de cette approche réside dans le fait que la comparaison entre protéines a toute les chances d’être plus sensible que la comparaison ADN/ADN. Les séquences protéiques sont en effet mieux conservées (dégénérescence du code génétique), et la taille plus importante de leur alphabet rend les identités fortuites moins probables. Néanmoins, les méthodes qui reposent sur ce type de comparaisons ne seront fructueuses qu’à la condition évidente qu’au moins une séquence suffisamment proche soit présente dans les banques. Les limites des approches actuelles L’identification des gènes constitue la partie la plus automatisée du processus d’annotation, en particulier chez les organismes procaryotes. Et même si des progrès restent à faire [ 21– 23], les procédures actuelles fournissent des résultats tout à fait honorables (on estime que la probabilité de «manquer» un gène bactérien est inférieure à quelques pour cent). Le cas des organismes eucaryotes est, en revanche, beaucoup plus délicat. L’évaluation des différents programmes de prédiction de gènes eucaryotes a révélé que, si plus de 90 % des nucléotides sont correctement identifiés comme étant codant ou non codant, la détermination exacte des bornes des exons et leur assemblage en une séquence codante complète et correcte est une toute autre affaire [24, 25]. Un premier problème auquel sont confrontées toutes les méthodes de prédiction est l’extrême diversité de la structure des gènes des organismes eucaryotes : en effet, si la taille moyenne d’un gène chez l’homme est de 27 kb (régions introniques incluses) [26], le gène de la dystrophine par exemple, mesure plus de 2 millions de nucléotides dont 99 % sont des introns de taille parfois supérieure à 100 kb. On comprend aisément que, dans cette situation, les programmes aient tendance à prédire plusieurs gènes au lieu d’un seul. À l’inverse, deux gènes peuvent être vus comme un seul, notamment lorsque la séquence inter-génique qui les sépare est courte voire inexistante, ou dans le cas de gènes se recouvrant. Par ailleurs, à chaque étape de la synthèse des protéines sont associées des possibilités alternatives pouvant conduire à la synthèse d’une protéine différente : promoteur alternatif, épissage alternatif, polyadénylation alternative et initiation alternative de la traduction. Les programmes de prédiction ne savent pas gérer ces différents cas et ne prédisent généralement qu’une seule structure «optimale» de gène. Enfin, d’autres cas particuliers, peu ou pas pris en compte dans les stratégies de recherche de gènes eucaryotes, sont les sites d’épissage non-standards (ne présentant pas le dinucléotide consensus GT ou AG) ou encore les cas dans lesquels la traduction ne débute pas sur un codon AUG. Ces exemples montrent à quel point la coordination de nombreux mécanismes biologiques est encore mal comprise. Le problème de la détection des gènes eucaryotes est donc encore loin d’être résolu, bien que l’on sache qu’il doit exister une solution : la cellule, elle, ne se trompe pas et réalise l’épissage des exons avec une grande efficacité et une grande précision. Un des problèmes majeurs de l’annotation in silico consiste à élaborer des prédictions à partir de données de séquences analysées sous une forme linéaire et « statique », alors que dans la cellule, cette même séquence présente des conformations bien plus complexes (secondaires et tertiaires), et se trouve placée dans un milieu «dynamique». |
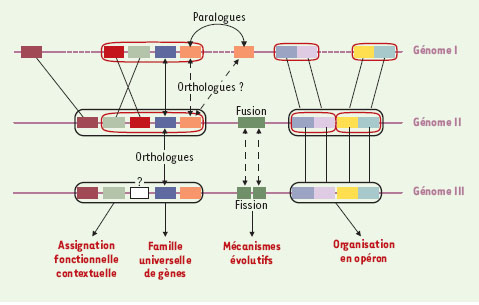
Du gène à la fonction biologique Une fois que les gènes sont repérés et délimités sur la séquence, il convient d’affecter une fonction à la protéine correspondante. L’étape de prédiction de la fonction biologique des gènes constitue en réalité le goulot d’étranglement le plus important de la génomique, la majeure partie de l’information étant perdue lorsqu’un gène ne peut pas être décrit correctement. Recherche de similarités On procède donc tout d’abord par similarité, en comparant la séquence de la protéine hypothétique avec des protéines de fonctions connues et de séquences voisines. Les programmes de recherche de similarités dans les banques de séquences du type BLAST permettent de rendre compte de « ressemblances » entre les séquences [ 27] et d’opérer un transfert, par similarité, de la fonction biologique présumée. Mais à partir de quel seuil de ressemblance entre leurs séquences peut-on considérer que deux protéines ont probablement la même fonction? Malheureusement, trop souvent, une similarité seulement partielle entre les séquences produit une annotation du type putative kinase par exemple. De proche en proche, on risque ensuite de propager cette information à d’autres protéines ressemblant de moins en moins à la première. D’une manière générale, le transfert d’information issue de l’analyse d’une protéine, même bien étudiée, est à considérer avec la plus grande prudence car, d’une part une similarité en séquence n’implique pas nécessairement une similarité de la structure protéique ou de la fonction et, d’autre part, l’annotation répertoriée dans les banques peut être incomplète ou bien fausse. Une telle annotation sera alors utilisée à tort pour les annotations ultérieures d’autres génomes, amplifiant ainsi l’erreur initiale. L’architecture modulaire de nombreuses protéines (c’est-à-dire composée en domaines protéiques) rend nécessaire le recours aux banques de motifs. Plusieurs banques de ce type ont été constituées. La plus ancienne, PROSITE [28], recense les motifs consensus caractéristiques de familles de protéines ayant une activité biologique voisine. Les domaines protéiques sont généralement répertoriés sous la forme de séquences consensus ou de tableaux poids-positions (comme cela a été décrit précédemment pour les motifs nucléiques). L’une des banques les plus utilisée à ce jour est PFAM, fondée sur la comparaison systématique de toutes les séquences protéiques de la banque SwissProt [29]. Il existe presque autant de programmes de recherche de domaines protéiques sur une séquence qu’il y a de banques de motifs différentes. Un effort de «standardisation» a été récemment entrepris pour définir les formats de ces banques et les «styles» d’annotation destinés à décrire les fonctions biologiques [30]. L’approche par motifs ne garantit, toutefois, pas plus que la comparaison de séquences deux à deux de l’effet de propagation des erreurs précédemment évoquée. En effet, de nombreuses protéines sont formées par l’assemblage de plusieurs domaines, plus ou moins conservés ou bien même remaniés dans d’autres protéines non apparentées. Ces mécanismes évolutifs sont à la source même de prédictions erronées, et les banques de motifs protéiques sont, dans ce sens, à utiliser également avec la plus grande prudence. Finalement, connaître la succession des acides aminés d’une séquence protéique (sa structure primaire) ne suffit pas pour comprendre pleinement son rôle biologique et ses interactions avec d’autres protéines ou avec ses substrats. Ces chaînes se replient sur elles-mêmes et adoptent des conformations secondaires puis tertiaires qui confèrent à la protéine ses propriétés. Si les méthodes de détection de régions transmembranaires, de peptides signaux, et de structures secondaires conduisent à des résultats généralement satisfaisants [24], prédire la structure tertiaire d’une protéine reste une entreprise beaucoup plus ardue. ll existe une banque de données très précieuse, la Protein Data Bank [31], répertoriant les coordonnées atomiques de plus de 1 500 protéines de séquences significativement différentes. Cet échantillon permet de mettre en œuvre des méthodes de modélisation moléculaire qui ont pour objectif de construire un modèle structural en comparant la séquence de la molécule étudiée avec celle des protéines de conformation 3D connue. Cette dernière approche reste délicate car, sauf cas particulier (par exemple les protéines membranaires), la liaison entre les classes de repliements et la fonction de la protéine n’est pas toujours évidente. Exploration des voisinages C’est une évidence aujourd’hui : un génome n’est pas «un sac de gènes» mais un ensemble ordonné et structuré d’informations qui permet le bon fonctionnement de la cellule et qui évolue en fonction des besoins adaptatifs des organismes. Notre vision a de fait évolué vers la notion de réseaux de gènes qui répondent de façon complexe aux stimulus de l’environnement. Aussi, une dernière phase de l’annotation des génomes consiste-t-elle à identifier les relations entre les objets génomiques caractérisés au cours des étapes précédentes, à savoir les gènes, les protéines et les éléments de régulation. S’il est tout naturel de prendre les gènes comme objets centraux de l’analyse in silico, une exploration plus globale consiste à trouver tous les voisins des gènes caractérisés. Chaque voisinage, c’est-à-dire l’identification d’objets qui partagent un espace donné, est destiné à apporter un éclairage spécifique sur le gène étudié, et à donner des éléments pour la recherche de sa fonction [ 32]. Un voisinage naturel est la proximité sur le chromosome. Chez les organismes procaryotes, il existe une structure de contrôle coordonnée, l’opéron, qui montre que des gènes situés à proximité les uns des autres peuvent être reliés fonctionnellement (Tableau I). Plusieurs bases de données contiennent des informations structurales sur la régulation de l’expression génique, signalant par exemple l’existence d’un site de fixation d’un facteur de transcription dans la région promotrice d’un gène, ou définissant un domaine polypeptidique impliqué dans les interactions protéines-protéines ou protéines-ADN [33, 34]. Il existe toutefois de nombreuses unités de transcription qui ne comprennent qu’un seul gène, et les organismes eucaryotes n’organisent généralement pas la transcription de leurs gènes sous la forme d’opérons. L’analyse de la ressemblance entre les gènes ou plus souvent, les produits des gènes, permet d’explorer un autre type de voisinage. La création de familles de paralogues ou de familles d’orthologues (voir glossaire) constitue des exemples d’exploration de l’espace de similarité en séquence entre les protéines. Les bases de données COG [35], WIT [36], ColiPage [37], ou encore HOBACGEN [38] ont pour objectif de décrire de telles familles. Cette étape est un point de départ vers la carac-térisation de groupes de synténie conservés entre plusieurs génomes, c’est-à-dire des ensembles de gènes dont les positions respectives ont été conservées au cours de l’évolution (voir glossaire) (Figure 4). Il est en effet remarquable de constater que, dans de nombreux génomes procaryotes, il existe une corrélation très forte entre cette organisation synténique et l’interaction physique entre les produits des gènes voisins [39]. Les groupes de synténie sont définis selon des critères plus ou moins stricts, liés à la similitude des séquences, la conservation de l’ordre des gènes sur le chromosome, la distance respective des gènes les uns par rapport aux autres, etc (Figure 4). Plusieurs approches ont été développées, parmi lesquelles WIT [36] et STRING [40], mais les correspondances multiples entre gènes ne sont pas toujours correctement prises en compte. D’un point de vue fonctionnel, des enzymes peuvent être voisines parce qu’elles utilisent le même substrat, produisent le même produit, ou encore se succèdent dans une même voie métabolique. L’exploitation de données métaboliques de référence [41, 42] constitue alors un outil puissant non seulement parce qu’elle permet de renforcer les résultats de l’analyse de similarité, mais aussi parce qu’elle autorise la découverte de fonctions ou voies métaboliques alternatives (Figure 5). Il existe, enfin, des voisinages plus complexes qui produisent des résultats parfois particulièrement riches. Les gènes sont en effet liés par leur façon «d’utiliser» le code génétique. Les codons synonymes (par exemple, les 4 codons CGU, CGC, CGA et CGG correspondent à l’acide aminé arginine) ne sont pas utilisés avec la même fréquence, et cette utilisation différentielle des codons (ou biais de codage) est caractéristique de chaque espèce. Ainsi, la connaissance du biais de codage de gènes déjà identifiés chez l’organisme étudié va-t-elle permettre de mieux prédire de nouveaux gènes du même organisme. 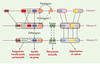 | Figure 4. Intérêts de l’analyse des groupes de synténie bactérienne. Des similarités significatives (ou parentés) entre gènes, au sein d’une même espèce ou entre plusieurs espèces, vont permettent d’inférer certaines propriétés aux protéines codées. Ainsi, la comparaison du génome III aux génomes I et II révèle que le premier ensemble constitué de cinq gènes contigus contient 4 gènes dont les orthologues ont une même organisation locale dans le génome II, et une organisation proche dans le génome I. On peut envisager que cet ensemble de gènes représente une famille de gènes universelle et que le gène situé au centre de cette famille (en blanc) a probablement une fonction proche du gène rouge situé au même endroit dans le génome II. Ce type d’analyse permet aussi d’identifier des mécanismes évolutifs résultant de la fusion/fission de gènes ainsi que des opérons conservés entre différentes espèces. |
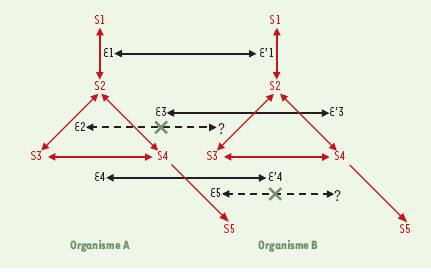
 | Figure 5. Reconstitution de voies métaboliques. L’ensemble des enzymes caractérisées au cours de la première étape d’annotation autorise une première reconstitution des voies métaboliques de l’organisme étudié (B), le plus souvent par référence aux voies métaboliques d’un organisme (A) proche et bien étudié. Ainsi, dans cet exemple, aucun gène homologue à ceux qui codent pour les enzymes E2 et E5 ne sont retrouvés dans le génome de l’organisme B. Dans l’hypothèse où l’annotation de premier niveau est correcte et exhaustive, cette observation suggère que les enzymes E2 et E5 n’existent pas chez l’organisme B qui utilise alors des voies métaboliques alternatives à découvrir. |
Idéalement, l’analyse de ces relations et voisinages devrait faire partie du processus complet d’annotation. Cependant, une telle exploration nécessite d’autres études expérimentales et informatiques, et les outils qui apparaissent tout juste dans ce domaine (qu’il s’agisse de méthodes informatiques ou de bases de données spécialisées) sont actuellement encore très dispersés dans les différents laboratoires. |
Les plates-formes d’annotation Au milieu des années 1990, face à la croissance du nombre de séquences, il devenait tentant de développer et d’utiliser des méthodes automatisées d’analyse. Or, l’annotation automatique des séquences génomiques n’est ni aisée, ni fiable, surtout chez les organismes eucaryotes. Pour certain d’entre eux, on estime qu’environ 50 % des prédictions de gènes reportées dans les banques comportent au moins une erreur. Ainsi, si le développement des premières plates-formes d’annotation reposait sur la mise en œuvre strictement automatique de programmes informatiques [43–45], de nombreux environnements de nature plus interactive leurs sont aujourd’hui préférés. Ces environnements proposent, en particulier, une représentation graphique des résultats d’analyse dont le but est de faciliter l’expertise finale du biologiste [46–50] (Tableau II). Les attributions fonctionnelles automatiques combinent de façon souvent astucieuse plusieurs résultats d’analyse afin d’attribuer une fonction unique à la protéine analysée : elles se heurtent alors au problème de l’accumulation des erreurs d’annotation dans les banques de séquences, mais aussi à l’organisation souvent modulaire des protéines, conduisant alors à des annotations incomplètes voire fausses [51]. Qu’il s’agisse de l’analyse de séquences génomiques procaryotes ou eucaryotes, il semble absolument nécessaire que ces annotations fonctionnelles soient validées, au cas par cas, par des experts biologistes. Il apparaît en effet clairement que, parmi l’ensemble des génomes de microorganismes aujourd’hui disponibles, la qualité des annotations est supérieure dans les laboratoires qui mettent en œuvre des interfaces graphiques destinées à l’interprétation méticuleuse des résultats bruts de méthodes informatiques [21]. Par exemple, le logiciel Artemis, développé au Sanger Centre (Tableau II), dispose d’une interface graphique très conviviale permettant, à partir des résultats de plusieurs méthodes, d’annoter chacun des objets caractérisés (CDS, introns et exons, etc.). Quoiqu’il en soit, les annotations seront inévitablement de qualité variable selon les programmes utilisés, les données de séquences disponibles au moment de l’annotation, mais aussi selon les annotateurs. De ce point de vue, il paraît essentiel que l’effort d’annotation soit de plus en plus placé sous la responsabilité de groupes d’experts de la communauté, et non plus sous celle d’un laboratoire ou d’un petit groupe d’annotateurs. On assiste ainsi à la mise en place de groupes et/ou projets d’annotation de génomes dont les résultats sont accessibles sur le web (pour l’instant, il est vrai, essentiellement en consultation). Pour le génome humain par exemple, le Sanger Centre développe le projet Ensembl, et l’Institut Suisse de Bio-informatique, le projet HPI (Tableau II). Finalement, l’hétérogénéité des informations disponibles dans les banques rendant très difficile la recherche d’information et les conclusions sur l’annotation fonctionnelle d’un gène, la nécessité d’une standardisation est aujourd’hui reconnue [30]. Des efforts vont dans ce sens, avec notamment la mise en place de plusieurs projets : Gene Ontology Project, Gene Feature Format et GAME (Tableau II).  | Tableau II. Exemples de plates-formes et de projets d’annotation de génomes procaryotes et eucaryotes. |
|
Conclusions et perspectives L’identification de la fonction des gènes nécessite la combinaison de plusieurs approches expérimentales qu’elles soient informatiques (analyse in silico) ou bien de nature biochimique ou génétique (analyses in vivo et in vitro). Ces différentes approches sont clairement complémentaires, les conclusions d’une approche pouvant être confirmées ou infirmées par l’autre. Les analyses in silico doivent en particulier guider l’interprétation de résultats expérimentaux, mais aussi suggérer de nouvelles expériences. Pouvoir prédire la fonction biologique d’un nombre important de gènes ouvre au chercheur un champ d’exploration plus important, par exemple dans le choix d’une stratégie d’inactivation des gènes: interruption de tous les gènes un par un, ou des gènes appartenant à une catégorie fonctionnelle donnée. Enfin, il nous semble important d’insister sur le fait que l’annotation ne se limite pas à l’étude de la séquence génomique. La génétique inverse, la génomique fonctionnelle et structurale, l’étude du transcriptome, du protéome, et du métabolome sont également des sources extraordinaires de connaissances nouvelles, indissociables de l’étude du génome. Il apparaît de plus en plus nécessaire d’intégrer ces différentes sources d’information au sein d’environnements informatiques «globaux» permettant de croiser, confronter et recouper ces sources afin d’essayer de franchir un petit pas supplémentaire vers ce qui constitue l’unité du vivant. |
Nous remercions vivement A. Danchin, P. Rouzé, F. Képes et M. Roux-Rouquier pour leur lecture attentive de ce manuscrit et leurs commentaires éclairés. |
| Chaîne de Markov | Une chaîne de Markov est un modèle probabiliste permettant de représenter des dépendances entre les observations successives d’une variable aléatoire. Dans le cas d’une séquence d’ADN, qui nous intéresse ici, cette variable représente la nature de la base nucléique (c’est-à-dire A,C,G ou T) et les observations successives correspondent aux positions successives sur la séquence (schéma ci-contre). Dans une chaîne de Markov d’ordre k, la probabilité d’apparition d’une base donnée en une position i ne dépend que de la nature des k bases qui la précédent (le cas k=0 correspond donc a l’hypothèse d’indépendance des positions et la figure représente le cas k=4). Les paramètres du modèle sont constitués de l’ensemble des probabilités conditionnelles (par exemple, sur la figure, la probabilité d’émettre A en position i sachant qu’il est précédé de GCAT). Ces paramètres sont généralement différents pour des régions de l’ADN codant pour des protéines (en raison de l’usage des codons) et pour des régions non codantes. C’est cette propriété qui est mise à profit pour discriminer les deux types de régions.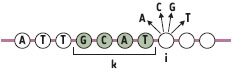 | | Homologie, orthologie et paralogie | On dit de deux gènes qu’ils sont homologues lorsqu’ils dérivent d’un gène ancestral commun. Suivant la définition proposée par Fitch [52], on distingue alors deux cas suivant que l’événement phylogénétique le plus récent séparant les deux gènes est une spéciation (apparition des deux espèces) - on dit alors que les deux gènes sont orthologues - ou une duplication (apparition de deux copies du gène) - on dit alors que les deux gènes sont paralogues. Le schéma de la page suivante (adapté de [53]) représente un arbre de gènes. Les cercles (Sp1 et Sp2) représentent un événement de spéciation (la couleur des branches correspond à une même espèce) et les carrés (Dup1 et Dup2) représentent un événement de duplication génique. Les gènes R1 et S1, par exemple, sont orthologues (de même que R2 et S2 ou R2 et S3). En revanche, S2 et S3 sont paralogues (de même que R1 et S2 ou R1 et S3 par exemple).Cet exemple permet de mettre en évidence deux propriétés importantes des relations d’orthologie/paralogie : 1) elles ne sont généralement pas transitives (par exemple 52 est orthologue à R2, R2 est orthologue à 53 mais S2 et S3 ne sont pas orthologues) et 2) elles ne sont généralement pas bijectives : c’est-à-dire qu’un gène, dans une espèce, peut présenter plusieurs orthologues dans une autre espèce (par exemple R2 est orthologue à S2 et S3 et H1 est orthologue à n’importe quel Ri et Si).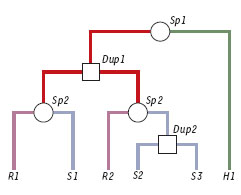 Enfin, il convient de remarquer que les définitions précédentes sont de nature essentiellement phylogénétique et ne font aucune hypothèse quant à la similarité des séquences ou à la fonction des gènes concernés (même si de telles hypothèses doivent parfois être posées pour parvenir à reconstruire l’histoire évolutive d’un gène ou d’une famille de gènes). Ainsi, l’emploi du terme homologue pour indiquer que deux gènes présentent des séquences similaires doit être considéré comme une impropriété. De même, le fait que deux gènes orthologues devraient présenter la même fonction doit être, le plus souvent, considéré comme une hypothèse de travail. Enfin, il convient de remarquer que les définitions précédentes sont de nature essentiellement phylogénétique et ne font aucune hypothèse quant à la similarité des séquences ou à la fonction des gènes concernés (même si de telles hypothèses doivent parfois être posées pour parvenir à reconstruire l’histoire évolutive d’un gène ou d’une famille de gènes). Ainsi, l’emploi du terme homologue pour indiquer que deux gènes présentent des séquences similaires doit être considéré comme une impropriété. De même, le fait que deux gènes orthologues devraient présenter la même fonction doit être, le plus souvent, considéré comme une hypothèse de travail. | | Synténies « bactériennes » | On dit de deux gènes d’un même organisme qu’ils sont en synténie s’ils sont portés par le même chromosome. Étant donné deux organismes A et B et deux gènes en synténie dans A, on dit que cette synténie est conservée si les orthologues des deux gènes dans l’espèce B sont également en synténie.Chez les bactéries, qui ne possèdent généralement qu’un seul chromosome, les définitions précédentes n’ont pas grand intérêt puisque, par définition, tous les gènes sont en synténie dans une espèce et en synténie conservée entre deux espèces. Dans ce cas, on parle alors de «synténie bactérienne» pour indiquer qu’un ensemble de gènes présente la même organisation locale dans une espèce A que leurs orthologues dans une espèce B. L’exemple typique est celui d’un opéron conservé entre deux espèces (Figure 5). Il faut noter que la définition précise du terme «organisation locale» diffère souvent d’un auteur à l’autre. |
|
1. Stormo GD. Consensus patterns in DNA. Meth Enzymol 1990; 183 : 211–21. 2. Sagot MF. Spelling approximate repeated or common motifs using a suffix tree. In : Lucchesi CL, Moura AV, eds. LATIN’98 : theoretical informatics lecture notes in computer science, vol. 1380. Berlin: Springer-Verlag, 1998 : 111–27. 3. Bailey TL, Elkan C. ParaMEME, a parallel implementation and a web interface for a DNA and protein motif discovery tool. Comput Appl BIOSci 1996; 12 : 303–10. 4. Reinert G, Schbath S, Waterman MS. Probabilistic and statistical properties of words: an overview. J Comput Biol 2000; 7 : 1–46. 5. d’Aubenton Carafa Y, Brody E, Thermes C. Prediction of rho-independent Escherichia coli transcription terminators. A statistical analysis of their RNA stem-loop structures. J Mol Biol 1990; 216 : 835–58. 6. Prestridge DS. Predicting Pol II promoter sequence using transcription factor binding sites. J Mol Biol 1995; 249 : 923–32. 7. Tolstrup N, Rouzé P, Brunak S. A branch point consensus from Arabidopsis found by non circular analysis allows for better prediction of acceptor sites. Nucleic Acids Res 1997; 25 : 3159–63. 8. Brendel V, Kleffe J, Carle Urioste JC, Walbot V. Prediction of splice sites in plant pre-mRNA from sequence properties. J Mol Biol 1998; 276 : 85–104. 9. Pedersen AG, Nielsen H. Neutral network prediction of translation initiation sites in eukaryotes: perspectives for EST and genome analysis. In : Gaasterland T, Karp P, Karplus K, Ouzounis C, Sander C, Valencia A, eds. The fifth international conference on intelligent systems for molecular biology. Halkidiki ,Greece: AAAI/MIT Press, 1997 : 226–33. 10. Borodovsky M, McIninch JD. GeneMark : parallel gene recognition for both DNA strands. Comp Chem 1993; 17 : 123–33. 11. Salzberg SL, Delcher AL, Kasif S, White O. Microbial gene identification using interpolated Markov models. Nucleic Acids Res 1998; 26 : 544–8. 12. Burge C, Karlin S. Prediction of complete gene structure in human genomic DNA. J Mol Biol 1998; 268 : 78–94. 13. Salzberg SL, Pertea M., Delcher AL, Gardner MJ, Tettelin H. Interpolated Markov models for eucaryotic gene finding. Genomic 1999; 59 : 24–31. 14. Snyder EE, Stormo GD. Identification of protein coding regions in genomic DNA. J Mol Biol 1998; 248 : 1–18. 15. Salzberg SL, Delcher AL, Fasman K, Henderson J. A decision tree system for finding genes in DNA. J Comput Biol 1998; 5 : 667–80. 16. Lukashin AV, Borodovsky M. GeneMark.hmm: new solutions for gene finding. Nucleic Acids Res 1998; 26 : 1107–15. 17. Krogh A. Two methods for improving performance of a HMM and their application for gene finding. In : Gaasterland T, Karp P, Karplus K, Ouzounis C, Sander C, Valencia A eds. The fifth international conference on intelligent systems for molecular biology. Halkidiki Greece: AAAI/MIT Press, 1997 : 179–86. 18. Florea L, Hartzell G, Zhang Z, Rubin GM, Miller W. A computer program for aligning a cDNA sequence with a genomic DNA sequence. Genome Res 1998; 8 : 967–74. 19. Jiang J, Jacob HJ. EbEST: an automated tool using expressed sequence tags to delineate gene structure. Genome Res 1998; 8 : 268–75. 20. Bairoch A, Apweiler R. The SWISS-PROT protein sequence database and its supplement TrEMBL in 2000. Nucleic Acids Res 2000; 28 : 45–8. 21. Bocs S, Danchin A, Médigue C. Re-annotation of genomes microbial CoDing Sequences : finding new genes and inaccurately annotated genes. BMC BioInformatics 2002 (sous presse). 22. Borodovsky M, McIninch J, Médigue C, Rudd K, Danchin A. Detection of new genes in the bacterial genome using Markov models for three gene classes. Nucleic Acids Res 1995; 17 : 3554–62. 23. Guédon Y. Computational methods for discrete hidden semi-Markov chains. Appl Stochastic Models Business Industry 1999; 15 : 195–224. 24. Bork P. Powers and pitfalls in sequence analysis: the 70% hurdle. Genome Res 2000; 10 : 398–400. 25. Pavy N, Rombauts S, Dehais P, et al. Evaluation of gene prediction software using a genomic data set : application to Arabidopsis thaliana sequences. Bioinformatics 1999; 15 : 887–99. 26. Lander ES, Linton LM, Birren B, et al. Initial sequencing and analysis of the human genome. Nature 2001; 409 : 860–921. 27. Altschul SF, Madden TL, Schäffer AA, et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res 1997; 25 : 3389–402. 28. Hofmann K, Bucher P, Falquet L, Bairoch A. The PROSITE database, its status in 1999. Nucleic Acids Res 1999; 27 : 215–9. 29. Bateman A, Birney E, Durbin R, Eddy SR, Howe KL, Sonnhammer ELL. The Pfam protein families database. Nucleic Acids Res 2000; 28 : 263–6. 30. Brazma A. On the importance of standardisation in life sciences. Bioinformatics 2001; 17 : 113–4. 31. Berman HM, Westbrook J, Feng Z, et al. The protein data bank. Nucleic Acids Res 2000; 28 : 235–42. 32. Nitschké P, Guerdoux-Jamet P, Chiapello H, et al. Indigo: a world-wide-web review on genomes and gene functions. FEMS Microbiol Rev 1998; 22 : 207–27. 33. Salgado H, Santos A, Garza-Ramos U, van Helden J, Diaz E, Collados-Vides J. RegulonDB (version 2.0): a database on transcriptional regulation in Escherichia coli. Nucleic Acids Res 1999; 27 : 59–60. 34. Wingender E, Chen X, Fricke E, et al. The TRANSFAC system on gene expression regulation. Nucleic Acids Res 2001; 29 : 281–3. 35. Tatusov RL, Galperin MY, Natale DA, Koonin EV. The COG database: a tool for genome-scale analysis of protein functions and evolution. Nucleic Acids Res 2000; 28 : 33–6. 36. Overbeek R, Larsen L, Maltsev N, Pusch GD, Selkov E. WIT: a system for metabolic reconstructions and comparative analysis of the genomes. In : Letovsky C, Kluwer S, eds. Mol Biol Databases 2002 (sous presse). 37. Riley M, Labedan B. Protein evolution viewed through Escherichia coli protein sequences: introducing the notion of structural segment of homology, the module. J Mol Biol 1997; 269 : 1–12. 38. Perrière G, Duret L, Gouy M. HOBACGEN: database system for comparative genomics in bacteria. Genome Res 2000; 10 : 379–85. 39. Overbeek R, Fonstein M, D’Souza M, Pusch GD, Maltsev N. The use of gene clusters to infer functional coupling. Proc Natl Acad Sci USA 1999; 96 : 2896–901. 40. Snel B, Lehmann G, Bork P, Huynen MA. STRING: a webserver to retrieve and display the repeatedly occuring neighbourhood of a gene. Nucleic Acids Res 2000; 28 : 3442–4. 41. Kanehisa M, Goto S. KEGG: Kyoto encyclopedia of genes and genomes. Nucleic Acids Res 2000; 28 : 29–34. 42. Karp PD. Integrated access to metabolic and genomic data. J Comp Biol 1996; 3 : 191–212. 43. Gaasterland T, Sensen CW. Fully automated genome analysis that reflects user needs and preferences. A detailled introduction to the MAGPIE system architecture. Biochimie 1996; 78 : 302–10. 44. Frishman D, Albermann K, Hani J, et al. Functional and structural genomics using PEDANT. Bioinformatics 2001; 17 : 44–57. 45. Andrade M, Brown N, Leroy C, et al. Automated genome sequence analysis and annotation. Bioinformatics 1999; 15 : 391–412. 46. Bailey LC, Fischer S, Schug J, Crabtree J, Gibson M, Overton GC. GAIA: framework annotation of genomic sequence. Genome Res 1998; 8 : 234–50. 47. Walker DR, Koonin EV. SEALS: a system for easy analysis of lots of sequences. In: Menlo Park A, ed. Proceedings of the international conference on intelligent systems for molecular biology. Halkidiki, Greece: AAAI/MIT Press, 1997 : 333–9. 48. Harris NL. Genotator: a workbench for sequence annotation. Genome Res 1997; 7 : 754–62. 49. Médigue C, Rechenmann F, Danchin A, Viari A. Imagene : an integrated computer environment for sequence annotation and analysis. Bioinformatics 1999; 15 : 2–15. 50. Rutherford J, Parkhill J, Crook T, et al. Artemis: sequence visualisation and annotation. Bioinformatics 2000; 16 : 944–5. 51. Galperin MY, Koonin EV. Sources of systematic error in functional annotation of genomes : domain rearrangement, non-orthologous gene displacement, and operon disruption. In Silico Biol 1998; 1 : 0007. 52. Fitch W. Distinguishing homologous from analogous protein. Syst Zool 1970; 19 : 99–113. 53. Fitch W. Homology a personal view on some of the problem. Trends Genet 2000; 16 : 2277–23. |



 Enfin, il convient de remarquer que les définitions précédentes sont de nature essentiellement phylogénétique et ne font aucune hypothèse quant à la similarité des séquences ou à la fonction des gènes concernés (même si de telles hypothèses doivent parfois être posées pour parvenir à reconstruire l’histoire évolutive d’un gène ou d’une famille de gènes). Ainsi, l’emploi du terme homologue pour indiquer que deux gènes présentent des séquences similaires doit être considéré comme une impropriété. De même, le fait que deux gènes orthologues devraient présenter la même fonction doit être, le plus souvent, considéré comme une hypothèse de travail.
Enfin, il convient de remarquer que les définitions précédentes sont de nature essentiellement phylogénétique et ne font aucune hypothèse quant à la similarité des séquences ou à la fonction des gènes concernés (même si de telles hypothèses doivent parfois être posées pour parvenir à reconstruire l’histoire évolutive d’un gène ou d’une famille de gènes). Ainsi, l’emploi du terme homologue pour indiquer que deux gènes présentent des séquences similaires doit être considéré comme une impropriété. De même, le fait que deux gènes orthologues devraient présenter la même fonction doit être, le plus souvent, considéré comme une hypothèse de travail.