| |
| Med Sci (Paris). 2002 February; 18(2): 169–180. Published online 2002 February 15. doi: 10.1051/medsci/2002182169.Cellules souches embryonnaires et clonage thérapeutique Alice Jouneau* and Jean-Paul Renard Unité de biologie du développement et biotechnologie, Inra, 78352 Jouy en Josas, France |
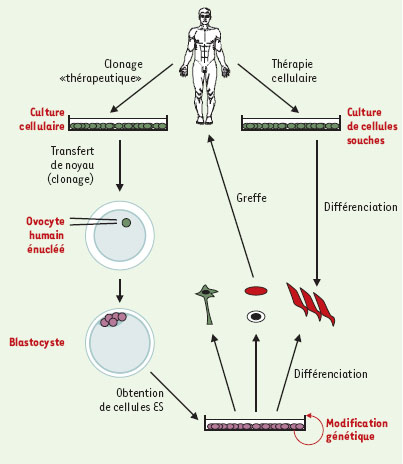
Depuis l’annonce très médiatisée de la naissance du mouton Dolly par la revue Nature en 1997 [1] (➜), le clonage désigne la technique qui consiste à introduire, dans le cytoplasme d’un ovocyte préalablement énucléé, un noyau d’une cellule issue d’un autre animal. Cette manipulation délicate permet de reconstituer un embryon qui, une fois transplanté dans une femelle receveuse, pourra se développer à terme. Le jeune ainsi obtenu possédera le même ensemble de gènes nucléaires que le donneur de cellules avec qui il formera, par définition, un clone. La synonymie, voulue par la revue Nature en 1997, entre la technique de transfert de noyau et le clonage apparaît néanmoins aujourd’hui largement abusive puisque cette technique peut, par exemple, être utilisée pour produire des organismes génétiquement différents par manipulation préalable des cellules donneuses. Mais le terme de clonage s’étant de fait imposé dans le public aux dépens de l’expres-sion « transfert de noyaux », nous l’utiliserons aussi dans cet article. (➜) m/s 1997 n°3, p. 426 et 428 L’importance de la question éthique soulevée par la perspective d’application de cette technique à l’homme a rapidement conduit à distinguer le clonage reproductif aboutissant à la naissance d’un jeune, du clonage non reproductif dit « clonage thérapeutique » se limitant à la production de cellules embryonnaires (Comité consultatif national d’éthique, 1997). Dans le premier cas, on laisse le noyau récapituler in vivo toutes les étapes du développement, apportant ainsi la preuve qu’il a pu retrouver l’état totipotent du noyau de l’œuf fécondé. Dans le deuxième cas, on interrompt très tôt cette reprogrammation des étapes du développement, avant l’implantation de l’embryon, alors que les cellules embryonnaires commencent à se différencier. Celles-ci possèdent alors un fort pouvoir de division et de différenciation et, en contrôlant en culture in vitro leur engagement dans une voie de différenciation donnée, on peut envisager de les utiliser à des fins de thérapie tissulaire (sous forme de greffons ou après leur injection par voie vasculaire) ou pour des recherches diagnostiques (Figure 1). Ce clonage dont la finalité est d’ordre thérapeutique pourrait constituer une approche complémentaire, voire alternative, à la thérapie dite somatique qui tire parti du fait que les cellules souches somatiques adultes peuvent aussi manifester des potentialités de différenciation dépassant largement celles de leur lignage d’origine [2, 3]. Mais l’éventail des potentiels de différenciation des cellules somatiques apparaît moins large que celui des cellules embryonnaires. Pour cette raison, et selon une terminologie issue de la langue anglaise, ces cellules sont dites multipotentes alors que les cellules embryonnaires sont qualifiées de pluripotentes. 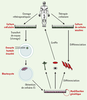 | Figure 1. Schéma comparant les différentes étapes qui seraient requises pour la mise en œuvre de procédures thérapeutiques soit à partir de lignées établies de cellules souches embryonnaires ou somatiques, soit après création de ces lignées par clonage. |
Le recours au clonage reproductif humain fait maintenant l’objet d’une désapprobation quasi unanime dans le monde. En revanche, le clonage thérapeutique suscite un grand intérêt, et de nombreux débats lui sont consacrés [4]. Pourtant l’histoire du clonage n’en est encore qu’à ses balbutiements. Les scientifiques se sont surtout attachés jusqu’à présent à en démontrer la faisabilité dans différentes espèces animales et à tenter d’en améliorer l’efficacité technique. Une nouvelle phase commence désormais pour essayer de comprendre la nature des mécanismes moléculaires et cellulaires mis en œuvre. Vu leur complexité, un long chemin reste à parcourir avant que le clonage thérapeutique puisse éventuellement devenir une réalité. C’est ce que nous voulons montrer dans cet article. Dans une première partie, nous décrirons certaines étapes clés au cours desquelles nous pensons que s’effectue la reprogrammation de l’activité du noyau. Puis nous ferons état des effets à long terme du clonage, en particulier les anomalies de développement observées chez l’animal. Enfin, nous montrerons comment ces effets, mal connus et moins encore maîtrisés, doivent être pris en compte dans la réflexion sur l’application à l’homme du clonage thérapeutique. |
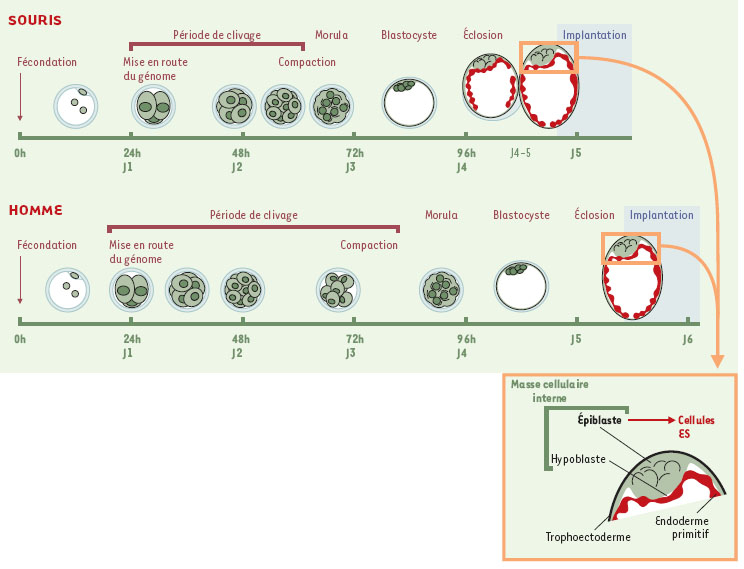
Les principales étapes de l’embryogenèse précoce Le début du développement de l’embryon est caractérisé par une période de division de l’œuf qui précède l’apparition des premières différenciations cellulaires. Cette période s’étend chez les mammifères sur plusieurs jours après la fécondation. Les deux premiers cycles de division sont lents (environ 24 h) et, contrairement à ce que l’on observe dans des espèces modèles comme le xénope, ils sont d’emblée étroitement réglés : les phases de réplication de l’ADN et de séparation des chromosomes à la mitose sont séparées par des phases G1 et G2, et la mise en place des points de contrôle (checkpoints) de chacune des phases résulte d’interactions moléculaires complexes entre les facteurs ovocytaires stockés au cours de l’ovogenèse. Après la première division chez la souris (stade 2 cellules), mais seulement après 2 à 4 divisions chez la plupart des autres mammifères (stade 8/16 cellules chez l’homme, 16/32 cellules chez le lapin), l’activité de synthèse du génome zygotique devient indispensable à la poursuite de l’embryogenèse. Cet événement marque la transition entre un contrôle maternel et un contrôle zygotique du développement (Figure 2). La première différenciation entre deux types cellulaires distincts, le trophoblaste et la « masse cellulaire interne », apparaît au stade blastocyste. Elle résulte de changements importants qui affectent l’organisation des cellules (à partir du stade 8 cellules chez la souris, au cycle de division suivant chez l’homme). Ces changements, qui caractérisent le stade morula, sont marqués par l’apparition d’une polarité cellulaire, par des remaniements des protéines de surface (qui favorisent l’adhérence intercellulaire), et par l’aplatissement des cellules situées à l’extérieur de l’embryon. Celles-ci établissent entre elles des relations fonctionnelles et forment un épithélium, le trophoblaste, qui, à l’intérieur de l’embryon, isole les cellules toujours indifférenciées de la masse cellulaire interne. À partir du 4e jour après la fécondation chez la souris, ou du 6e jour chez l’homme, soit, dans les deux cas juste avant l’implantation, deux lignages cellulaires se forment au sein de la masse cellulaire interne: les cellules au contact de la cavité blastocœlique (ou hypoblaste) se différencient en endoderme primitif qui vient tapisser la face interne du trophoblaste (que l’on nomme alors le tropho-ectoderme). Les autres cellules (ou épiblaste), au nombre de quelques dizaines seulement (20 à 40 chez la souris), sont encore indifférenciées. Ce sont elles qui, placées en culture dans des conditions qui favorisent leur multiplication tout en empêchant leur différenciation, peuvent donner les lignées de cellules embryonnaires souches (ES) pluripotentes. La gastrulation, qui correspond à la mise en place des trois feuillets de cellules à partir desquels se définira le plan du corps, puis l’organogenèse (l’ectoderme, le mésoderme et l’endoderme) débute plus tard, 6,5 jours après la fécondation chez la souris, et 11 à 13 jours chez l’homme. Ce stade de l’embryogenèse, que l’embryologiste Lewis Wolpert considérait comme le moment le plus important de la vie d’un individu, est marqué par l’apparition de la ligne primitive, dans la partie de l’épiblaste adjacente à la jonction entre la partie embryonnaire et la partie extraembryonnaire, région qui marque la future extrémité postérieure de l’embryon.  | Figure 2. Le début du développement embryonnaire et l’origine des cellules embryonnaires souches (cellules ES) chez la souris et chez l’homme. |
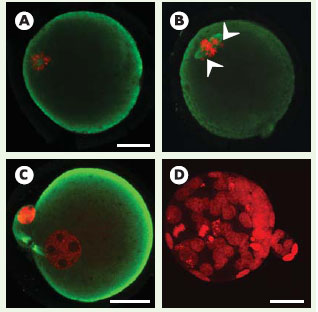
Ce résumé des premières étapes de l’embryogenèse montre que les cellules souches pluripotentes ES sont dérivées d’un tissu, l’épiblaste, que l’on peut isoler très tôt à partir du stade blastocyste, et bien avant l’apparition des migrations cellulaires à partir desquelles se définissent les axes définitifs du développement de l’organisme. La réorganisation d’un noyau somatique en un noyau embryonnaire doit donc prendre en compte les caractéristiques du tout début du développement précoce pour faire face à une double exigence: assurer le maintien de la constitution chromosomique du noyau donneur et permettre la mise en route des synthèses transcriptionelles requises pour la réalisation complète des modifications du stade morula, qui déterminent l’apparition des premières différenciations cellulaires au stade blastocyste. Reprogrammation et coordination des cycles cellulaires Le contrôle du premier cycle de division est largement dépendant des mécanismes associés à l’inactivation de l’activité cytostatique (CSF) qui stabilise le facteur MPF ( maturation promoting factor) pendant l’arrêt de l’ovocyte en métaphase de la deuxième division méiotique (MII) (➜ ). Chez la souris, cette dépendance se traduit par une réorganisation progressive de la chromatine zygotique étroitement associée à des changements d’activités enzymatiques (MAP-kinases notamment) de l’ovocyte [ 5]. Elle est aussi marquée par une plus grande durée de la métaphase qui suggère l’existence de mécanismes de contrôle spécifiques [ 6]. (➜) m/s 2001 n°10, p. 1046 Deux stratégies prenant en compte l’état du cytoplasme receveur ont été utilisées avec succès pour la reconstitution d’embryons. La première vise à mimer le déroulement complet de la séquence d’événements qui aboutissent, lors du développement normal (après fécondation), à la formation d’un pronoyau. Elle nécessite l’introduction du noyau étranger dans le cytoplasme d’un ovocyte énucléé, mais non encore activé: l’activité MPF élevée provoque une condensation rapide (prématurée) de la chromatine (PCC pour premature chromatine condensation). La chromatine des noyaux donneurs en phase G1 (équivalent 2n) ou G2 (équivalent 4n) du cycle mitotique formera alors des chromosomes allongés, respectivement à une et deux chromatides et un ou deux centromères. Dans le premier cas, il faudra empêcher l’extrusion de tout matériel génétique pour laisser le temps aux chromosomes de se décondenser et de répliquer leur ADN, avant de reformer une métaphase à la première division mitotique. En pratique, ce résultat peut être obtenu par exposition du cytoplasme à la cytochalasine, une molécule qui prévient la polymérisation des fibres d’actine. Dans le deuxième cas, la métaphase se formera normalement et la ségrégation des chromosomes se produira lors de l’expulsion du deuxième globule polaire (Figure 3). Les noyaux donneurs en phase S prendront en revanche une apparence « pulvérisée » typique, avec des régions plus ou moins condensées et des fragments dispersés, et présenteront de nombreux défauts de l’ADN. 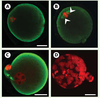 | Figure 3. Remodelage d’un noyau de cellule ES (R1) après injection dans un ovocyte de souris énucléé au stade métaphase II de la deuxième division de méiose et embryon au stade blastocyste obtenu in vitro 4 jours après l’injection. A. État du noyau 15 min après injection à l’état de métaphase; la cellule donneuse a été lysée dans la pipette d’injection juste avant le transfert, et seuls les chromosomes alignés sur la plaque métaphasique (4n chromatides) associée au fuseau mitotique (vue équatoriale) ont été injectés. B. État du noyau une heure après transfert, juste avant l’activation de l’ovocyte; la chromatine est encore en interphase et le fuseau a commencé une rotation qui va le placer perpendiculairement à la membrane plasmique; les fragments de chromatine dispersés (flèches), fréquemment observés à ce stade, pourraient être à l’origine des taux élevés d’arrêts précoces du développement. C. État du noyau 6 heures après activation: les deux lots de chromosomes mitotiques (équivalent chacun à 2n chromatides) viennent d’entrer en interphase et sont en fin d’isolement au sein de deux cellules de taille très différente (division asymétrique) encore reliées par un pont de microtubules; la cinétique des remaniements est très voisine de celle observée après fécondation in vitro. Fixation paraformaldéhyde 2,5 %; coloration de l’ADN à l’iodure de propidium en rouge; détection des microtubules en immunofluorescence indirecte en vert (anticorps primaire, anti-α tubuline de souris; anticorps secondaire, anti-Ig de souris couplé au FITC). D. Embryon au stade blastocyste : l’ADN du noyau (n≈55) a été coloré à l’iodure de propidium. Barres d ’échelle: A-C, 20 µm; D, 40 µm. |
La seconde stratégie consiste à introduire le noyau donneur dans un ovocyte déjà activé ou que l’on a laissé « vieillir » quelques heures in vitro de façon à introduire le noyau après (ou lors de) la chute d’activité du MPF. Cette approche vise à éviter les risques d’aneuploïdie associés à la condensation de la chromatine lors du PCC. Au moins théoriquement, les noyaux en phase G1, S ou G2 peuvent être utilisés indifféremment, puisque leur chromatine ne se condensera qu’à partir de la première mitose. En revanche, la durée d’exposition au cytoplasme ovocytaire est plus courte puisque celui-ci est déjà engagé dans la voie des cascades de régulations biochimiques qui aboutissent à la première division de mitose. Reprogrammation et remaniements chromatiniens Dès son introduction dans le cytoplasme de l’ovocyte, le noyau somatique va subir des modifications importantes de son organisation. Mise en présence d’un cytoplasme d’ovocyte non activé, son enveloppe nucléaire est rapidement désorganisée alors que l’exposition à un cytoplasme interphasique se traduit par un gonflement marqué de l’ensemble du noyau. Dans les deux situations, la chro-matine se trouve brutalement exposée aux facteurs de l’ovocyte et l’activité de synthèse du noyau est réprimée comme en atteste la disparition du nucléole. Cette répression n’est toutefois pas immédiate, et nous avons pu déceler, tant chez la souris que chez le bovin, le maintien d’une activité transcriptionnelle pendant les premières heures qui suivent l’introduction du noyau. L’activation transcriptionnelle du génome se produit au cours du développement normal dès le stade une cellule [7], mais ne devient nécessaire pour le développement qu’après une (stade 2 cellules) chez la souris, ou trois à quatre (stade 8/16 cellules) chez l’homme, divisions de l’œuf. Un état transitoire de répression transcriptionelle marqué par un remaniement profond des interactions ADN-protéines de la chromatine marque la fin du stade une cellule jusqu’à la deuxième réplication de l’ADN zygotique [8]. Il n’est pas certain que le noyau donneur doive récapituler toutes les étapes du développement embryonnaire précoce pour se trouver à même de conduire un développement complet et normal de l’embryon cloné. En effet, nous avons montré qu’après fécondation de l’œuf de souris, la structure répressive qui se met en place pour contrôler étroitement l’expression de certains gènes s’établit progressivement, tout au long de la période de clivage, et donc au-delà du stade deux cellules qui marque chez cette espèce le moment de l’activation majeure du génome zygotique [9]. Au cours du développement normal, la période de clivage paraît correspondre à une sorte de maturation de la chromatine qui précède l’apparition des premières différenciations au stade blastocyste (Figure 2). Elle pourrait offrir la fenêtre de temps suffisante à la chromatine somatique pour être réorganisée en chromatine embryonnaire. Deux observations faites au laboratoire à partir d’embryons fécondés in vivo sont en accord avec cette hypothèse. La première concerne les remaniements conformationnels des protéines de la chromatine et de leurs interactions avec l’ADN. Celles-ci impliquent une forte dépense d’énergie et des modifications covalentes des extrémités amino-terminales des histones, protéines basiques qui assurent une organisation fonctionnelle au nucléosome, élément de base de la chromatine. Des complexes multiprotéiques comme le complexe SWI/SNF, initialement identifiés chez la levure, sont de véritables machineries cellulaires utilisées pour régler cette architecture nucléaire. Des composantes de ce complexe, les protéines Brm (brahma) et Brg1, directement impliquées dans la régulation de la croissance des cellules [10], sont présentes dans presque toutes les cellules eucaryotes. Toutefois, nos travaux montrent qu’au cours du développement pré-implantatoire, la protéine Brg1 reste présente en grande quantité chez l’embryon de souris alors que la protéine brm, elle aussi d’origine maternelle, disparaît à partir du stade quatre cellules pour n’être à nouveau synthétisée qu’au stade blastocyste et apparemment d’abord dans les cellules du bouton embryonnaire [11]. Cette expression différentielle des deux éléments du complexe SWI/SNF, impliqués préférentiellement dans la création de sites d’hypersensibilité à la DNAse dans les régions promotrices de gènes transcrits au cours de l’anaphase [12], pourrait révéler une action spécifique s’exerçant au cours de la période de clivage. La seconde observation porte sur le profil de méthylation de l’ADN après son introduction dans le cytoplasme ovocytaire. La méthylation de l’ADN est associée à la formation d’une structure chromatinienne compacte et inactive [13]. Les noyaux des cellules somatiques ont un niveau de méthylation élevé mais une activité méthyltransférase faible, alors que les noyaux des cellules embryonnaires ont un niveau de méthylation faible et subissent une méthylation active après la formation du blastocyste [14]. En utilisant un anticorps dirigé contre les résidus 5–méthylcytosine de l’ADN, nous avons observé dans un travail réalisé en collaboration avec le groupe d’Evani Péquignot (Inserm U383, Hôpital Necker, Paris, France) avec des embryons bovins clonés,que les noyaux somatiques conservent un niveau de méthylation élevé pendant plusieurs divisions de clivage mais sont déméthylés brutalement à partir du stade morula, et non progressivement à chaque clivage comme au cours du développement normal [15]. Cette différence dans le profil de méthylation au cours de la période de clivage de l’embryon pourrait expliquer les profils anormaux de méthylation de régions spécifiques du génome observés à la naissance chez des veaux clonés [16]. Mais elle n’est pas incompatible avec l’obtention d’animaux physiologiquement normaux et fertiles comme cela est maintenant bien établi dans cette espèce. |
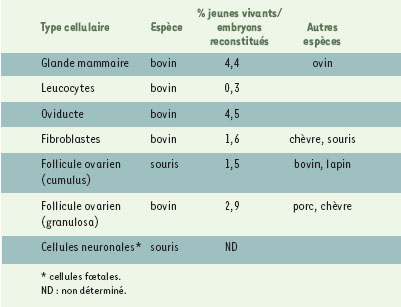
Les cellules donneuses de noyaux et l’efficacité du clonage Le Tableau I présente des résultats de clonage obtenus avec différents types de cellules somatiques adultes. L’observation la plus évidente est celle de la médiocre efficacité de la technique, toutes espèces confondues, et quel que soit le type de cellule donneuse. Le faible taux de naissance peut conduire à faire l’hypothèse d’une hétérogénéité de la population de cellules utilisée comme source de noyaux. Or, nous avons observé plusieurs fois au laboratoire des différences importantes entre les taux de développement de groupes d’embryons obtenus à partir des noyaux de plusieurs sous-cultures de fibroblastes issus d’un même explant tissulaire. Ces différences qui peuvent aller de 1 à 5 se manifestent tant en terme de taux de blastocystes que de taux de gestations après transfert [17]. On peut donc en déduire que la mise en culture, en parallèle, de plusieurs lots du même explant a accentué une hétérogénéité initiale de la population cellulaire. Cette hétérogénéité peut concerner différents aspects de la biologie des cellules que nous tentons maintenant de caractériser pour définir de proche en proche les conditions de leur maintien in vitro qui seraient compatibles avec des taux de développement à terme plus réguliers.  | Tableau I. Types de cellules somatiques adultes utilisées à ce jour avec succès comme source de noyaux pour produire des animaux physiologiquement normaux et fertiles, et exemples de jeunes vivants obtenus. |
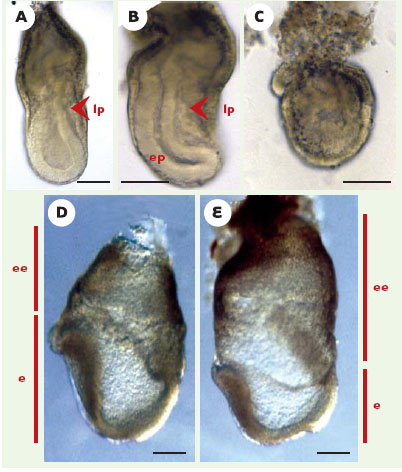
Un premier aspect concerne l’état de différenciation de cellules provenant d’un tissu en renouvellement constant comme c’est le cas des explants de peau auxquels nous avons largement recours. On ne peut exclure l’hypothèse selon laquelle seules de rares cellules indifférenciées, présentes dans la culture, permettraient un développement à terme. En effet, l’efficacité du clonage décroît fortement quand on passe de l’utilisation de cellules embryonnaires (blastomères) à celle de cellules fœtales puis adultes. Nous avons pu le montrer clairement chez les bovins en comparant l’évolution des taux de gestation obtenus à partir de ces trois types de cellules donneuses utilisées simultanément avec les mêmes lots d’ovocytes receveurs [18]. Toutefois, les taux de gestation obtenus avec les cellules adultes ne dépendent que très peu de la durée de chacune des cultures et plusieurs veaux, maintenant adultes, ont pu être produits après plus de quinze passages, ce qui correspond environ à 3 mois de culture. Le maintien de cellules à l’état indifférencié paraît dans ces conditions très peu probable. Un deuxième aspect concerne le cycle cellulaire des cellules donneuses. Dès les premiers succès du clonage, il y a cinq ans, l’accent a été mis sur l’importance du stade du cycle cellulaire de la cellule donneuse, par rapport à celui du cytoplasme de la cellule receveuse -l’ovocyte - et les conséquences selon les différents cas de figure ont été étudiées [19]. Des cellules ont été choisies en G0, G1 ou G2. Le problème est que l’identification certaine et reproductible des cellules dans l’une de ces phases est le plus souvent une gageure. De plus, ces phases sont assez étendues et recouvrent de fait plusieurs états de la chromatine. Dans notre laboratoire, nous avons cherché à réduire cette incertitude en prenant des cellules en métaphase, état plus ponctuel, et surtout bien identifiable par l’observation au microscope. Nous avons comparé le taux de développement in vitro jusqu’au stade blastocyste d’embryons clonés à partir de noyaux en métaphase ou interphase. Des mesures préalables de cytométrie ayant montré que la taille des cellules et la quantité d’ADN étaient corrélées, nous avons utilisé ce critère de taille pour enrichir les populations de cellules donneuses en noyaux en GI ou G2. Le Tableau II montre clairement que les noyaux de cellules ES en métaphase donnent des taux de développement in vitro meilleurs que des noyaux présumés en G1 ou G2 [20]. Les blastocystes issus du transfert de noyaux de cellules ES en métaphase ont le même nombre total de cellules et le même taux de répartition entre cellules du bouton embryonnaire et cellules trophoblastiques que les blastocystes issus de fécondation in vitro. Les deux types d’embryons ont une cinétique de développement voisine de celle observée après fécondation in vitro. Si on laisse les blastocystes clonés en culture, ils forment des corps embryoïdes mais si on les transplante dans l’utérus d’une femelle receveuse, leur taux de développement in vivo reste faible. Ces données montrent néanmoins que des blastocystes clonés peuvent aisément être produits pour servir de source de cellules embryonnaires (Figure 4). 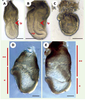 | Figure 4. Morphologie des embryons clonés de souris au jour 7,5 (A-C) et 8 (D, E) de gestation. A. L’embryon est d’apparence normale. B. Certains embryons clonés présentent des anomalies de gastrulation: les cellules accumulées dans la ligne primitive (lp) ont repoussé l’épiblaste (ep). D’autres embryons ne gastrulent pas, et restent très petits. C, D, E. Au jour 8, la plupart des embryons issus de clonage sont retardés. E. Certains présentent une partie embryonnaire (e) trop petite par rapport à l’extra-embryonnaire (ee). Barres d’échelle: (A-C), 100 µm; (D, E), 200 µm. |
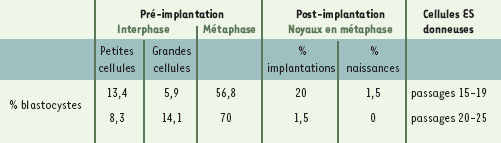
Un troisième aspect de l’étude du développement précoce des embryons clonés qui reçoit une attention croissante concerne l’état épigénétique du noyau donneur au moment de son utilisation. C’est une source d’hétérogénéité pour le clonage, mais elle est plus difficile à définir et plus encore à maîtriser. L’idée est que certaines cellules seraient dans un état plus propice à la reprogrammation que d’autres parce que leur chromatine serait dans une configuration qui se prêterait mieux au remodelage qu’elle subit quand elle est confrontée à l’environnement ovocytaire. À cela s’ajoute le problème inhérent à la culture cellulaire : comment contrôler l’apparition de modifications génétiques (mutations) ou épigénétiques (par exemple les changements de méthylation) au cours des passages en culture? Nous-mêmes avons constaté les effets d’une culture prolongée des cellules ES utilisées comme source de noyaux. En principe, les cellules ES sont pluripotentes, et l’on pourrait théoriquement attendre un taux de réussite proche de celui obtenu avec des cellules embryonnaires. Néanmoins, ce taux reste très faible, à peine plus élevé qu’avec des cellules somatiques différenciées (voir Tableau II) [21]. La mise en culture de ces cellules serait cause de leur moindre capacité à se reprogrammer. Après un certain nombre de passages, pour la même lignée de cellules ES, nous avons observé une importante chute de l’implantation [20]. D’autres travaux récents ont également montré que l’état épigénétique des cellules ES était particulièrement instable. Des sous-clones cellulaires issus de la même lignée de cellules ES présentent rapidement des différences importantes d’expression de gènes soumis à l’empreinte, reflétant l’apparition rapide de modifications épigénétiques pendant la culture [22]. Le choix d’une « bonne » cellule donneuse pour le transfert de noyaux devra tenir compte de ce phénomène, d’autant plus que les conséquences de ces mutations épigénétiques sur le potentiel de développement des embryons clonés ne sont pas encore appréhendées.  | Tableau II. Développement d’embryons murins après transfert de noyaux de cellules ES à différentes phases du cycle cellulaire. |
|
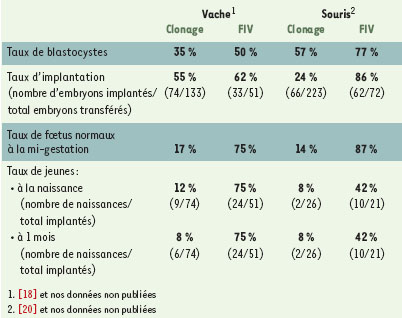
Mortalité fœtale et post-natale Le transfert d’embryons issus de noyaux somatiques se traduit fréquemment par l’apparition d’anomalies tardives de développement. C’est ce qui ressort du Tableau III qui compare les développements après transfert de noyaux somatiques adultes (vache) ou de noyaux de cellules ES (souris) obtenus dans notre laboratoire. Pour les deux espèces, des pertes importantes sont enregistrées jusqu’à la mi-gestation. Elles s’accompagnent d’anomalies du développement placentaire mises en évidence de façon récurrente chez la vache [18, 23] et chez la souris où les placentas à la naissance sont très fréquemment plus développés que la normale. Chez les bovins, où la durée de la gestation est de neuf mois, le suivi échographique des femelles porteuses révèle en outre que plus de 40 % des fœtus d’apparence morphologique et de croissance normales à la mi- gestation, ne se développeront pas à terme alors que dans les conditions naturelles ou après transfert d’embryons fécondés in vitro, ce taux d’échec est inférieur à 5 %. Après la naissance, les deux tiers seulement des veaux nés survivent. Chez la souris, plus de la moitié des embryons clonés s’implantent, mais seulement 10 % d’entre eux survivront au-delà de la mi-gestation; une telle mortalité n’est pas observée avec des embryons issus de fécondation in vitro. Les conséquences d’une reprogrammation incomplète, ou entachée d’erreurs, sont donc bien à l’origine des avortements tardifs observés. En comparant l’ADN de souriceaux nouveau-nés issus d’un transfert nucléaire, il a été observé des anomalies de méthylation dans certains îlots CpG, ainsi que des différences de niveau d’expression de gènes soumis à l’empreinte [22, 24]. De ce point de vue, les clones ne sont pas des copies conformes, ni entre eux, ni avec les cellules dont ils sont issus.  | Tableau III. Développement in vivo des embryons clonés de vache et de souris ; comparaison avec les embryons issus de fécondation in vitro (FIV). |
Ces constatations ont conduit R. Jaenish [25] à affirmer que les clones portaient tous des anomalies épigénétiques. La qualification de normal ou d’anormal nous paraît toutefois être ici mal définie, dans la mesure où la variabilité permise, c’est-à-dire sans conséquences phénotypiques, des profils de méthylation au sein d’une population n’est pas connue. La question qui se pose est donc plutôt celle de savoir jusqu’à quel point les animaux issus du transfert de noyaux somatiques de même génotype différent entre eux. Vu sous cet angle, il serait intéressant de comparer le profil de méthylation de l’ADN d’animaux issus de fécondation naturelle, de fécondation in vitro, ou d’ICSI (injection intracytoplasmique de spermatozoïdes). On sait en effet que la culture des embryons, même de courte durée, peut affecter l’expression de gènes soumis à empreinte [26, 27]. Cela n’empêche pas un certain nombre d’animaux clonés de paraître tout à fait normaux, du moins selon les critères utilisés pour en juger. Nous suivons depuis maintenant quatre ans les vaches clonées dans notre laboratoire et depuis trois ans, nos premières souris. Ces animaux ne manifestent aucune différence marquante avec des animaux contemporains issus d’une reproduction sexuée. Nous allons tenter d’enrichir la batterie de mesures à réaliser en routine en vue de déceler d’éventuelles différences physiologiques y compris comportementales. Ces considérations ne cherchent en aucun cas à minimiser le rôle de mutations épigénétiques dans la mortalité très élevée des clones au cours du développement embryonnaire. Le défi actuel est de pouvoir relier ces anomalies, qui peuvent affecter la méthylation mais aussi la structure chromatinienne, à la dérégulation précise de gènes essentiels pour le développement. L’approche que nous avons mise en place consiste à étudier le développement des clones dans deux espèces, vache et souris, avec l’objectif de mettre en évidence des gènes dont l’expression serait altérée précocement dans les embryons et les fœtus clonés. Chez la souris, une forte proportion d’anomalies est décelable plus tôt, dès la gastrulation: par une analyse moléculaire fine, couplée à l’utilisation des outils de la génomique expressionnelle*, nous espérons mieux comprendre chez cette espèce les causes de cette forte mortalité. Cette recherche nous paraît importante non seulement pour mieux caractériser les régulations les plus sensibles à la reprogrammation de l’activité d’un noyau somatique, mais aussi pour fournir une base scientifique à l’important débat éthique sur le recours ou non au clonage thérapeutique. Car la question préalable, sur le plan scientifique, que pose l’utilisation des blastocystes clonés comme source de cellules souches embryonnaires est bien celle de savoir si les anomalies des fœtus clonés sont dues avant tout à un défaut de développement des tissus extra-embryonnaires (placenta, sac vitellin) ou bien avant tout à l’embryon lui-même d’où sont dérivées les cellules souches. |
Le clonage thérapeutique en question Trois équipes ont montré qu’on pouvait dériver des cellules ES apparemment normales de blastocystes murins clonés [28–30]. Les cellules donneuses étaient dans tous les cas des cellules somatiques adultes (cumulus, fibroblastes et cellules neurales). Le taux d’obtention de ces cellules ES (nombre de lignées engendrées par rapport au nombre de blastocystes explantés) est normal. Elles se différencient en nombreux lignages cellulaires, y compris des neurones, participent à la formation d’embryons chimères, et colonisent la lignée germinale. Ces critères sont ceux habituellement requis pour démontrer le réel statut « embryonnaire souche » d’une lignée cellulaire. Ce qui est très intrigant dans ces expériences est que, dans le premier cas rapporté, aucune naissance à partir des mêmes blastocystes n’avait été obtenue par le laboratoire. En d’autres termes, les embryons à partir desquels avaient été isolés les cellules ES étaient compromis dans leur développement. Ces résultats suscitent un certain nombre de questions: l’épiblaste, tissu à partir duquel sont dérivées les cellules ES, est-il ou non complètement normal? Sinon pourquoi obtient-on si « facilement » des cellules ES (Figure 2) ayant des propriétés apparemment normales? Si l’épiblaste était normal dans les embryons clonés, cela signifierait que leurs anomalies de développement proviendraient des tissus extra-embryonnaires, endoderme primitif et/ou trophoectoderme. Des interactions anormales entre tissus extra-embryonnaires et embryonnaires peuvent entraîner des perturbations dans l’émission de signaux de croissance, de différenciation, de migration cellulaire, et donc perturber la construction tridimensionnelle de l’embryon. Le trophectoderme est impliqué dans l’attachement du blastocyste à la membrane utérine et dans sa nidation. Il a également un rôle trophique, en participant à la formation du sac vitellin pariétal et du placenta. L’endoderme primitif se différencie en deux lignages cellulaires, l’endoderme pariétal et l’endoderme viscéral. Le premier a pour fonction principale de sécréter la membrane de Reichert, barrière entre l’embryon et le tissu maternel. Le second a une fonction trophique pour l’épiblaste; diverses études montrent qu’il a également un rôle essentiel dans la spécification antérieure de l’embryon [31]. L’absence de certaines protéines telles que vHNF1, HNF-4, ou Hex, qui sont exprimées dans l’endoderme viscéral, provoque un défaut de croissance de l’épiblaste, une gastrulation anormale voire inexistante, une réduction du cerveau antérieur [32–34] (➜). (➜) m/s 2000 n°2, p. 192 L’hypothèse d’un épiblaste normal serait évidemment très favorable au clonage thérapeutique, puisque les anomalies observées lors de la différenciation in vivo de l’épiblaste ne seraient pas intrinsèques aux cellules de l’épiblaste. Il serait néanmoins très surprenant que les défauts de reprogrammation n’affectent pas toute une partie de l’œuf. Une explication possible pourrait être la lenteur du procesus de reprogrammation, qui s’étendrait au-delà du stade pré-implantatoire, et se ferait à des vitesses différentes selon les lignages cellulaires. Elle se terminerait plus tardivement dans les tissus extra-embryonnaires, ce décalage étant létal pour l’embryon. On sait par exemple que, dans l’embryon fécondé, des modifications génétiques interviennent jusqu’à la gastrulation: la déméthylation massive qui intervient pendant le stade pré-implantatoire est suivie, avant la gastrulation, d’une reméthylation progressive qui diffère selon les tissus [35]. De même, l’inactivation du chromosome X dans les embryons femelles, est contrôlée par un processus différent dans l’embryon proprement dit et dans le lignage du trophoectoderme [36, 37]. La seconde alternative est que l’épiblaste est luimême défectueux. Dans ce cas, toute anomalie n’entraînera pas nécessairement une réduction du potentiel de différenciation des cellules ES. En effet, la genèse d’un embryon nécessite la construction d’une architecture tridimensionnelle, dont les composantes se mettent en place suivant une chronologie et une géométrie précises. Une erreur de position ou un décalage temporel affectant un petit groupe de cellules pourrait par exemple désorganiser un gradient morphogénétique et modifier l’ordre d’entrée des cellules dans la ligne primitive. Au début de la gastrulation, cet ordre détermine leur future position le long de l’axe dorso-ventral de l’embryon [38]. L’inactivation du récepteur transmembranaire ActRIB, essentielle pour la formation de la ligne primitive, entraîne l’arrêt du développement avant la gastrulation. L’expression de ce gène n’est cependant pas essentielle pour la formation du mésoderme; en effet, les cellules ES n’exprimant plus ActRIB restent capables de se différencier en dérivés mésodermiques [39]. En outre, les cellules de l’environnement peuvent compenser au niveau local l’absence de synthèse de facteurs diffusibles dans une cellule mutante. C’est le cas du facteur de croissance Nodal, de la famille des TGFβ (transforming growth factor β), exprimé dans l’endoderme viscéral mais aussi dans l’épiblaste. L’inactivation de ce gène inhibe la gastrulation et, plus tardivement, la formation des structures antérieures [40]. Les cellules ES issues d’embryons nodal−/− sont toutefois capables de se différencier de façon normale [41]. Quoi qu’il en soit, l’hypothèse d’un épiblaste défectueux reste la moins favorable au clonage thérapeutique. Si elle s’avérait exacte, il serait nécessaire de vérifier soigneusement l’éventail de différenciation possible des cellules ES issues d’embryons clonés, d’autant plus qu’il n’y a pas chez l’homme les mêmes moyens de mettre à l’épreuve la « normalité » de ces cellules que chez la souris. La possibilité de différencier in vitro des cellules ES n’est pas un gage suffisant de leur comportement une fois greffées dans un tissu adulte. L’analyse de chimères d’agrégation entre des embryons clonés et des embryons normaux devrait permettre de répondre à certaines des questions posées. Ces techniques permettent d’associer l’épiblaste d’un type d’embryon aux tissus extra-embryonnaires d’un autre, afin de mieux comprendre la contribution de chaque tissu au phénotype observé. Les nombreuses questions présentées ci-dessus sont à l’heure actuelle sans réponse et la poursuite d’un travail expérimental approfondi avec plusieurs systèmes animaux modèles apparaît plus que jamais nécessaire. |
Le recours au clonage pour la production de cellules embryonnaires à des fins thérapeutiques chez l’homme prend en compte l’aptitude qu’ont les cellules souches embryonnaires de pouvoir être différenciées aisément in vitro en un grand nombre de types cellulaires. Mais ce clonage thérapeutique nous place dans le même temps devant une situation redoutable: celle d’avoir à décider a priori de créer des embryons humains dont le statut ontologique, dès les tous premiers stades du développement, est au cœur du débat éthique. L’emploi des seules cellules souches adultes permettrait, au moins en partie, de faire l’économie de ce débat. Les connaissances actuellement acquises sont toutefois loin de pouvoir fonder un choix exclusif en leur faveur: la voie du clonage à finalités thérapeutiques nous paraît donc ne pas devoir être d’emblée écartée. Avant d’être en mesure de pouvoir définir, à partir de données scientifiques, la place éventuelle que cette technique pourrait occuper dans l’ensemble des stratégies de recherche actuellement engagées sur les cellules souches humaines, il faut mieux connaître les potentialités effectives du clonage animal, ne serait-ce que pour mieux cerner, puis tenter de lever les incertitudes qui pèsent sur son innocuité. Les premiers essais avec des cellules et des ovocytes humains annoncés récemmment par une firme privée [42] nous paraissent plus relever d’une impatience technologique et d’un désir médiatique que d’une démarche solidement fondée. La prise en compte de l’état des connaissances scientifiques est une phase importante dans les débats éthiques suscités par le développement des connaissances en biologie. Les nombreuses questions sur le développement des embryons clonés, présentées ci-dessus, sont à l’heure actuelle sans réponse chez l’animal. Si des progrès notables devaient être obtenus alors, mais alors seulement, il faudrait explorer chez l’homme les conditions qui pourraient permettre de décider, en toute connaissance de cause, de la place que le clonage à finalités thérapeutiques pourrait occuper pour engendrer des lignées de cellules embryonnaires humaines. Et apporter une réponse aux attentes que l’on place aujourd’hui, sans doute à juste titre, en elles. |
1. Wilmut I, Schnieke AE, Mc Whir J, Kind AJ, Campbell KHS. Viable offspring derived from fetal and adult mammalian cells. Nature 1997; 385 : 810–3. 2. Weissman IL. Stem cells : units of development, units of regeneration, and units in evolution. Cell 2000; 100 : 157–68. 3. Fuchs E, Segre JA. Stem cells: a new lease on life Cell 2000; 100 : 143–55. 4. Claeys A, Huriet C. Le clonage, la thérapie cellulaire et l’utilisation thérapeutique des cellules embryonnaires. Rapport de l’Office Parlementaire d’Évaluation des Choix Scientifiques et Technologiques, 2000; 2198 : 181 p. 5. Verlhac MH, Kubiak JZ, Clarke HJ, Maro B. Microtubule and chromatin behavior follow MAP kinase activity but not MPF activity during meiosis in mouse oocytes. Development 1994; 120 : 1017–25. 6. Ciemerych MA, Maro B, Kubiak JZ. Control of duration of the first two mitoses in a mouse embryo. Zygote 1999; 7 : 293–300. 7. Aoki F, Worrad DM, Schultz RM. Regulation of transcriptional activity during the first and second cell cycles in the preimplantation mouse embryo. Dev Biol 1997; 181 : 296–307. 8. Schultz RM, Davis W Jr, Stein P, Svoboda P. Reprogramming of gene expression during preimplantation development. J Exp Zool 1999; 285 : 276–82. 9. Thompson EM, Legouy E, Renard JP. Mouse embryos do not wait for the MBT: chromatin and RNA polymerasere modeling in genome activation at the onset of development. Dev Genet 1998; 22h31–42. 10. Mutchardt C, Yaniv M. ATP-dependent chromatin remodelling: SWI/SNF and Co are on the job. J Mol Biol 1999; 293 : 187–98. 11. Legouy E, Thompson EM, Muchardt C, Renard JP. Differential preimplantation regulation of two mouse homologues of the yeast SWI2 protein. Dev Dyn 1998; 212 : 38–48. 12. Krebs JE, Fry CJ, Samuels ML, Peterson CL Global role for chromatin remodeling enzymes in mitotic gene expression. Cell 2000; 102 : 587–98. 13. Bird AP, Wolffe AP. Methylation-induced repression-belts, braces, and chromatin. Cell 1999, 99 : 451–4. 14. Reik W, Dean W, Walter J. Epigenetic reprogramming in mammalian development. Science 2001; 293 : 1089–93. 15. Bourc’his D, Le Bourhis D, Patin D, et al. Delayed and incomplete reprogramming of chromosome methylation patterns in bovine cloned embryons. Curr Biol 2001; 11 : 1542–6. 16. Kang YK, Koo DB, Park JS, et al. Aberrant methylation of donor genome in cloned bovine embryos. Nat Genet 2001; 28 : 173–7. 17. Renard JP, Zhou Qi, LeBourhis D, et al. Nuclear transfer technologies: between successes and doubts. Theriogenology 2002; 57 : 203–22. 18. Heyman Y, Chavatte-Palmer P, LeBourhis D, Camous S, Vignon X, Renard JP. Frequency and occurrence of late gestation losses from cattle cloned embryos. Biol Reprod 2002; 66 : 6–13. 19. Campbell KHS, Loi P, Otaegui PJ, Wilmut I. Cell cycle coordination in embryo cloning by nuclear transfer. Rev Reprod 1996; 1 : 40–6. 20. Zhou Q, Jouneau A, Brochard V, Adenot P, Renard JP. Developmental potential of mouse embryos reconstructed from metaphase embryonic stem cell nuclei. Biol Reprod 2001; 65 : 412–9. 21. Wakayama T, Perry AC, Zuccotti M, Johnson KR, Yanagimachi R. Full-term development of mice from enucleated oocytes injected with cumulus cell nuclei. Nature 1998; 394 : 369–74. 22. Humpherys D, Eggan K, Akutsu H, et al. Epigenetic instability in ES cells and cloned mice. Science 2001; 293 : 95–7. 23. Hill JR, Burghardt RC, Jones K, et al. Evidence for placental abnormality as the major cause of mortality in first trimester somatic cell bovine fetuses. Biol Reprod 2000; 63 : 1787–94. 24. Ohgane J, Wakayama T, Kogo Y, et al. DNA methylation variation in cloned mice. Genesis 2001; 30 : 45–50. 25. Eggan K, Akutsu H, Loring J, et al. Hybrid vigor, fetal overgrowth, and viability of mice derived by nuclear cloning and tetraploid embryo complementation. Proc Natl Acad Sci USA 2001; 98 : 6209–14. 26. Doherty AS, Mann MRW, Tremblay KD, Bartolomei MS, Schultz RM. Differential effects of culture on imprinted H19 expression in the preimplantation mouse embryo. Biol Reprod 2000; 62 : 1526–35. 27. Khosla S, Dean W, Brown D, Reik W, Feil R. Culture of preimplantation mouse embryos affects fetal development and the expression of imprinted genes. Biol Reprod 2001; 64 : 918–26. 28. Munsie MJ, Michalska AE, O’Brien CM, Trounson AO, Pera MF, Mountford PS. Isolation of pluripotent embryonic stem cells from reprogrammed adult mouse somatic cell nuclei. Curr Biol 2000; 10 : 989–92. 29. Kawase E, Yamazaki Y, Yagi T, Yanagimachi R, Pedersen RA. Mouse embryonic stem (ES) cell lines established from neuronal cell-derived cloned blastocyts. Genesis 2000; 28 : 156–63. 30. Wakayama T, Tabar V, Rodriguez I, Perry AC, Studer L, Mombaerts P. Differentiation of embryonic stem cell lines generated from adult somatic cells by nuclear transfer. Science 2001; 292 : 740–3. 31. Beddington RS, Robertson EJ. Anterior patterning in mouse. Trends Genet 1998; 14 : 277–84. 32. Chen WS, Manova K, Weinstein DC, et al. Disruption of the HNF-4 gene, expressed in visceral endoderm, leads to cell death in embryonic ectoderm and impaired gastrulation of mouse embryos. Genes Dev 1994; 8 : 2466–77. 33. Barbacci E, Michaël R, Ott MO, Breillat C, Huetz F, Cereghini S. Variant hepatocyte nuclear factor 1 is required for visceral endoderm specification. Development 1999; 126 : 4795–805. 34. Martinez Barbera JP, Clements M, Thomas P, et al. The homeobox gene Hex is required in definitive endodermal tissues for normal forebrain, liver and thyroid formation. Development 2000; 127 : 2433–45. 35. Jaenish R. DNA methylation and imprinting: why bother? Trends Genet 1997; 13 : 323–9. 36. Epstein CJ, Smith S, Travis B, Tucker G. Both X chromosomes function before visible X-chromosome inactivation in female mouse embryos. Nature 1978; 274 : 500–3. 37. Monk M, Harper MI. Sequential X chromosome inactivation coupled with cellular differentiation in early mouse embryos. Nature 1979; 281 : 311–3. 38. Tam PPL, Gad JM, Kinder SJ, Tsang TE, Behringer RR. Morphogenetic tissue movement and the establishment of body plan during development from blastocyst to gastrula in the mouse. Bioessays 2001; 23 : 508–17. 39. Gu Z, Nomura M, Simpson BB, et al. The type I activin receptor ActRIB is required for egg cylinder organization and gastrulation in the mouse. Genes Dev 1998; 12 : 844–57. 40. Zhou X, Sasaki H, Lowe L, Hogan BL, Kuehn MR. Nodal is a novel TGF-beta-like gene expressed in the mouse node during gastrulation. Nature 1993; 361 : 543–7. 41. Conlon FL, Lyons KM, Takaesu N, et al. A primary requirement for nodal in the formation and maintenance of the primitive streak in the mouse. Development 1994; 120 : 1919–28. 42. Cibelli, JB, Kiessling AA, Cunniff K, Richards C, Lanza R, West M. Somatic cell nuclear transfer in humans: pronuclear and early embryonic development. J Regen Med 2001; 2 : 25–31. |