| |
| Med Sci (Paris). 2003 April; 19(4): 477–480. Published online 2003 April 15. doi: 10.1051/medsci/2003194477.Les méthyltransférases de l’ADN : du remodelage de la chromatine au cancer François Fuks* Laboratoire de Virologie Moléculaire, CP 614, Faculté de Médecine, Université Libre de Bruxelles, 808, route de Lennik, B-1070 Bruxelles, Belgique |
Comprendre comment les gènes sont exprimés dans un type cellulaire particulier reste l’une des questions fondamentales de la biologie. En effet, chaque cellule du corps humain contient initialement le même nombre de gènes, pourtant seul un petit nombre d’entre eux est exprimé dans une cellule, selon le stade de développement de l’organisme ou le type cellulaire considéré. Pour appréhender les bases moléculaires de la régulation de l’expression des gènes, il est essentiel de comprendre à la fois comment ceux-ci sont activés et réprimés. Parmi les mécanismes de verrouillage de l’expression des gènes, la méthylation des cytosines, modification chimique spécifique de l’ADN, apparaît de plus en plus comme un niveau de contrôle important. Cette modification est dite « épigénétique » parce qu’elle module l’activité du génome sans en affecter directement la séquence. Elle est localisée dans des régions génomiques spécifiques au sein de dinucléotides CpG. Bien qu’identifiée dès 1948, la méthylation a été perçue pendant de longues années par la communauté scientifique comme un épiphénomène biologique. Cependant, les progrès considérables réalisés au cours des dernières années ont permis au contraire de démontrer le rôle essentiel joué par la méthylation au cours de plusieurs processus biologiques associés au développement, comme l’inactivation du chromosome X chez les mammifères femelles, l’imprinting génomique ou encore l’expression de gènes spécifiques de tissus [
1]. En outre, il semble de plus en plus clair qu’une méthylation aberrante de l’ADN joue un rôle primordial dans le processus de cancérogenèse. En particulier, une méthylation altérée peut conduire à l’inactivation de gènes suppresseurs de tumeurs et à un processus de tumorigenèse comparable à celui induit par les mutations génétiques [
2]. L’importance de la méthylation de l’ADN dans ces différents phénomènes biologiques est probablement à rapprocher de son rôle dans la répression de la transcription. La question actuelle n’est donc plus de savoir si la méthylation est un processus biologique important, mais bien de cerner comment elle participe au développement et à la cancérogenèse. Plus particulièrement, il est fondamental de comprendre comment la méthylation verrouille l’expression génique. À cet égard, des progrès marquants ont récemment été obtenus grâce à l’identification des enzymes qui catalysent la réaction de transfert de résidus méthyls sur les cytosines: les méthyltransférases de l’ADN (Dnmt). Trois enzymes ont été clonées: Dnmt1, Dnmt3a et Dnmt3b. Plusieurs observations indiquent qu’elles interviennent dans les processus de développement et de tumorigenèse. En particulier, les souris dont l’un des gènes dnmt a été invalidé ont un développement anormal et meurent au stade de la gastrulation. Par ailleurs, les Dnmt sont souvent surexprimées dans les tissus tumoraux et l’inhibition pharmacologique de Dnmt1 réduit la tumorigenèse dans un modèle murin de néoplasie intestinale [
3]. |
Comment les Dnmt répriment-elles la transcription ? Connexion étroite entre les Dnmt et la désacétylation des histones Les travaux initiaux de A. Bird [
4] et A. Wolffe [
5] ont révélé une relation mécanistique entre la méthylation de l’ADN et une modification post-traductionnelle de la chromatine bien connue, la désacétylation des histones (→) [
6]. Ces travaux ont montré que les répresseurs à domaine MBD ( methyl-CpG-binding domain), qui se fixent spécifiquement sur l’ADN méthylé, sont capables de recruter les histone-désacétylases (HDAC) au niveau des séquences génomiques méthylées, ce qui a pour effet de désacétyler les histones et de maintenir une structure condensée de la chromatine, réprimant ainsi la transcription [ 4, 5]. Récemment, une connexion bien plus étroite entre la méthylation des cytosines et la désacétylation des histones a été caractérisée. Trois équipes ont démontré, par des expériences de transfection transitoire, que les protéines Dnmt1, Dnmt3a et Dnmt3b, répriment activement la transcription lorsqu’elles sont fusionnées à un domaine de liaison à l’ADN [
7–
10]. De plus, ces enzymes chimériques interagissent avec les histone-désacétylases et leur effet répresseur est levé par un inhibiteur spécifique des HDAC, la trichostatine A [ 7– 10]. Ces travaux ont permis de révéler l’un des mécanismes par lesquels les Dnmt verrouillent l’expression génique. Étonnamment, l’effet répresseur des Dnmt associées aux HDAC peut s’exercer indépendamment de leur activité méthyltransférase. Ainsi, Dnmt3L, une Dnmt dépourvue de domaine catalytique, donc d’activité méthyltransférase, peut cependant recruter les enzymes HDAC et inhiber la transcription [
11]. Un nouveau concept émerge de ces observations, quant aux fonctions des Dnmt: ce sont des protéines multi-fonctionnelles qui jouent des rôles cellulaires complémentaires et/ou additionnels à leur capacité de méthyler l’ADN. Les Dnmt pourraient donc constituer des plates-formes permettant le recrutement, au sein du génome, des enzymes de modification de la chromatine. (→) m/s 1998, n° 4, p. 455
Quand la méthylation de l’ADN rencontre la méthylation des histones Des travaux très récents révèlent que l’inhibition de la transcription résulte à la fois de la méthylation de l’ADN et d’un type de modification de la chromatine nouvellement caractérisé, la méthylation des histones. Cette modification post-traductionnelle des histones, et en particulier de la lysine 9 de l’histone H3, s’avère fondamentale pour la répression de l’expression génique [
12]. La méthylation de la lysine 9 par les histone-méthyltransférases (HMT) crée un site de fixation pour le répresseur HP1 ( heterochromatin protein 1), une protéine associée à la formation de l’hétérochromatine. Les équipes de E. Selker et S. Jacobsen ont montré que des mutations du gène dim-5 chez le champignon Neurospora crassa et du gène kryptonite chez la plante Arabidopsis thaliana, ont pour conséquence une nette réduction de la méthylation de l’ADN chez ces organismes. Il s’est avéré que les gènes dim-5 et kryptonite codent pour des enzymes qui méthylent l’histone H3 sur la lysine 9. De plus, des mutations de ces gènes qui affectent l’activité de méthylation des histones induisent également des défauts de la méthylation de l’ADN [
13,
14]. Dans le contexte d’ A, thaliana, des données d’interaction in vitro suggèrent que LHP1, homologue du répresseur HP1 des mammifères, est associé à la méthyltransférase de l’ADN CMT3 [ 14]. Enfin, chez les mammifères, Dnmt1 et Dnmt3a interagissent avec la protéine HP1 et sont associées à une activité de méthylation de l’histone H3 sur la lysine 9. Ces études mettent pour la première fois en évidence une connexion entre la méthylation de l’ADN et la méthylation des histones. En outre, il découle de ces travaux que la méthylation de l’ADN est dépendante de la méthylation des histones. Notons que la relation inverse semble également vraie, c’est-à-dire que la méthylation de l’ADN pourrait faciliter la méthylation des histones. Des expériences d’immunoprécipitation de la chromatine (ChIP) réalisées sur des cellules inductibles pour le répresseur MeCP2, qui se fixe spécifiquement sur l’ADN méthylé, indiquent que la présence de MeCP2 est requise pour la méthylation de la lysine 9 de l’histone H3 dans le cas de l’ imprinting d’un gène [
15]. Bien que ces observations doivent encore être étayées par d’autres études, elles suggèrent d’ores et déjà l’existence d’une boucle de régulation entre la méthylation de l’ADN et la méthylation des histones, qui aurait pour conséquence de maintenir et de propager les états épigénétiques répressifs de la chromatine au cours des divisions cellulaires successives (→) (Figure 1), (→) m/s 2001, n° 10, p. 1056
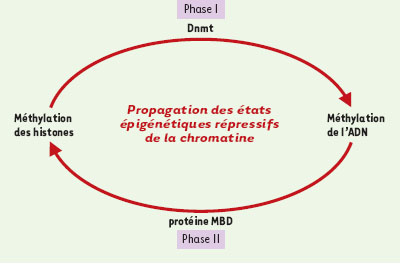
 | Figure 1.
Modèle de boucle de régulation pour la propagation des états épigénétiques de la chromatine. La méthylation des histones permet la fixation du répresseur HP1 sur la lysine 9 de l’histone H3 préalablement méthylée. La fixation de HP1 facilite le recrutement des méthyltransférases de l’ADN (Dnmt) (phase I du cycle) [ 13, 14]. Par ailleurs (phase II), la protéine à domaine MBD ( methyl-CpG-binding domain), MeCP2, se fixe aux CpG méthylés par les Dnmt et pourrait influencer la méthylation de la lysine 9 de l’histone H3 [ 15]. La méthylation de l’ADN et des histones pourrait donc constituer une boucle de régulation qui aurait pour conséquence de propager et de maintenir de façon stable les états épigénétiques répressifs de la chromatine. |
En résumé, les récents développements mentionnés cidessus suggèrent que le verrouillage de la transcription par méthylation de l’ADN est intimement connecté au remodelage de la chromatine par des enzymes de modification des histones (Figure 2). Ces données permettent l’ébauche d’un modèle de répression de la transcription (Figure 2). Dans un premier temps, les méthyltransférases de l’ADN coopèrent avec les histone-désacétylases (HDAC) et les histone-méthyltransférases (HMT) pour modifier la chromatine et la rendre moins permissive pour la transcription. Les données obtenues chez Neurospora et Arabidopsis démontrent que le système HMT/HP1 permet de recruter les Dnmt aux sites de méthylation. Dans un deuxième temps, les protéines MBD se fixent aux cytosines méthylées par les Dnmt, recrutant ainsi des activités HDAC additionnelles. En outre, des premiers travaux effectués dans le contexte de MeCP2 suggèrent que les MBD pourraient cibler le complexe HMT/HP1 aux résidus H3Lys9 à méthyler et ainsi contribuer à la répression de la transcription. Globalement, ces différentes étapes liant méthylation de l’ADN et modifications de la chromatine conduiraient à un cycle de répression qui permettrait de renforcer le verrouillage des locus concernés. L’établissement d’une boucle de répression de la chromatine pourrait s’avérer particulièrement adapté aux situations cellulaires qui nécessitent l’extinction totale et stable d’un gène ou d’une région génomique spécifique. C’est par exemple le cas de l’inactivation du chromosome X chez les mammifères femelles, dont l’ADN est méthylé et les lysines 9 de l’histone H3 désacétylées et méthylées [
16,
17].
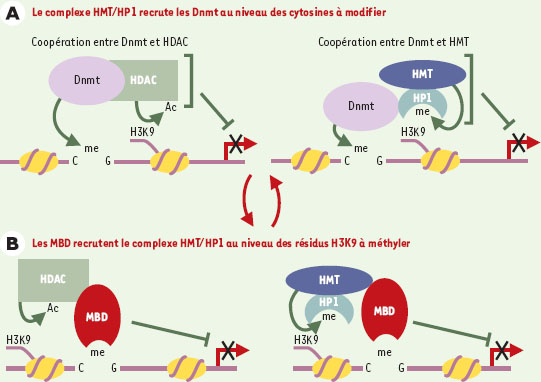
 | Figure 2.
Mécanismes moléculaires de verrouillage de la transcription par méthylation de l’ADN et modifications de la chromatine. Dans une première phase (A), l’association entre les méthyltransférases de l’ADN (Dnmt) et les histonedésacétylases (HDAC) conduit à la désacétylation des histones et, au moins dans certaines situations, à la méthylation des cytosines, provoquant une compaction de la chromatine et donc une répression de la transcription. Par ailleurs, les Dnmt sont recrutées par le répresseur HP1 (heterochromatin protein 1), lui-même fixé spécifiquement sur les histones H3 méthylées sur la lysine 9 (H3K9) par action de l’enzyme HMT (histone-méthyl-transférase). Dans une deuxième étape (B) , la méthylation des CpG permet la fixation de la protéine MBD (methyl-CpG-binding domain) qui recrute une activité HDAC. De plus, le facteur MBD recrute une activité HMT qui permet la liaison de HP1 et la répression, par un mécanisme encore inconnu, de la transcription. La méthylation de l’ADN pourrait ainsi en retour influencer la modification des histones, permettant ainsi le verrouillage total et stable de l’expression génique. Ac: groupe acétyl; me: groupe méthyl. |
|
Du verrouillage de la transcription par les Dnmt au cancer : perspectives Retrouvée dans près de 65 % des cancers, l’hyperméthylation de l’ADN joue sans doute un rôle clé dans la tumorigenèse [2]. La caractérisation d’une association physique entre les Dnmt et les histonedésacétylases constitue la première base moléculaire de ce phénomène. De nombreux promoteurs hyperméthylés dans les cancers sont effectivement associés à une hypoacétylation des histones. C’est le cas par exemple des promoteurs BRCA1, MTA1 ou encore p14ARF/p16 [
18]. De plus, le groupe de S. Baylin a récemment rapporté que les inhibiteurs des HDAC (trichostatine A) et des Dnmt (la 5-azacytidine) agissent de façon synergique dans la réactivation de gènes suppresseurs de tumeurs hyperméthylés dans des cellules cancéreuses du côlon [
19]. Ces observations pourraient ouvrir de nouvelles voies thérapeutiques par réactivation des gènes dont la méthylation est altérée dans les cancers. En effet, l’hyperméthylation des gènes étant un phénomène réversible, une thérapie épigénétique des cancers à base d’inhibiteurs des méthyltransférases de l’ADN et des histone-désacétylases est certainement envisageable. De toute évidence, ce traitement thérapeutique devra être très spécifique et sélectif quant aux gènes à réactiver. En vue d’atteindre cet objectif, il sera fondamental de cerner de façon précise les mécanismes par lesquels les Dnmt et le remodelage de la chromatine sont intimement connectés. Des avancées notables ont d’ores et déjà été réalisées dans ce sens. Les recherches futures viseront notamment à déterminer la séquence des événements qui vont de la méthylation de l’ADN à la modification des histones et qui conduisent à l’extinction de l’expression génique. Il sera également important d’examiner la relation entre les Dnmt et d’autres modifications post-traductionnelles des histones, telles que la phosphorylation ou la ribosylation. Étant donné l’engouement actuel pour l’étude de la méthylation de l’ADN et de la chromatine, gageons que le monde de l’épigénétique continuera, dans les années à venir, à révéler ses secrets à grands pas. |
Nous tenons à remercier Yvan de Launoit et Michel Goldman pour leur soutien et leur lecture critique de ce manuscrit. Les recherches effectuées dans notre laboratoire sont financées par l’Association Belge contre le Cancer et le FNRS ainsi que par l’Action de Recherche Concertée de la Communauté Française de Belgique. François Fuks est « Chercheur Qualifié » au Fonds National de la Recherche Scientifique (FNRS).
|
1. Jones PA, Takai D. The role of DNA methylation in mammalian epigenetics. Science 2001; 293: 1068–70. 2. Baylin SB, Herman JG. DNA hypermethylation in tumorigenesis: epigenetics joins genetics. Trends Genet 2000; 16: 168–74. 3. Jones PA. DNA methylation and cancer. Oncogene 2002; 21: 5358–60. 4. Nan X, Ng HH, Johnson CA, et al. Transcriptional repression by the methyl- CpG-binding protein MeCP2 involves a histone deacetylase complex. Nature 1998; 393: 386–9. 5. Jones PL, Veenstra GJ, Wade PA, et al, Methylated DNA and MeCP2 recruit histone deacetylase to repress transcription. Nat Genet 1998; 19: 187–91. 6. Taddei A, Almouzni G. Les acétyl-transférases et désacétylases des histones: des co-régulateurs de la transcription, Med Sci 1997; 13: 1205–9. 7. Fuks F, Burgers WA, Brehm A, Hughes-Davies L, Kouzarides T. DNA methyltransferase Dnmt1 associates with histone deacetylase activity. Nat Genet 2000; 24: 88–91. 8. Robertson KD, Ait-Si-Ali S, Yokochi T, Wade PA, Jones PL, Wolffe AP. DNMT1 forms a complex with Rb, E2F1 and HDAC1 and represses transcription from E2Fresponsive promoters. Nat Genet 2000; 25: 338–42. 9. Rountree MR, Bachman KE, Baylin SB. DNMT1 binds HDAC2 and a new corepressor, DMAP1, to form a complex at replication foci. Nat Genet 2000; 25: 269–77. 10. Fuks F, Burgers WA, Godin N, Kasai M, Kouzarides T. Dnmt3a binds deacetylases and is recruited by a sequence-specific repressor to silence transcription. EMBO J 2001; 20: 2536–44. 11. Deplus R, Brenner C, Burgers WA, et al, Dnmt3L is a transcriptional repressor that recruits histone deacetylase. Nucleic Acids Res 2002; 30: 3831–8. 12. Nicolas E, Vandel L, Trouche D. Quand la méthylation des histones entre en scène. Med Sci 2001; 17: 476–9. 13. Tamaru H, Selker EU. A histone H3 methyltransferase controls DNA methylation in Neurospora crassa, Nature 2001; 414: 277–83. 14. Jackson JP, Lindroth AM, Cao X, Jacobsen SE. Control of CpNpG DNA methylation by the KRYPTONITE histone H3 methyltransferase. Nature 2002; 416: 556–60. 15. Fuks F, Hurd PH, Wolf D, Nan X, Bird AP, Kouzarides T. The methyl-CpG-binding protein MeCP2 links DNA methylation to histone methylation. J Biol Chem 2003; 278: 4035–40. 16. Tazi J, Bird A. Alternative chromatin structure at CpG islands. Cell 1990; 60: 909–20. 17. Peters AH, Mermoud JE, O’Carroll D, et al. Histone H3 lysine 9 methylation is an epigenetic imprint of facultative heterochromatin. Nat Genet 2002; 30: 77–80. 18. Ballestar E, Esteller M. The impact of chromatin in human cancer: linking DNA methylation to gene silencing. Carcinogenesis 2002; 23: 1103–9. 19. Cameron EE, Bachman KE, Myohanen S, Herman JG, Baylin SB. Synergy of demethylation and histone deacetylase inhibition in the re- expression of genes silenced in cancer. Nat Genet 1999; 21: 103–7. |